Une équipe de scientifiques de l'Institut du génome de Singapour (GIS) et de l'Institut de bioinformatique (BII) d'A*STAR a développé un nouvel outil logiciel d'IA appelé « BANKSY » qui reconnaît automatiquement les types de cellules présents dans un tissu, tels que les cellules musculaires, les cellules nerveuses. , et les cellules graisseuses.
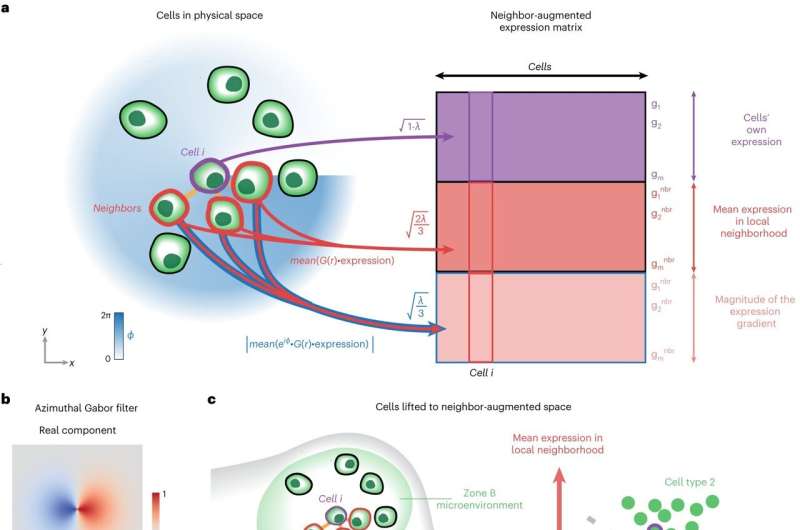
Allant au-delà des outils d'IA conventionnels, qui peuvent regrouper les cellules en clusters si elles contiennent des molécules similaires, BANKSY prend également en compte la similitude de l'environnement des cellules dans le tissu.
Avec BANKSY, les chercheurs pourraient améliorer leur compréhension des processus tissulaires dans diverses maladies plus rapidement et avec plus de précision, ce qui pourrait soutenir le développement de diagnostics et de traitements plus efficaces contre le cancer, les troubles neurologiques et d'autres maladies. Cette recherche a été publiée dans l'article "BANKSY unifie le typage cellulaire et la segmentation du domaine tissulaire pour une analyse de données omiques spatiales évolutives" dans Nature Genetics .
BANKSY sait identifier des groupes de cellules subtilement distincts dans des profils moléculaires spatiaux générés à partir d’échantillons de tissus. De plus, BANKSY aborde le problème distinct mais connexe de la démarcation de régions anatomiques fonctionnellement distinctes dans des coupes de tissus. Par exemple, il peut distinguer des structures en couches dans le cerveau antérieur humain.
Les technologies de profilage moléculaire spatial (Spatial Omics) sont des microscopes puissants qui permettent aux scientifiques d'étudier les tissus de manière très détaillée en révélant l'emplacement exact des molécules biologiques individuelles dans les cellules, ainsi que la disposition des cellules dans les tissus.
Cela les aide à comprendre comment les cellules se rassemblent dans les tissus pour remplir leurs fonctions physiologiques normales et également comment elles se comportent (ou se comportent mal) dans des maladies telles que le cancer, l'autisme ou des maladies infectieuses telles que le COVID-19. Cette compréhension est essentielle pour un diagnostic plus précis et un traitement adapté aux patients, ainsi que pour la découverte de nouveaux médicaments.
BANKSY peut aider les biologistes à interpréter et à extraire des informations des dernières technologies Spatial Omics apparues au cours des dernières années. Polyvalente, précise, rapide et évolutive, BANKSY se démarque des méthodes existantes pour analyser les données Spatial Omics basées sur l'ARN et les protéines.
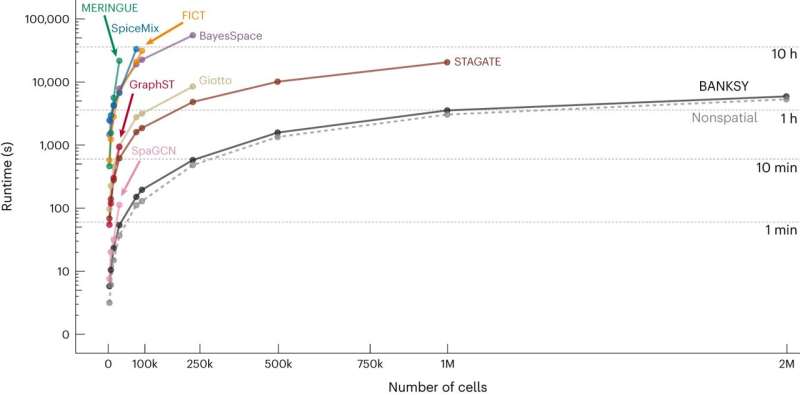
Capable de gérer de grands ensembles de données de plus de deux millions de cellules, BANKSY est 10 à 1 000 fois plus rapide que les méthodes concurrentes testées et 2 à 60 fois plus évolutive. Cela signifie que la méthode peut également être appliquée à d'autres étapes clés du traitement des données, telles que la détection et la suppression des zones de mauvaise qualité de l'échantillon et la fusion d'échantillons prélevés sur différents patients pour une analyse combinée.
BANKSY a été évalué de manière indépendante et s'est révélé être l'algorithme le plus performant pour les données omiques spatiales par deux études indépendantes, dont l'une a conclu que BANKSY pourrait être une solution puissante pour l'identification de domaines. L'autre étude a testé six algorithmes et a sélectionné BANKSY comme le plus précis pour son analyse de données.
Le Dr Shyam Prabhakar, chef de groupe principal au Laboratoire de biologie des systèmes et d'analyse des données et directeur associé des systèmes spatiaux et unicellulaires au SIG d'A*STAR, a déclaré :« Nous prévoyons que BANKSY sera un outil révolutionnaire qui aidera à débloquer le potentiel des technologies émergentes Spatial Omics."
"Nous espérons que cela améliorera notre compréhension des processus tissulaires dans diverses maladies, nous permettant ainsi de développer des traitements plus efficaces contre les cancers, les troubles neurologiques et de nombreuses autres pathologies."
Le professeur Liu Jian Jun, directeur exécutif par intérim du GIS d'A*STAR, a déclaré :« Les travaux sur BANKSY font progresser notre stratégie consistant à combiner des technologies à haut débit avec un logiciel d'IA évolutif et robuste pour résoudre les problèmes et identifier les indices de ce qui peut faire la différence. dans la vie des patients."
Le Dr Iain Tan, consultant principal à la division d'oncologie médicale du National Cancer Center de Singapour et clinicien-chercheur principal au laboratoire SIG de génomique appliquée du cancer d'A*STAR, a déclaré :« Nous utilisons BANKSY pour identifier les cellules qui aident les tumeurs à se développer et à se propager. d'autres parties du corps – des médicaments ciblant ces cellules pourraient constituer une voie prometteuse pour le traitement du cancer. "
Plus d'informations : Vipul Singhal et al, BANKSY unifie le typage cellulaire et la segmentation des domaines tissulaires pour une analyse de données omiques spatiales évolutive, Nature Genetics (2024). DOI :10.1038/s41588-024-01664-3
Informations sur le journal : Génétique naturelle
Fourni par l'Agence pour la science, la technologie et la recherche (A*STAR), Singapour