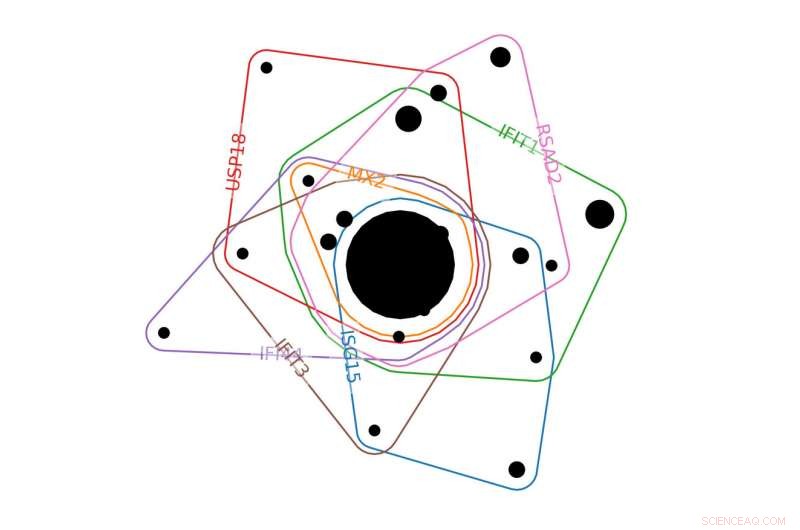
Dans cette partie d'un exemple d'hypergraphe d'un système biologique, les lignes colorées représentent les gènes et encerclent les conditions expérimentales (cercles noirs) où le gène était significatif. Plus le cercle noir est grand, plus les conditions expérimentales sont dans ce groupe. Crédit :Émilie Purvine | PNNL
Les scientifiques se précipitent pour suivre le rythme de COVID-19, créer de nouveaux outils pour comprendre comment fonctionne le nouveau coronavirus.
Pour les chercheurs du Pacific Northwest National Laboratory (PNNL), comprendre l'infection virale est une question de mathématiques plutôt qu'une analyse purement moléculaire. Ils utilisent un outil mathématique avancé appelé hypergraphes pour identifier comment les cellules humaines réagissent à une infection virale, y compris le nouveau coronavirus. Les protéines clés participant à cette réponse pourraient être des cibles pour le développement de médicaments pour traiter le COVID-19.
La mathématicienne du PNNL Emilie Purvine et le biologiste computationnel Jason McDermott ont récemment présenté leurs travaux virtuellement au SIGKDD (Special Interest Group on Knowledge Discovery and Data Mining) de l'Association for Computing Machinery, une conférence annuelle pour l'exploration de données, science des données, et analytique.
Hypergraphes pour infection virale
Dans une étape clé, l'équipe a testé la nouvelle approche avec les données d'un virus similaire, le coronavirus qui cause le syndrome respiratoire aigu sévère, ou SRAS. Ce virus a infecté plus de 8, 000 personnes lorsqu'il a balayé le monde en 2003.
L'équipe du PNNL a constaté que les résultats de la nouvelle méthode correspondaient aux données précédemment collectées sur ce virus. À l'aide d'hypergraphes, l'équipe a identifié et classé l'activité de plusieurs gènes maintenant connus pour être importants pour l'activité du virus qui a causé l'épidémie de SRAS-1.
"Notre travail a identifié de manière indépendante les mêmes gènes connus pour être importants avec l'activité du SRAS. C'était une étape importante à franchir avant d'appliquer nos travaux au virus qui cause COVID-19, " a déclaré McDermott.
Maintenant, l'équipe du PNNL applique la nouvelle technologie au virus actuel, en utilisant des hypergraphes pour trier et classer l'importance de plusieurs des centaines de gènes actifs dans COVID-19.
Purvine et McDermott ont utilisé des hypergraphes pour explorer comment les cellules humaines réagissent aux infections virales au cours des deux dernières années. Ils ont travaillé avec des données recueillies par la biologiste du PNNL Katrina Waters, qui a suivi l'expression des gènes, expression des protéines, et les changements moléculaires dans les cellules humaines infectées par des virus, y compris la grippe, Zika, Ebola, et les coronavirus pendant environ une décennie.
Pour appliquer des hypergraphes à ce grand ensemble de données, les chercheurs ont d'abord dû trouver comment identifier des groupes de protéines de manière à les préparer à construire un hypergraphe significatif. L'équipe s'est attaquée à ce défi plus tôt cette année, en même temps, la pandémie de coronavirus a frappé.
Des graphiques aux hypergraphes
La collaboration avec Purvine offre un nouvel outil à McDermott, qui a utilisé des techniques mathématiques basées sur des graphes pour analyser les connexions entre les gènes, protéines, et des molécules de signalisation dans les cellules pendant des années.
Lui et ses collègues identifient les relations entre deux molécules à la fois. Ensuite, ils catégorisent les connexions entre de nombreuses interactions distinctes. Ces connexions s'emmêlent rapidement dans des graphiques complexes représentant des réseaux moléculaires qui maintiennent le fonctionnement des cellules.
Les chercheurs analysent la structure et la forme de ces graphiques, à la recherche de modèles significatifs qui indiquent des composants moléculaires avec des rôles clés. Centralité, ou lorsqu'une molécule a de nombreuses connexions avec d'autres, est un type de modèle.
La structure entière d'un graphique est un autre modèle significatif. Certaines connexions centrales agissent comme des ponts pour maintenir la circulation des informations entre les différentes parties du réseau. Les gènes ou les protéines impliqués dans ces connexions « intermédiaires » permettent probablement à une cellule entière de fonctionner correctement.
Les hypergraphes représentent un bond en avant potentiel. Au lieu de représenter les connexions entre les composants individuels, les hypergraphes montrent des relations entre des groupes de choses. Étant donné que les réseaux biologiques fonctionnent à travers des groupes moléculaires, les scientifiques pensent que les hypergraphes pourraient représenter leur structure de manière plus réaliste que les graphiques standard.
Les scientifiques ont utilisé des hypergraphes pour représenter les groupes sociaux et l'infrastructure de réseau informatique, mais leur complexité de calcul en fait une technique rare pour étudier les réseaux biologiques à grande échelle qui découlent de données expérimentales.
Un outil logiciel d'hypergraphe open source appelé HyperNetX, développé au PNNL, rend cette analyse plus accessible aux chercheurs de diverses disciplines. Mais appliquer la technique à des données provenant de divers domaines nécessite encore un peu de bricolage.
"Comme il y a tellement de façons de construire des hypergraphes à partir de données biologiques, les biologistes ont probablement besoin d'impliquer un mathématicien informaticien pour ce faire, pour l'instant, " dit Purvine.