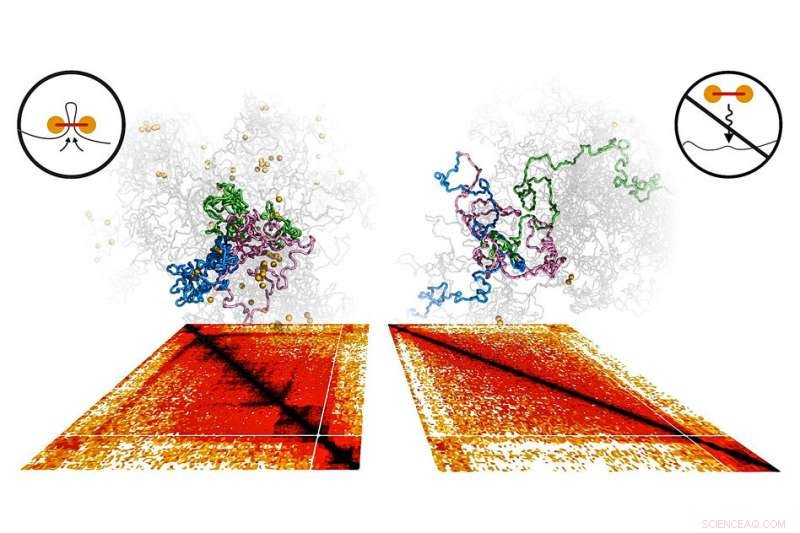
Les modèles informatiques des chromosomes et les cartes expérimentales Hi-C présentées ci-dessous révèlent le rôle central de la cohésine dans le repliement du génome en domaines distincts (surlignés en bleu, vert, et rose). Lorsque la cohésine est épuisée expérimentalement et dans les simulations, les domaines se déploient et se mélangent (à droite). Crédit :Massachusetts Institute of Technology
Un "moteur" moléculaire qui organise le génome en quartiers distincts en formant des boucles d'ADN a été caractérisé par des chercheurs du MIT et de l'Institut Pasteur en France.
Dans une étude publiée en 2016, une équipe dirigée par Leonid Mirny, professeur de physique à l'Institute for Medical Engineering and Sciences du MIT, ont proposé que les moteurs moléculaires transforment les chromosomes d'un état vaguement enchevêtré en une série dynamique de boucles en expansion.
Le processus, connu sous le nom d'extrusion de boucle, On pense qu'il rassemble des éléments régulateurs avec les gènes qu'ils contrôlent. L'équipe a également suggéré que l'ADN est décoré de barrières, semblables à des panneaux d'arrêt, qui limitent le processus d'extrusion.
De cette façon, l'extrusion de la boucle divise les chromosomes en quartiers régulateurs séparés, connus sous le nom de domaines d'association topologique (TAD).
Cependant, tandis que les chercheurs ont suggéré qu'un complexe protéique en forme d'anneau appelé cohésine était un candidat probable pour ces moteurs moléculaires, cela restait à prouver.
Maintenant, dans un article publié dans la revue La nature , une équipe dirigée par Mirny et François Spitz à l'Institut Pasteur, ont démontré que la cohésine joue bien le rôle de moteur dans le processus d'extrusion de boucle.
"Chacune de ces machines atterrit sur l'ADN et commence à extruder des boucles, mais il y a des limites sur l'ADN que ces moteurs ne peuvent pas traverser, " dit Mirny. " Donc, à la suite de cette activité motrice, le génome est organisé en de nombreuses boucles dynamiques qui ne franchissent pas les frontières, ainsi le génome se divise en une série de voisinages."
Les chercheurs ont également découvert qu'un mécanisme différent, qui n'utilise pas de cohésine, est à l'œuvre en organisant les régions actives et inactives de l'ADN en compartiments séparés dans le noyau de la cellule.
Pour déterminer le rôle de la cohésine dans la formation du génome, l'équipe a d'abord supprimé une molécule connue sous le nom de Nipbl, qui est responsable du chargement de la cohésine sur l'ADN.
Ils ont ensuite utilisé une technique expérimentale connue sous le nom de Hi-C, dans lequel des parties d'ADN proches les unes des autres dans l'espace 3-D sont capturées et séquencées, dans le but de mesurer la fréquence des interactions physiques entre différents points le long des chromosomes.
Cette technique, qui a été lancé par Job Dekker, professeur de biochimie et de pharmacologie moléculaire au centre médical de l'Université du Massachusetts à Worcester, a déjà été utilisé pour démontrer l'existence de TAD.
L'équipe a d'abord utilisé la technique Hi-C pour évaluer l'organisation des chromosomes avant de retirer la molécule Nipbl des souris. Ils ont ensuite retiré la molécule et effectué à nouveau la même mesure.
Ils ont constaté que les quartiers avaient pratiquement disparu.
Cependant, la compartimentation entre régions actives et inactives du génome s'était encore accentuée.
L'équipe pense que les moteurs de la cohésine permettent à chaque gène d'atteindre ses éléments régulateurs, qui contrôlent si les gènes doivent être activés ou désactivés.
Quoi de plus, il semble que les moteurs de la cohésine soient arrêtés par une autre protéine, CTCF, qui délimite les limites de chaque quartier. Dans une étude récente de la revue Cellule , le laboratoire Mirny, en collaboration avec des chercheurs de l'Université de Californie à San Francisco et de la faculté de médecine de l'Université du Massachusetts a démontré que si cette protéine de démarcation est supprimée, les frontières entre les quartiers disparaissent, permettre aux gènes d'un quartier de parler à des éléments régulateurs auxquels ils ne devraient pas parler dans un autre quartier, et conduisant à une mauvaise régulation des gènes dans la cellule.
"La cohésine est centrale pour la régulation des gènes, et nous soulignons qu'il s'agit d'une fonction motrice, ce n'est donc pas seulement qu'ils (les gènes et leurs éléments régulateurs) se retrouvent quelque part au hasard dans l'espace, mais ils étaient réunis par cette activité motrice, " dit Mirny.
Cet article fournit de nouvelles informations moléculaires importantes sur les mécanismes par lesquels les cellules replient leurs chromosomes, selon Dekker, qui n'a pas participé à la présente étude.
"Dans ce travail, les laboratoires Mirny et Spitz combinent des modèles de souris avec des approches génomiques pour étudier le repliement des chromosomes et révéler que la machine qui charge le complexe de cohésine est essentielle à la formation de TAD, " Dit Dekker. " D'après cette étude et une autre étude précédente, un mécanisme moléculaire apparaît où les TAD se forment par extrusion de boucle de chromatine dépendante de la cohésine et de Nipbl, qui est bloqué par les sites liés par le CTCF."
Les chercheurs tentent maintenant de caractériser comment l'absence du moteur moléculaire affecterait la régulation des gènes. Ils effectuent également des simulations informatiques dans le but de déterminer comment l'extrusion de la boucle à base de cohésine a lieu en même temps que le génome subit le processus indépendant de ségrégation en compartiments actifs et inactifs.
"C'est comme deux pianistes jouant sur le même piano, " dit Nezar Abdennur, un doctorant au laboratoire de Mirny, qui a participé à l'étude aux côtés de son collègue doctorant Anton Goloborodko. "Ils s'immiscent et s'imposent des contraintes, mais ensemble, ils peuvent produire un beau morceau de musique."
Cette histoire est republiée avec l'aimable autorisation de MIT News (web.mit.edu/newsoffice/), un site populaire qui couvre l'actualité de la recherche du MIT, innovation et enseignement.