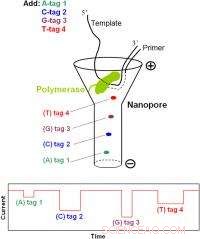
Schéma du séquençage de l'ADN d'une molécule unique par un nanopore avec des nucléotides marqués au phosphate. Chacun des quatre nucléotides portera une étiquette différente. Pendant le SBS, ces balises, attaché via le terminal-phosphate du nucléotide, seront libérés un par un dans le nanopore où ils produiront des signatures de blocage de courant uniques pour la détermination de la séquence. Un large éventail de ces nanopores conduira à un séquençage d'ADN à haut débit.
(Phys.org)—Le séquençage de l'ADN est la force motrice des principales découvertes en médecine et en biologie. Par exemple, la séquence complète du génome d'un individu fournit des marqueurs et des lignes directrices importants pour le diagnostic médical et les soins de santé. Jusqu'à maintenant, le principal obstacle a été le coût et la rapidité d'obtention de séquences d'ADN très précises. Bien que de nombreuses avancées aient été réalisées au cours des 10 dernières années, la plupart des instruments de séquençage à haut débit actuels reposent sur des techniques optiques pour la détection des quatre éléments constitutifs de l'ADN :A, C, G et T. Pour faire progresser davantage la capacité de mesure, le séquençage électronique de l'ADN d'un ensemble de matrices d'ADN a également été développé.
Récemment, il a été démontré que l'ADN peut être enfilé à travers des pores de protéines à l'échelle nanométrique sous un courant électrique appliqué pour produire des signaux électroniques au niveau d'une seule molécule. Cependant, parce que les quatre nucléotides sont très similaires dans leurs structures chimiques, ils ne peuvent pas être facilement distingués à l'aide de cette technique. Ainsi, la recherche et le développement d'une plate-forme de séquençage d'ADN électronique à molécule unique est le domaine d'investigation le plus actif et a le potentiel de produire un séquenceur d'ADN portable capable de déchiffrer le génome pour la médecine personnalisée et la recherche biomédicale fondamentale.
Une équipe de chercheurs de l'Université Columbia, dirigé par le Dr Jingyue Ju (le professeur d'ingénierie Samuel Ruben-Peter G. Viele, Professeur de génie chimique et de pharmacologie, directeur du Centre de technologie du génome et de génie biomoléculaire), avec des collègues du National Institute of Standards and Technology (NIST) dirigé par le Dr John Kasianowicz (Fellow of the American Physical Society), ont développé une nouvelle approche pour potentiellement séquencer l'ADN dans les nanopores électroniquement au niveau d'une seule molécule avec une résolution à base unique. Ce travail, intitulé « PEG-Labeled Nucleotides and Nanopore Detection for Single Molecule DNA Sequencing by Synthesis » est désormais disponible dans la revue en ligne en libre accès, Rapports scientifiques , du groupe Publication Nature.
La stratégie de séquençage par synthèse basée sur les nanopores (Nano-SBS) rapportée peut distinguer avec précision quatre bases d'ADN en détectant 4 balises de tailles différentes libérées à partir de nucléotides modifiés par 5'-phosphate au niveau de la molécule unique pour la détermination de la séquence. Le principe de base de la stratégie Nano-SBS est décrit comme suit. Comme chaque analogue de nucléotide est incorporé dans le brin d'ADN en croissance pendant la réaction de polymérase, son marqueur est libéré par la formation de liaisons phosphodiester. Les tags entreront dans un nanopore dans l'ordre de leur libération, produire des signatures de blocage de courant ionique uniques en raison de leurs structures chimiques distinctes, déterminant ainsi la séquence d'ADN électroniquement au niveau d'une seule molécule avec une résolution de base unique. Comme preuve de principe, l'équipe de recherche a attaché quatre étiquettes polymères de longueurs différentes au phosphate terminal du 2'-désoxyguanosine-5'-tétraphosphate (un élément constitutif de l'ADN modifié) et a démontré une incorporation efficace des analogues nucléotidiques au cours de la réaction de polymérase, ainsi qu'une discrimination meilleure que la ligne de base entre les quatre balises au niveau d'une seule molécule sur la base de leurs signatures de blocage de courant ionique nanopore. Cette approche couplée à la polymérase attachée aux nanopores dans un format de matrice devrait produire une plate-forme électronique Nano-SBS à molécule unique.
Dans des travaux antérieurs, le Center of Genome Technology &Biomolecular Engineering de l'Université de Columbia, dirigé par le professeur Ju et le Dr Nicholas J. Turro (William P. Schweitzer professeur de chimie), développé une plateforme de séquençage d'ADN par synthèse (SBS) à quatre couleurs utilisant des terminateurs réversibles de nucléotides fluorescents clivables (NRT), qui est licencié à Intelligent Bio-Systems, Inc., une société QIAGEN. Le SBS avec des NRT fluorescents clivables est l'approche dominante utilisée dans les systèmes de séquençage d'ADN de nouvelle génération. Le Dr Kasianowicz et son groupe du NIST ont été les pionniers de l'étude des nanopores pour l'analyse d'une seule molécule. Ils ont précédemment signalé que des polymères de longueurs différentes, les polyéthylène glycols (PEG), ont pu être distingués par leurs effets uniques sur les lectures actuelles dans les nanopores d'une protéine α-hémolysine au niveau d'une seule molécule et ont ensuite développé une théorie pour la méthode. Leurs résultats fournissent la preuve de concept pour la spectrométrie de masse à molécule unique. La combinaison du concept SBS avec les étiquettes électroniques distinctes détectables par les nanopores pour marquer les blocs de construction de l'ADN a conduit au développement de l'approche électronique à molécule unique Nano-SBS a décrit le courant Rapports scientifiques article.
Comme le souligne l'auteur principal, le Dr Shiv Kumar, "La nouveauté de notre approche réside dans la conception et l'utilisation de quatre nucléotides marqués différemment, qui lors de l'incorporation par l'ADN polymérase, libèrent quatre étiquettes de tailles différentes qui se distinguent les unes des autres au niveau de la molécule unique lorsqu'elles traversent le nanopore. Cette approche surmonte toutes les contraintes imposées par les petites différences entre les quatre nucléotides, un défi auquel la plupart des méthodes de séquençage des nanopores sont confrontées depuis des décennies. la technique est assez souple; avec des balises PEG comme prototypes, d'autres marqueurs chimiques peuvent être choisis pour fournir une séparation optimale dans différents systèmes de nanopores.
Avec le développement ultérieur de cette approche Nano-SBS, comme l'utilisation de larges réseaux de protéines ou de nanopores solides, ce système a le potentiel de séquencer avec précision un génome humain entier rapidement et à faible coût, lui permettant ainsi d'être utilisé dans les diagnostics médicaux de routine.