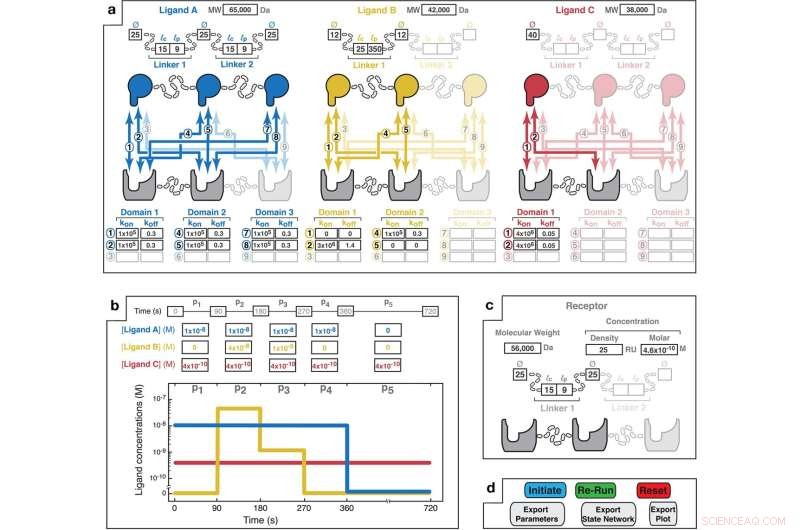
L'interface de conception d'entrée MVsim fournit une spécification interactive des paramètres pour les systèmes d'interaction multivalente et multimoléculaire. un Une interface pointer-cliquer permet à l'utilisateur de sélectionner le nombre de ligands (jusqu'à trois) et les valences du ou des ligands et du récepteur (jusqu'à trivalent) qui composent le système multivalent. Sur la base de la conception choisie, l'utilisateur spécifie la structure de chacun des ligands en entrant le poids moléculaire applicable (MW); les diamètres des domaines de liaison (Ø); les longueurs de contour (lc des lieurs (c. lieurs. De plus, les interactions combinatoires applicables (numérotées de 1 à 9) uniques à chaque appariement récepteur-ligand sont mises en évidence. Les champs de paramètres permettent la saisie de constantes de vitesse monovalentes pour chaque interaction par paires. Les interactions non contraignantes peuvent être indiquées avec k sur et k désactivé valeurs de zéro (par exemple, comme illustré avec le ligand B en jaune pour les interactions "1" et "5"). b Un champ de saisie permet à l'utilisateur de spécifier les schémas des concentrations totales de ligands en vrac. Une phase d'association se produit pendant les périodes de concentration de ligand en vrac non nulle (par exemple, 90 à 270 s pour le ligand B). Les phases de dissociation se produisent lorsque le ligand est retiré de la solution en vrac (par exemple, 360–720 s pour le ligand A). Ici, le ligand C est spécifié comme présent en continu dans la solution pendant les 720 s du cours temporel de l'interaction. L'affichage graphique permet de visualiser le modèle d'impulsion de concentration en vrac spécifié. c Paramètres d'entrée utilisateur pour le récepteur. La concentration du récepteur peut être spécifiée soit comme une densité de surface imitant la SPR (mesurée en RU ; où 1 RU équivaut à ~1 pg/mm 2 ) ou une concentration molaire. La topologie des récepteurs est spécifiée sous la même forme que celle décrite ci-dessus pour les ligands. j Le MVsim L'onglet contrôleur permet le lancement, l'itération et l'exportation des simulations de liaison. "Initier" exécute une simulation. "Re-run" exécute une simulation abrégée utilisée lorsqu'aucune modification n'a été apportée à la valence ou à la topologie du système. "Réinitialiser" relance l'application et efface les paramètres d'entrée de l'utilisateur de tous les champs. Crédit :Nature Communications (2022). DOI :10.1038/s41467-022-32496-6
Une équipe dirigée par des ingénieurs biomédicaux de l'Université du Minnesota Twin Cities a développé une application universellement accessible qui peut simuler des interactions moléculaires complexes, ce qui permettra aux chercheurs de concevoir de meilleurs traitements pour des maladies comme le cancer et le COVID-19.
L'article s'appuie sur une étude que les chercheurs ont publiée en 2019. Maintenant, ils ont élargi la technologie pour simuler des interactions moléculaires encore plus complexes, rendu l'application facile à utiliser pour les non-experts et appliqué leurs découvertes pour faire la lumière sur la façon dont le SRAS -Le virus CoV-2 infecte le corps.
L'étude est publiée dans Nature Communications , et l'application, appelée MVsim, est disponible gratuitement pour les autres chercheurs sur GitHub.
Le simulateur prédit la force, la vitesse et la sélectivité des interactions multivalentes, qui impliquent des molécules qui ont plusieurs sites de liaison et peuvent être utilisées pour développer des médicaments pour des maladies, en particulier le cancer et le COVID-19.
"Les interactions multivalentes sont vraiment importantes dans les systèmes biologiques naturels, et elles commencent maintenant à être exploitées de manière créative pour créer de nouveaux médicaments thérapeutiques qui tirent parti de leurs propriétés de liaison uniques", a déclaré Casim Sarkar, auteur principal de l'article et professeur à l'Université du Minnesota. Département de génie biomédical.
"Avec les médicaments multivalents, vous pouvez, en principe, cibler les cellules très spécifiquement d'une manière qui n'est pas possible avec les médicaments monovalents standard, mais il y a de nombreuses variables à prendre en compte dans leur conception et une grande partie du travail dans le domaine à ce jour a été fait. par essais et erreurs expérimentaux », a ajouté Sarkar. "Maintenant, grâce à MVsim, nous sommes en mesure de faire de bonnes prédictions qui peuvent être utilisées pour concevoir de manière plus rationnelle de telles thérapies."
De nombreux médicaments anticancéreux se lient non seulement aux cellules tumorales, mais également aux cellules qu'ils ne sont pas censés cibler, ce qui crée souvent des effets secondaires indésirables pour le patient. En optimisant la spécificité des interactions multivalentes à l'aide de MVsim, les chercheurs peuvent concevoir des médicaments qui ciblent plus spécifiquement les cellules d'une tumeur tout en minimisant la liaison à d'autres cellules du corps.
Un autre exemple est le virus SARS-CoV-2. Les scientifiques savent que le virus évolue pour mieux infecter nos cellules et échapper à notre système immunitaire, mais les mécanismes moléculaires derrière la façon dont le virus le fait sont relativement inconnus. Grâce à leur technologie MVsim, les chercheurs de l'Université du Minnesota ont pu explorer ce processus plus en profondeur, en découvrant les taux auxquels les domaines de liaison individuels au sein de la protéine de pointe multivalente du virus passent d'un état d'infection cellulaire à un état d'évasion immunitaire.
"We essentially have a computational microscope that allows us to look under the hood and see what multivalent proteins such as the SARS-CoV-2 spike protein are doing at a molecular level," Sarkar explained. "This level of molecular detail is hard to capture with a physical experiment. One of the real powers of MVsim is that we can not only learn more about how these systems work but we can also use this tool to design new multivalent interactions for diseases like cancer and COVID-19."
The researchers have already identified potential ways to limit the infectivity of current and future SARS-CoV-2 variants, which they plan to test soon. Engineered multivalent self-assembled binder protein against SARS-CoV-2 RBD