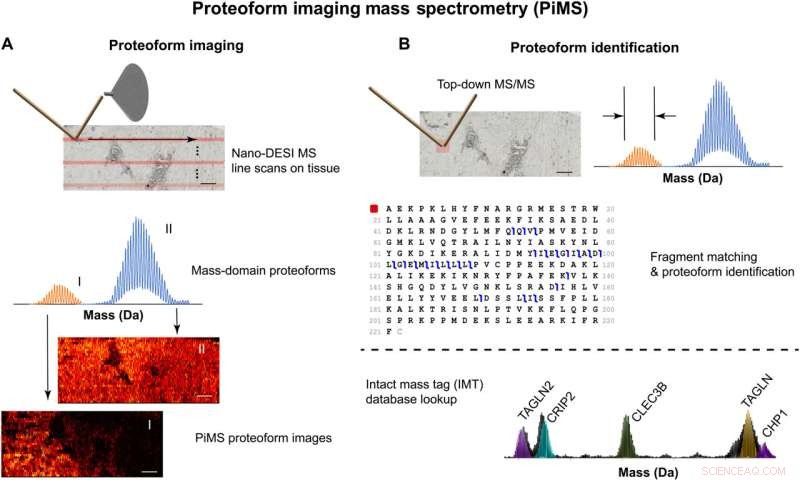
Illustration du flux de travail PiMS pour l'imagerie et l'identification des protéoformes. (A) Approche de numérisation (en haut), détection de protéoformes dans le domaine de masse (au milieu) et reconstruction d'image (en bas). (B) Deux approches pour identifier les protéoformes en utilisant soit la fragmentation directe des ions protéoformes et la lecture spectrale par ion individuel MS/MS (en haut) ou la recherche dans la base de données des valeurs de masse précises (IMT, en bas). Barres d'échelle, 1 mm. Crédit :Progrès scientifiques (2022). DOI :10.1126/sciadv.abp9929
Les enquêteurs dirigés par Neil Kelleher, Ph.D., professeur de médecine à la Division d'hématologie et d'oncologie et de biochimie et de génétique moléculaire, ont mis au point une nouvelle technique d'imagerie qui quadruple la détection des protéoformes intactes par rapport aux méthodes d'imagerie actuelles des protéines .
La technique d'imagerie, détaillée dans un article récent publié dans Science Advances , fournit une imagerie haute résolution et à haut débit des protéoformes ou de toutes les versions modifiées des protéines. Surtout, la technique est "sans étiquette", ne nécessite pas d'anticorps et peut identifier des protéoformes entières directement à partir de n'importe quel tissu non fixé. La technique peut actuellement détecter environ 1 000 protéoformes et localiser les protéoformes avec une résolution spatiale de 40 à 70 microns.
Plusieurs techniques sont couramment utilisées pour imager les protéines dans les tissus humains, mais très peu sont capables d'imager les protéoformes. Ceux qui peuvent imager des protéoformes entières le font en séparant la protéoforme du tissu et en les ionisant pour la spectrométrie de masse. Cependant, ces techniques offrent une faible spécificité moléculaire.
Pour résoudre ce problème, l'équipe de Kelleher a développé la spectrométrie de masse par imagerie protéoforme (PiMS). La technique fonctionne en extrayant les protéoformes du tissu avec des nanogouttelettes, en "pesant" les protéoformes extraites pour les identifier, puis en utilisant ces données pour construire des images protéoformes du tissu scanné.
« La véritable innovation du PiMS est qu'il associe une technique existante robuste d'extraction et d'ionisation des protéoformes, nanoDESI, à une technologie révolutionnaire de spectrométrie de masse ionique individuelle co-inventée par Thermo Fisher Scientific et Northwestern Proteomics. Par rapport aux techniques de détection habituelles, La spectrométrie de masse ionique individuelle offre jusqu'à 500 fois plus de sensibilité et 20 fois plus de pouvoir de résolution. Cela augmente considérablement la puissance de la technique, et le PiMS détecte des protéoformes plus grandes et plus rares et étend considérablement les limites de la couverture du protéome », a déclaré Kelleher, qui est également directeur du Northwestern's Proteomics Center of Excellence, du Robert H. Lurie Comprehensive Cancer Center's Proteomics Core Facility et du Northwestern's Chemistry of Life Processes Institute.
Pour démontrer les capacités de PiMS, l'équipe de Kelleher a utilisé la technique pour imager des protéoformes à partir d'unités fonctionnelles du rein humain. Ces images ont révélé des localisations spatiales distinctes de protéoformes de différentes régions anatomiques et unités tissulaires fonctionnelles comme le cortex rénal par rapport à la moelle.
Selon Kelleher, la couverture protéomique accrue du PiMS ouvre également la porte à des applications plus larges dans la cartographie moléculaire des tissus, l'identification de nouveaux biomarqueurs et l'amélioration du diagnostic des maladies.
"Récemment, il y a eu une grande poussée dans la génomique et la protéomique pour la biologie unicellulaire :pour mieux capturer l'hétérogénéité des maladies en utilisant des approches spatiales ou unicellulaires qui préservent les nombreux signaux divers au lieu d'approches en vrac qui mélangent tous les types de cellules et L'approche spatiale en particulier ajoute une bien plus grande précision pour l'imagerie des protéines et nous la poussons actuellement à identifier des milliers de protéoformes avec une résolution unicellulaire », a déclaré Kelleher. Projet de protéoforme humaine pour cartographier les protéines du corps humain