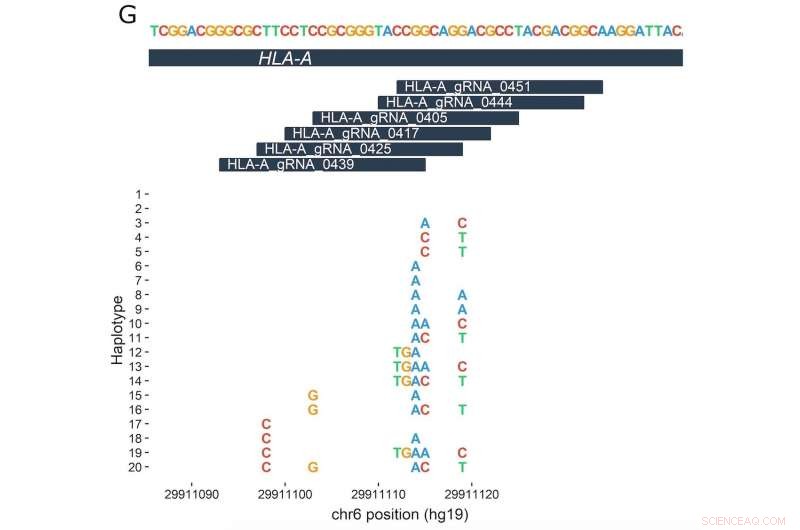
Le haut de ce diagramme montre une région de 30 paires de bases d'un seul locus génétique (HLA-A) qui est impliquée dans la réponse immunitaire et pourrait potentiellement être ciblée par l'édition de gènes. Les six barres plus petites en dessous représentent différents ARN guides conçus pour se lier à différentes parties de ce locus HLA-A. Sur l'axe Y (vertical) se trouvent des haplotypes avec des variations d'ADN mises en évidence identifiées à partir de différents individus. Ils sont alignés selon leurs positions dans la séquence génétique (représentées horizontalement sur l'axe X). Crédit :Lessard S; et al. PNAS Première édition , semaine du 11 décembre 2017.
L'édition de gènes a commencé à être testée dans des essais cliniques, en utilisant CRISPR-Cas9 et d'autres technologies pour éditer directement l'ADN à l'intérieur des cellules des personnes, et plusieurs essais sont en cours de recrutement ou en cours de planification. Une nouvelle étude menée par le Boston Children's Hospital et l'Université de Montréal met en garde, constater que les différences génétiques d'une personne à l'autre peuvent nuire à l'efficacité du processus d'édition des gènes ou, dans des cas plus rares, provoquer un effet "hors cible" potentiellement dangereux.
L'étude ajoute à la preuve que l'édition de gènes peut devoir être adaptée au génome de chaque patient, pour s'assurer qu'il n'y a pas de variantes dans la séquence d'ADN dans ou à proximité du gène ciblé qui perturberaient la technologie. Les résultats paraissent cette semaine dans le Actes de l'Académie nationale des sciences (11-15 décembre).
"Les humains varient dans leurs séquences d'ADN, et ce qui est considéré comme la séquence d'ADN "normale" de référence ne peut pas expliquer toutes ces différences, " dit Stuart Orkin, MARYLAND, du Dana-Farber Boston Children's Cancer and Blood Disorders Center et co-auteur de l'étude avec Matthew Canver, un étudiant MD-PhD à la Harvard Medical School. "Nous recommandons que la variation commune soit prise en compte dans la conception des systèmes de ciblage pour l'édition thérapeutique, pour maximiser l'efficacité et minimiser les problèmes de sécurité potentiels."
L'étude a analysé 7, 444 séquences du génome entier précédemment publiées. Sur la base d'une liste d'environ 30 cibles d'ADN liées à des maladies que les chercheurs souhaitent modifier par le biais de l'édition de gènes, les chercheurs ont dressé une deuxième liste de près de 3, 000 ARN guides (ARNg). Ce sont des morceaux de code génétique qui ont été développés pour diriger les enzymes CRISPR-Cas9 vers le bon emplacement d'édition sur ou à côté de la cible, comme l'adresse sur une enveloppe.
L'équipe, dirigé par Orkin, Canver et Samuel Lessard de l'Université de Montréal, puis regardé pour voir si l'un des 7, 444 individus portaient des variantes de séquences d'ADN (« changements de lettres » ou insertions/suppressions) dans les zones recherchées par les ARNg.
« S'il existe des différences génétiques sur le site que les réactifs CRISPR ciblent pour la thérapie, vous présentez un risque de diminution de l'efficacité ou d'échec du traitement, " explique Canver, qui a conçu et dirigé l'étude dans le laboratoire du Boston Children's Hospital d'Orkin. "Une différence dans une seule paire de bases peut entraîner une diminution de l'efficacité de liaison en raison d'un décalage avec l'ARN guide. Dans l'ensemble, cela peut entraîner une réduction de l'efficacité du traitement."
L'équipe a découvert que de telles occurrences dans le génome ne sont pas rares; environ 50 pour cent des ARNg analysés avaient le potentiel d'être affectés par des variantes sur leurs sites cibles. Dans quelques cas, l'équipe a découvert des variantes génétiques qui font que les séquences d'ADN dans le génome correspondent plus étroitement à un ARNg qui pourrait potentiellement l'attirer au mauvais endroit, entraînant une modification d'un gène ou d'une autre région d'ADN qui n'est pas censée être ciblée.
"Dans des cas rares, il y avait le potentiel de créer des sites "hors cible" très puissants - où les réactifs CRISPR pourraient se lier et couper là où ils ne sont pas destinés, " dit Canver. " Si un effet hors cible se produit, dire, un gène suppresseur de tumeur, ce serait une grande préoccupation."
Bien que l'étude ait porté sur l'édition du gène CRISPR-Cas9, les chercheurs pensent que leurs découvertes s'étendent à d'autres outils d'édition de gènes tels que les nucléases à doigts de zinc (ZFN) et les nucléases effectrices TAL.
"Le thème unificateur est que toutes ces technologies reposent sur l'identification d'étendues de bases d'ADN très spécifiquement, " dit Canver. " Alors, une variante qui affecte la séquence cible pourrait réduire la liaison à l'ARN guide. Des variantes peuvent également conduire à une liaison sur de nouveaux sites qui pourraient potentiellement causer des dommages. Alors que ces thérapies d'édition de gènes continuent de se développer et commencent à s'approcher de la clinique, il est important de s'assurer que chaque thérapie sera adaptée au patient qui va être traité."