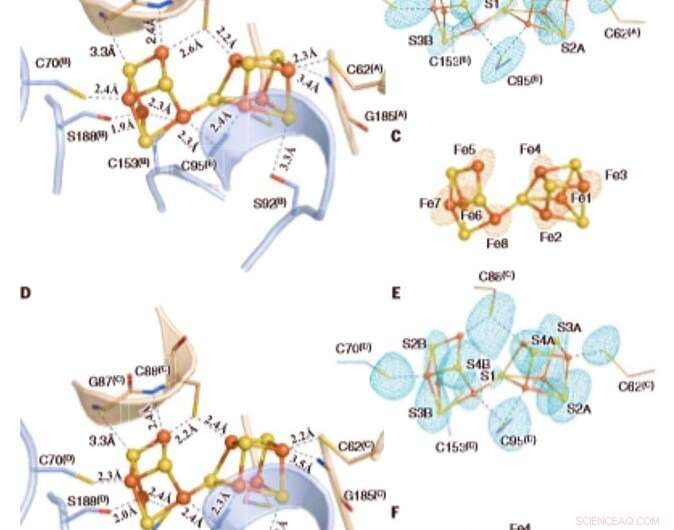
Les clusters P oxydés dans Av1*. Structures des P-clusters à (A à C) l'interface des chaînes A et B [P-cluster(A/B)] et (D à F) l'interface des chaînes C et D [P-cluster(C/D) ] de Av1*. Les chaînes A et C sont les sous-unités a, et les chaînes B et D sont les sous-unités b des deux dimères ab de Av1*. [(A) et (D)] Les clusters P sont présentés en forme de boule et de bâton, et les résidus clés interagissant avec les clusters P sont indiqués sous forme de bâtonnets. Les chaînes A et C sont représentées comme des rubans de blé, et les chaînes B et D sont représentées par des rubans bleu clair. [(B) et (C)] P-cluster(A/B) et [(E) et (F)] P-cluster(C/D) superposés avec [(B) et (E)] les cartes de densité anormale calculé à 7100 eV à une résolution de 2,18 Å et contourné à 4,0 s, montrant la position des atomes de soufre (maille menthe-bleu); et avec [(C) et (F)] les cartes de densité anormale calculées à 7141 eV à une résolution de 2,1 Å et contournées à 15,0s, montrant la position des atomes de fer (maille rouge). Les atomes sont colorés comme suit :Fe, Orange; S, jaune; , rouge; N, bleu. Les abréviations à une lettre pour les résidus d'acides aminés sont les suivantes :C, Cys; G, Gly; H, Le sien; R, Arg ; S, Sér. Crédit :Sciences, doi:10.1126/science.aaz6748
L'enzyme nitrogénase est un catalyseur biologique qui peut réduire le diazote (N2) en ammoniac en présence d'une suite de métallocofacteurs complexes. Cependant, les détails mécanistiques de la réaction restent rares. Dans un nouveau rapport sur Science , Wonchull Kang et une équipe de recherche en chimie, biologie moléculaire et biochimie à l'Université de Californie-Irvine, NOUS., ont rapporté une structure cristalline de 1,83 angström pour la protéine nitrogénase molybdène-fer (MoFe), qu'ils ont capturés dans des conditions physiologiques de renouvellement du diazote. Les résultats de l'étude peuvent évaluer les mécanismes possibles de N
La nitrogénase est le catalyseur d'une étape critique du cycle mondial de l'azote, lors de la réduction ambiante du diazote atmosphérique (N
Comprendre le mécanisme d'action de l'enzyme nitrogénase
De nombreux efforts ont été déployés pour comprendre les mécanismes de la nitrogénase depuis sa découverte, où certains s'étaient concentrés sur les interactions substrats et inhibiteurs de l'enzyme. De ces efforts, Kang et al. a déterminé une stratégie digne de considération en limitant les réserves d'électrons excédentaires qui ont conduit par inadvertance le N
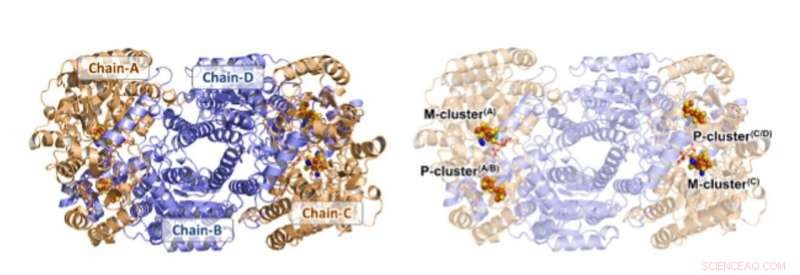
Structure d'Av1*. Présentations en ruban solide (gauche) et transparent (droite) de l'hétérotétramère d'Av1*, avec les sous-unités a (Chaîne-A et Chaîne-C) et les sous-unités b (Chaîne-B et Chaîne-D) colorées en blé et en bleu, respectivement. Les clusters M et P sont illustrés comme des modèles remplissant l'espace. Code couleur des atomes :Fe, Orange; S, jaune; , rouge; N, bleu; Mo, cyan; C, gris. Les M-clusters dans Chain-A et Chain-C sont désignés M-cluster(A) et M-cluster(C), respectivement. Les P-clusters aux interfaces Chain-A/Chain-B et Chain-C/Chain-D sont désignés P-cluster(A/B) et P-cluster(C/D), respectivement. PYMOL a été utilisé pour préparer cette figure. Crédit :Sciences, doi:10.1126/science.aaz6748
Comme preuve de concept, Kang et al. ont préparé l'extrait brut d'une souche bactérienne anaérobie Azotobacter vinelandii avec ou sans addition de dithionite après rupture cellulaire. La souche A. vinelandii a exprimé activement une Mo-nitrogénase contenant une protéine MoFe marquée à l'histidine dans les deux cas. Lorsqu'ils ont analysé l'activité de ces échantillons, les échantillons d'extraits bruts sans dithionite étaient presque inactifs pendant la réduction du substrat, en raison de l'épuisement des électrons dans les extraits bruts lors de la rupture des cellules. Kang et al. pourrait donc restaurer complètement l'activité des échantillons en ajoutant du dithionite et de l'ATP (c'est-à-dire en fournissant des électrons).
Amas de nitrogénase — deux métalloclusters uniques :le P-cluster et le M-cluster.
Sur la base des conditions décrites, lorsqu'une culture exprimant la nitrogénase qui effectue activement N
Un mécanisme plausible de réduction de N2 par la Mo-nitrogénase. Crédit :Markus Ribbe et Yilin Hu, Science , doi:10.1126/science.aaz6748
Un mécanisme d'action plausible en accord avec les observations expérimentales comprenait la réduction progressive du diazote (N
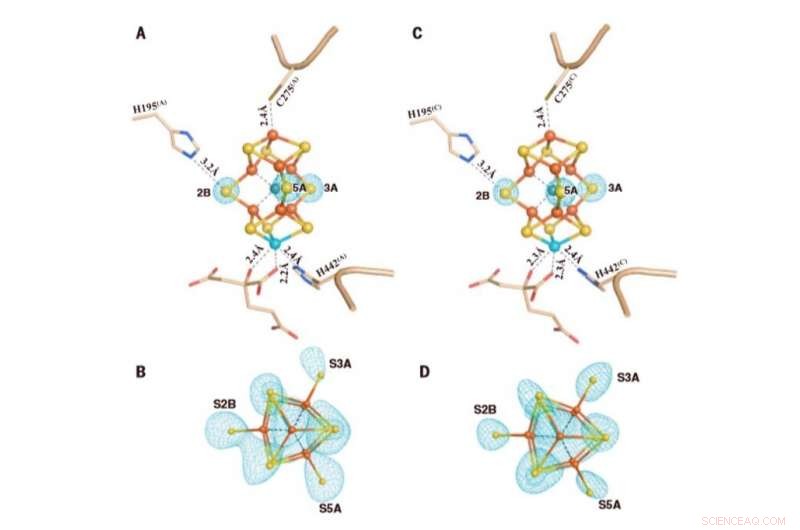
Les clusters M dans Av1*(TOD). Structures des M-clusters dans (A et B) chaîne A [M-cluster(A)] et (C et D) chaîne C [M-cluster(C)] affinées à une résolution de 1,73 Å. Vue latérale de (A) M-cluster (A) et (C) M-cluster (C) avec des résidus clés interagissant avec les clusters indiqués comme des bâtons. M-cluster(A) et M-cluster(C) sont superposés avec les cartes d'omission Fo-Fc des soufres de la ceinture contournées à 13s (maille bleu menthe). View along the Fe1-C-Mo direction of (B) M-cluster(A) and (D) M-cluster(C) superimposed with the anomalous density maps calculated at 7100 eV at a resolution of 2.17 Å and contoured at 4.0s, showing the presence of the anomalous sulfur density (mint-blue mesh) at all belt sulfur positions (S2B, S3A, and S5A) in (B) M-cluster(A) and (D) M-cluster(C). Credit:Science, doi:10.1126/science.aaz6748
To understand the sulfur-displaced conformation of AV1* under limited electron flux, the team formed AV1* turnover with dithionite (designated as AV1*TOD), to yield brown crystals that diffracted to a resolution of 1.73 Å. The observations were consistent with the mechanism of bound dinitrogen species on the compound and illustrated the physiological relevance of the conformation during catalysis. The capacity to displace three different sites by a dinitrogen species was consistent with previous investigations on catalysis-dependent selenium. Kang et al. proposed many mechanisms to explain the observations, however they seek further experimental support to verify them. The team highlighted the possibility for all belt-sulfur sites to be involved in the process of catalysis due to the presence of asymmetric belt-sulfur displacements in the compound. The results aim to provoke a paradigm shift in the mechanistic thinking of nitrogenase activity, ultimately to understand the intricate mechanism of the enzyme.
© 2020 Réseau Science X