Une équipe de recherche a réalisé un assemblage de haute qualité à l’échelle chromosomique du génome du carthame Chuanhonghua 1. Ce travail met en lumière les fondements génétiques de caractères cruciaux tels que la production d’acide linoléique (LA) et d’hydroxysafflor jaune A (HSYA). Il crée un nouveau précédent pour l'amélioration des cultures et les études de génomique fonctionnelle.
Le carthame (Carthamus tinctorius L.), une plante connue pour ses fleurs vibrantes et ses graines riches en nutriments, est cultivée depuis des milliers d'années dans la région fertile du croissant. Elle est très appréciée pour son huile, riche en LA; et ses fleurs, qui contiennent du HSYA, des substances aux multiples applications industrielles et médicinales. Malgré sa valeur, la recherche génétique sur le carthame a été limitée par la compréhension limitée de son génome.
Une étude parue dans Horticulture Research élucide les fondements génétiques du carthame, ouvrant ainsi la voie à des stratégies de sélection ciblées.
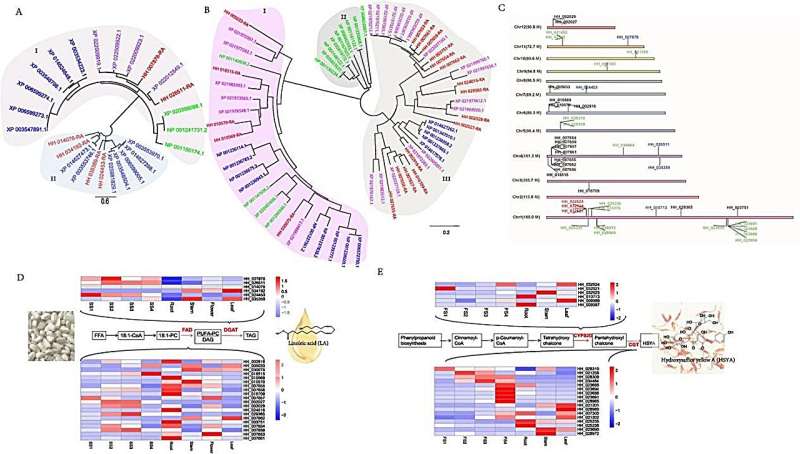
L'équipe de recherche a utilisé une stratégie de séquençage intégrée combinant les technologies Illumina, Oxford Nanopore et Hi-C pour obtenir un assemblage génomique de haute qualité de 1,17 Gb avec des contigs attribués à 12 chromosomes, reflétant une couverture d'environ 100 fois par rapport à la taille estimée du génome. Cette assemblée a facilité l'identification d'un événement récent de duplication du génome entier, étayant les vastes réarrangements génomiques et l'histoire évolutive du carthame, corroborés par une analyse phylogénétique utilisant 274 gènes à copie unique répartis dans 12 espèces, ainsi qu'un examen détaillé de l'expansion et de la contraction des familles de gènes.
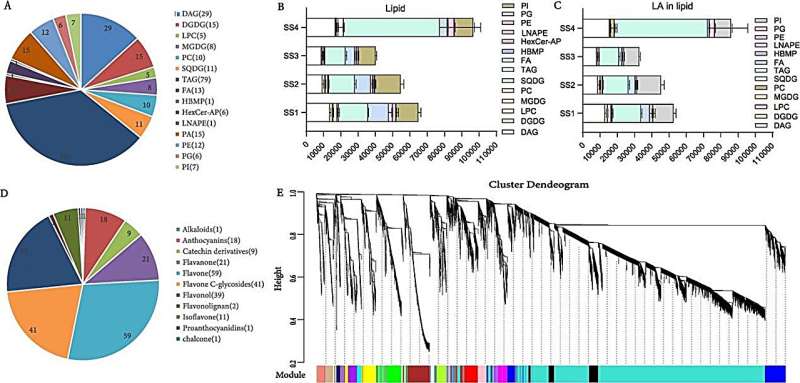
Le profilage métabolomique et transcriptomique à différents stades de développement des graines et des fleurs a révélé un lipidome complet avec une présence prédominante de triacylglycérols (TAG) et un spectre diversifié de métabolites flavonoïdes, mettant en évidence les voies de biosynthèse de composés clés tels que LA et HSYA. Grâce à l'analyse génomique, les chercheurs ont prédit 39 809 gènes codant pour des protéines, avec des annotations substantielles dans des bases de données publiques, et ont identifié des familles de gènes spécifiques :diacylglycérol acyltransférase (DGAT) et désaturases d'acides gras (FAD) pour la biosynthèse de LA, et CYP et CGT pour la biosynthèse de HSYA. indiquant leurs rôles centraux dans ces voies métaboliques.
Notamment, le re-séquençage de 220 lignées de carthame a donné 7 402 693 SNP de haute qualité, facilitant ainsi une étude d’association pangénomique (GWAS) qui a identifié des SNP significatifs associés à des caractères agronomiques, en particulier la teneur en huile et la couleur des fleurs. Cette analyse GWAS, ainsi que la vérification fonctionnelle du gène candidat HH_034464 (CtCGT1), impliqué dans la biosynthèse de HSYA, ont souligné le potentiel des marqueurs moléculaires dans l'amélioration des programmes de sélection pour les caractères souhaités. Les tests fonctionnels ont confirmé le rôle de CtCGT1 dans la glycosylation des flavonoïdes, cruciale pour la production de HSYA, mettant ainsi en lumière les fondements génétiques des traits métaboliques clés du carthame.
Le Dr John Smith, expert en génomique végétale, a salué cette réussite en déclarant :« Cet assemblage complet du génome constitue une étape importante dans la recherche sur le carthame. Il améliore non seulement notre compréhension de l'évolution des plantes, mais fournit également une riche ressource pour identifier les gènes responsables des gènes clés. traits agricoles et médicinaux."
En fournissant une base pour la sélection moléculaire du carthame, cette recherche offre le potentiel d’améliorer la résilience des cultures, leur contenu nutritionnel et leur efficacité thérapeutique. Pour l’avenir, le défi consiste à exploiter cette vaste ressource génomique pour développer des variétés de carthame supérieures qui répondent aux exigences d’un climat mondial changeant et d’une population croissante.
L'assemblage du génome à l'échelle des chromosomes du carthame Chuanhonghua 1 représente une étape cruciale dans l'exploration génétique de cette culture précieuse. Cette recherche ouvre non seulement de nouvelles voies pour l'extraction et la sélection des gènes fonctionnels du carthame, mais a également des implications plus larges pour les industries agricole et pharmaceutique.
Plus d'informations : Jiang Chen et al, Des études d'association sur le génome entier et à l'échelle du génome améliorent les caractéristiques agricoles clés du carthame à des fins industrielles et médicinales, Recherche horticole (2023). DOI : 10.1093/hr/uhad197
Informations sur le journal : Recherche horticole
Fourni par l'Université agricole de NanJing