La technologie de séquençage du génome fournit chaque année des milliers de nouveaux génomes végétaux. En agriculture, les chercheurs fusionnent ces informations génomiques avec des données d'observation (mesurant divers traits des plantes) pour identifier les corrélations entre les variantes génétiques et les traits des cultures comme le nombre de graines, la résistance aux infections fongiques, la couleur ou la saveur des fruits.
Cependant, la compréhension de la façon dont la variation génétique influence l’activité des gènes au niveau moléculaire est assez limitée. Ce manque de connaissances entrave la sélection de « cultures intelligentes » avec une qualité améliorée et un impact environnemental négatif réduit obtenu grâce à la combinaison de variantes génétiques spécifiques de fonction connue.
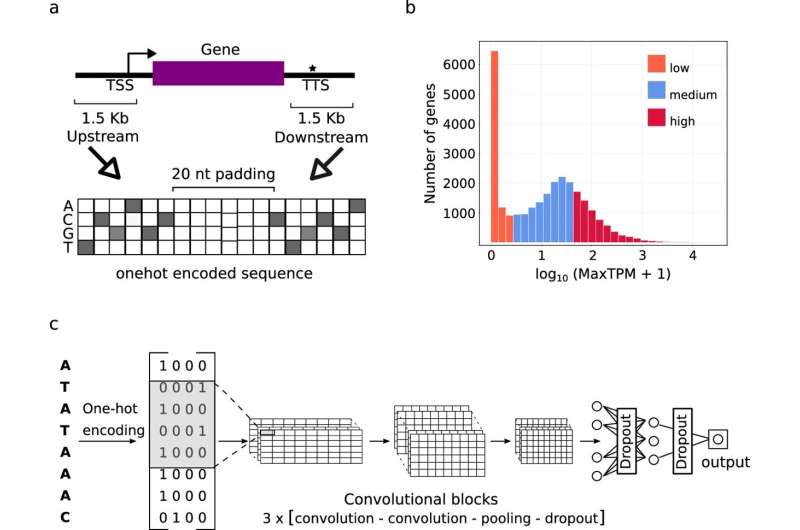
Des chercheurs de l'Institut IPK Leibniz et du Forschungszentrum Jülich (FZ) ont réalisé une avancée majeure pour relever ce défi. Dirigée par le Dr Jedrzej Jakub Szymanski, l'équipe de recherche internationale a formé des modèles d'apprentissage profond interprétables, un sous-ensemble d'algorithmes d'IA, sur un vaste ensemble de données d'informations génomiques provenant de diverses espèces végétales.
"Ces modèles ont non seulement pu prédire avec précision l'activité des gènes à partir de séquences, mais également identifier les parties de séquence qui contribuent à ces prédictions", explique le chef du groupe de recherche "Analyse et modélisation de réseau" de l'IPK. La technologie d'IA appliquée par les chercheurs s'apparente à celle utilisée en vision par ordinateur, qui consiste à reconnaître les traits du visage dans les images et à en déduire des émotions.
Contrairement aux approches précédentes basées sur l'enrichissement statistique, les chercheurs ont combiné ici l'identification des caractéristiques de la séquence avec la détermination du nombre de copies d'ARNm dans le cadre d'un modèle mathématique qui a été formé pour tenir compte des informations biologiques sur la structure du modèle génétique et l'homologie de la séquence, donc du gène. évolution.
"Nous avons été vraiment étonnés par l'efficacité. Quelques jours après la formation, nous avons redécouvert de nombreuses séquences de régulation connues et constaté qu'environ 50 % des caractéristiques identifiées étaient entièrement nouvelles. Ces modèles se sont parfaitement généralisés à toutes les espèces végétales sur lesquelles ils n'avaient pas été formés, ce qui en fait ils sont précieux pour analyser les génomes nouvellement séquencés", explique le Dr Szymanski.
"Et nous avons spécifiquement démontré leur application dans divers cultivars de tomates avec des données de séquençage à lecture longue. Nous avons identifié des variations spécifiques de séquences régulatrices qui expliquaient les différences observées dans l'activité des gènes et, par conséquent, les variations de forme, de couleur et de robustesse. Il s'agit d'une amélioration remarquable par rapport à associations statistiques classiquement utilisées de polymorphismes mononucléotidiques. "
L'équipe a ouvertement partagé ses modèles et fourni une interface Web pour leur utilisation. "Il est intéressant de noter que beaucoup d'efforts ont été déployés pour dégrader les performances de notre modèle. Pour éviter des résultats trop optimistes dus à la recherche de raccourcis par l'IA, il m'a fallu une plongée approfondie dans la biologie de la régulation génique pour éliminer tout biais potentiel, réduire les fuites de données et le surajustement", explique Fritz Forbang Peleke, chercheur principal en apprentissage automatique et premier auteur de l'étude, publiée dans la revue Nature Communications .
Le Dr Simon Zumkeller, co-auteur et biologiste évolutionniste de FZ Jülich, déclare :« Grâce aux analyses présentées, nous pouvons étudier et comparer la régulation des gènes dans les plantes et déduire son évolution. Pour les applications pratiques, la méthode fournit également une nouvelle base. Nous approchons de l'identification de routine d'éléments régulateurs de gènes dans les génomes végétaux connus et nouvellement séquencés, dans divers tissus et dans différentes conditions environnementales. "
Plus d'informations : Fritz Forbang Peleke et al, Apprentissage approfondi du code cis-régulateur pour l'expression des gènes dans des plantes modèles sélectionnées, Nature Communications (2024). DOI :10.1038/s41467-024-47744-0
Informations sur le journal : Communications naturelles
Fourni par l'Institut Leibniz de génétique végétale et de recherche sur les plantes cultivées