Des recherches conjointes menées par Yutaro Shuto, Ryoya Nakagawa et Osamu Nureki de l'Université de Tokyo ont déterminé la structure spatiale de divers processus d'un nouvel outil d'édition de gènes appelé « éditeur principal ». L'analyse fonctionnelle basée sur ces structures a également révélé comment un « éditeur principal » pouvait réaliser une transcription inverse, synthétisant l'ADN à partir de l'ARN, sans « couper » les deux brins de la double hélice.
La clarification de ces mécanismes moléculaires contribue grandement à la conception d’outils d’édition génétique suffisamment précis pour les traitements de thérapie génique. Les résultats sont publiés dans la revue Nature .
Le prix Nobel de chimie 2020 a été décerné à Jennifer Doudna et Emmanuelle Charpentier pour avoir développé un moyen simple et révolutionnaire de modifier l'ADN, le « modèle » des organismes vivants. Bien que leur découverte ait ouvert de nouvelles voies de recherche, l'exactitude de la méthode et les problèmes de sécurité liés à la « coupe » des deux brins d'ADN ont limité son utilisation pour les traitements de thérapie génique. A ce titre, des recherches sont en cours pour développer des outils qui ne présentent pas ces inconvénients.
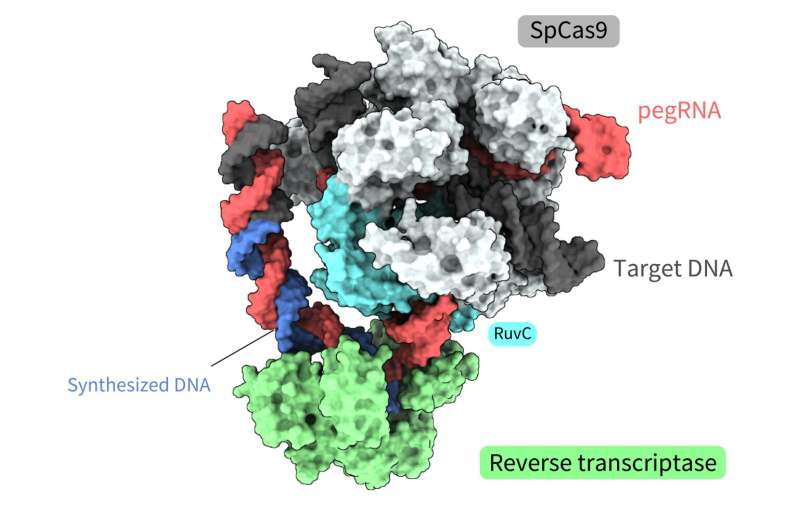
Le système d’édition principal est l’un de ces outils, un complexe moléculaire composé de deux composants. L'un des composants est l'éditeur principal, qui combine une protéine SpCas9, utilisée dans la première technologie d'édition génétique CRISPR-Cas, et une transcriptase inverse, une enzyme qui transcrit l'ARN en ADN.
Le deuxième composant est l’ARN guide d’édition principal (pegRNA), un ARN guide modifié qui identifie la séquence cible dans l’ADN et code l’édition souhaitée. Dans ce complexe, l'éditeur principal fonctionne comme un « traitement de texte », remplaçant avec précision les informations génomiques. L’outil a déjà été mis en œuvre avec succès dans des cellules vivantes d’organismes tels que des plantes, des poissons zèbres et des souris. Cependant, la manière précise dont ce complexe moléculaire exécute chaque étape du processus d'édition n'est pas claire, principalement en raison d'un manque d'informations sur sa structure spatiale.
"Nous sommes devenus curieux de savoir comment la combinaison non naturelle des protéines Cas9 et de la transcriptase inverse fonctionne ensemble", explique Shuto, le premier auteur de l'article.
L’équipe de recherche a utilisé la microscopie électronique cryogénique, une technique d’imagerie qui permet des observations à une échelle quasi atomique. La méthode nécessitait que les échantillons soient dans de la glace vitreuse pour les protéger des dommages potentiels causés par les faisceaux d'électrons, ce qui posait des défis supplémentaires.
"Nous avons constaté que le complexe de l'éditeur principal était instable dans des conditions expérimentales", explique Shuto. "Il était donc très difficile d'optimiser les conditions pour que le complexe reste stable. Pendant longtemps, nous ne pouvions déterminer que la structure de Cas9."
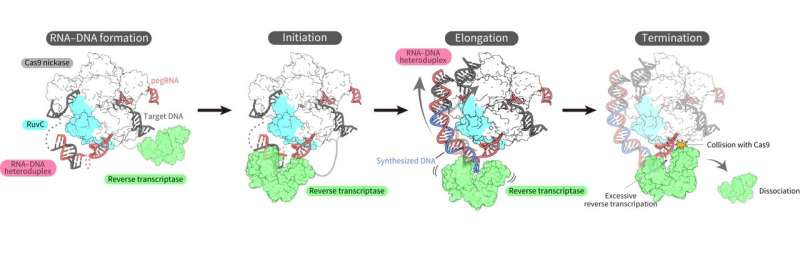
Finalement, surmontant ces défis, les chercheurs ont réussi à déterminer la structure tridimensionnelle du complexe éditeur principal dans plusieurs états lors de la transcription inverse sur l'ADN cible.
Les structures ont révélé que la transcriptase inverse s'est liée au complexe ARN-ADN formé le long de la « partie » de la protéine Cas9 associée au clivage de l'ADN, la division d'un seul brin de la double hélice. Lors de la transcription inverse, la transcriptase inverse a maintenu sa position par rapport à la protéine Cas9. Les analyses structurales et biochimiques ont également indiqué que la transcriptase inverse pourrait conduire à des insertions supplémentaires indésirables.
Ces résultats ont ouvert de nouvelles voies pour la recherche fondamentale et appliquée. Ainsi, Shuto présente les prochaines étapes.
"Notre stratégie de détermination de structure dans cette étude peut également être appliquée aux éditeurs principaux composés d'une protéine Cas9 différente et d'une transcriptase inverse. Nous souhaitons utiliser les informations structurelles nouvellement obtenues pour conduire au développement d'éditeurs principaux améliorés."
Plus d'informations : Base structurelle pour la transcription inverse guidée par pegRNA par l'éditeur principal, Nature (2024). DOI :10.1038/s41586-024-07497-8, https://www.nature.com/articles/s41586-024-07497-8
Informations sur le journal : Nature
Fourni par l'Université de Tokyo