Le lysosome est un organite intracellulaire acide crucial pour la dégradation de divers composants cellulaires. En cas de lésion de la membrane lysosomale, les hydrolases lysosomales sont libérées dans le cytosol et induisent des réponses au stress ou la mort cellulaire. Ici, nous montrons qu'un complexe protéique nouvellement identifié reconnaît les lysosomes endommagés et ubiquitine un substrat, LAMP2. Cette reconnaissance induit une autophagie sélective. Crédit :Keisuke Tabata
L'autophagie est un processus d'autodégradation que les cellules utilisent pour éliminer les composants inutiles ou endommagés. Il existe plusieurs formes d'autophagie, y compris la macroautophagie, qui est un système de dégradation en vrac utilisé pour cibler les matériaux du cytosol de la cellule vers des organites appelés lysosomes pour la dégradation enzymatique. Cependant, même les lysosomes eux-mêmes doivent parfois être dégradés. Récemment, des chercheurs de l'Université d'Osaka ont examiné les détails moléculaires spécifiques de la manière dont les lysosomes endommagés sont sélectionnés et marqués pour la clairance.
Dans un article récent publié dans Cell Reports , l'équipe a décrit un processus appelé lysophagie, la forme spécifique d'autophagie sélective responsable de l'élimination des lysosomes endommagés. Des études antérieures ont montré que des substances comme les toxines, les lipides et les cristaux de cholestérol ou d'urate peuvent rompre les lysosomes. En plus de rendre l'organite dysfonctionnel, ces dommages peuvent également induire un stress oxydatif et une inflammation pouvant entraîner le développement de maladies. Par conséquent, la cellule utilise la lysophagie pour résoudre ce problème. Cependant, les mécanismes contrôlant la façon dont les cellules peuvent reconnaître les lysosomes endommagés et les cibler pour la dégradation ne sont pas entièrement compris.
"Nous savons par des enquêtes antérieures que les lysosomes peuvent être marqués par une enzyme spécifique, SCFFBXO27 via un processus appelé polyubiquitination", explique l'un des auteurs principaux, Hirofumi Teranishi. "L'expression de SCFFBXO27 n'a été observée que dans les tissus cérébraux et musculaires, nous avons donc émis l'hypothèse qu'une autre enzyme plus omniprésente doit exister pour la lysophagie dans d'autres types de cellules."
L'équipe a utilisé des billes de polystyrène recouvertes d'un réactif qui pourrait induire des lésions endosomales et ensuite être ubiquitinées. Ils ont ensuite isolé les billes par centrifugation et ont utilisé une méthode appelée spectrométrie de masse pour identifier les protéines qui leur sont associées, réduisant finalement la liste à 123 protéines.
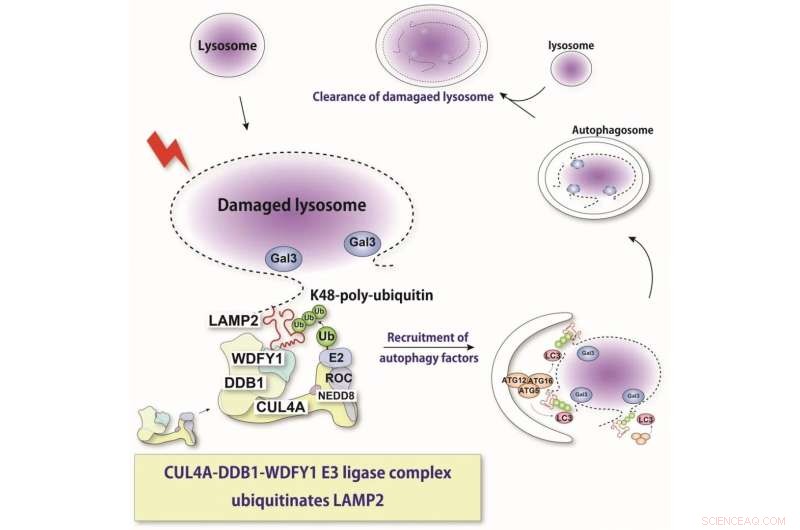
"Avec l'aide de techniques moléculaires permettant de réduire l'expression de ces différentes protéines, nous avons découvert que les protéines appelées CUL4A, DDB1 et WDFY1 composent un complexe qui répond aux dommages lysosomal", explique Maho Hamasaki, auteur principal de l'étude.
Une caractérisation plus poussée a indiqué que ce complexe agit préférentiellement pendant la lysophagie et facilite l'ajout des molécules d'ubiquitine. La protéine WDFY1 est nécessaire pour reconnaître spécifiquement les lysosomes endommagés.
"Nous nous sommes alors demandé quelle partie du lysosome est reconnue par ce complexe protéique", explique Teranishi. "De nombreuses protéines lysosomales ont été examinées, jusqu'à ce que nous découvrions que LAMP2 est celle qui est ubiquitinée par le complexe CUL4A."
L'équipe a également constaté que la présence de LAMP2 et son interaction avec WDFY1 sont essentielles pour initier le processus de lysophagie. Dans l'ensemble, ces découvertes fournissent des informations essentielles sur les mécanismes moléculaires qui sont au cœur de la lysophagie. Cela peut également aider à lutter contre les maladies dans lesquelles ce processus est dérégulé. À l'avenir, les chercheurs prévoient de déterminer des détails plus précis sur la façon dont le complexe CUL4A reconnaît LAMP2. Voie PITT :les scientifiques découvrent comment les cellules réparent le "système de recyclage" favorisant la longévité