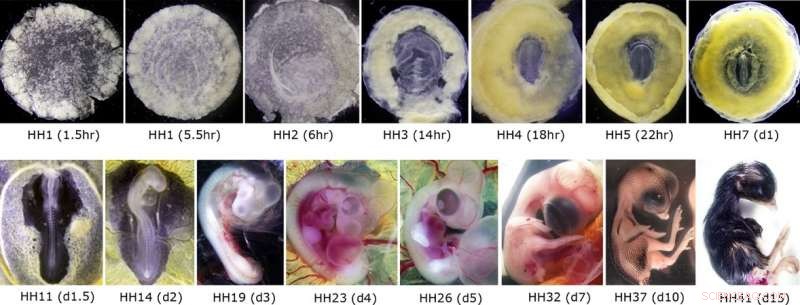
Images représentatives d'embryons de poulet utilisés pour la préparation d'ARN et le séquençage CAGE ultérieur. Les panneaux supérieurs montrent les embryons au premier jour du développement embryonnaire (21 jours au total), équivalent aux cinq premières semaines de gestation humaine (trois premières semaines de développement post-fécondation humaine). Les panneaux inférieurs montrent les embryons depuis l'établissement du système circulatoire jusqu'à environ une semaine avant l'éclosion, similaire au début du troisième trimestre du développement humain. Crédit :Professeur Guojun Sheng
Les premières recherches sur le développement des embryons humains sont impossibles pour des raisons éthiques, et est techniquement difficile à réaliser chez d'autres mammifères, rendant les ressources pour ce type de recherche extrêmement rares. Par exemple, le projet Functional Annotation of Mammalian Genome (FANTOM) 5 basé au Japon, créé en 2000, est un projet collaboratif mondial dont l'objectif est d'identifier tous les éléments fonctionnels des génomes des mammifères, mais il n'a pas de données de site de début de transcription (TSS) basées sur l'analyse de la coiffe de l'expression génique (CAGE) pour les embryons humains ou pour les embryons murins représentant les premiers stades de développement.
Pour pallier ce manque d'informations, une collaboration internationale de chercheurs du Japon (Kumamoto University &RIKEN), Russie (Université fédérale de Kazan), Espagne (Université de Cantabrie), et l'Australie (University of Western Australia) a mis en place le premier ensemble de TSS aviaires à l'échelle du génome au monde. Ces sites marquent le point de départ de la transcription et sont extrêmement importants pour la différenciation cellulaire et le développement embryonnaire jusqu'à la naissance, ou, dans le cas d'une espèce aviaire, jusqu'à l'éclosion. Alors que les oiseaux sont très différents des humains, les premiers stades de développement sont très similaires, et il a été démontré qu'environ 60 % des gènes codant pour les protéines humaines ont une correspondance biunivoque avec les orthologues du poulet (gènes qui ont évolué à partir d'un ancêtre commun).
Grâce à la technologie CAGE, une méthode très fiable pour trouver des TSS et des éléments de régulation cis (ces blocs de séquence régulant la position et la robustesse de la transcription) dans le génome sur toute la période de développement du poussin, les collaborateurs ont pu cartographier 60% de tous les TSS de poulet en développement au génome de poulet le plus récent, les 40 % restants étant probablement des promoteurs alternatifs non encore caractérisés ou des gènes d'ARN non codants. Le génome mis à jour a été ajouté à l'interactif de RIKEN, plate-forme en ligne ZENBU (Le mot japonais "zenbu" se traduit par "tous" en anglais.) sous le nom de "Chicken-ZENBU" et est gratuit pour tous.
Les chercheurs ont également montré que leur cartographie TSS à l'échelle du génome pouvait être appliquée à la technologie CRISPR-on pour activer des gènes spécifiques au cours du développement en activant avec succès le gène T pour la protéine Brachyury codant au stade de développement HH10 (environ 1,5 jour de développement après la ponte). Cette réalisation a été suivie par plusieurs autres activations de gènes réussies, montrant ainsi l'efficacité de CRISPR-on lorsqu'il est combiné avec la cartographie TSS.
"Dans le processus de développement de notre méthode de profilage de développement TSS basée sur CAGE, nous avons identifié de nouveaux facteurs de transcription et leurs modules de régulation, ainsi que plusieurs nouveaux gènes de ménage, " a déclaré le chef de projet, le professeur Guojun Sheng de l'IRCMS de l'Université de Kumamoto. " Nous espérons que ce travail et les données que nous avons ajoutées à la base de données en ligne ZENBU feront considérablement progresser la recherche sur le développement des oiseaux et des mammifères. "