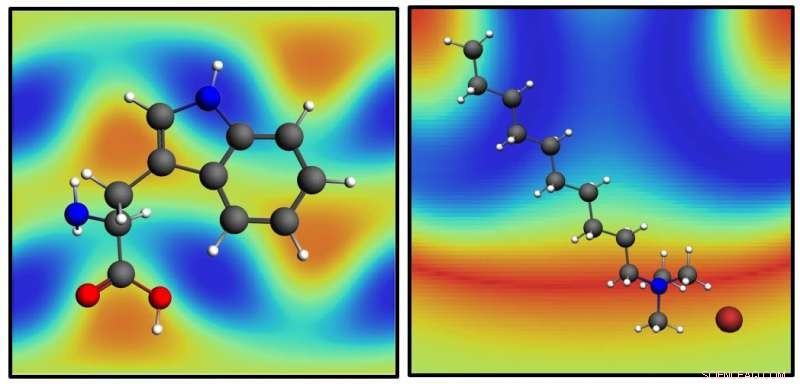
Les molécules tryptophane, la gauche, et le bromure de décyltriméthylammonium, droit, sur leurs cartes SABRE. SABRES, une nouvelle méthode d'analyse développée à l'Université Rice, est capable d'obtenir des détails structurels de molécules dans des membranes lipidiques à proximité de nanoparticules d'or sans marqueurs moléculaires. Crédit :Hafner Lab/Rice University
Cinq années de travail acharné et un peu de "chance cosmique" ont conduit les chercheurs de l'Université Rice à une nouvelle méthode pour obtenir des détails structurels sur les molécules dans les biomembranes.
La méthode du laboratoire Rice du physicien Jason Hafner combine des techniques expérimentales et informatiques et s'appuie sur les propriétés plasmoniques des nanoparticules d'or. Il tire parti de la capacité unique des nanoparticules à focaliser la lumière sur de très petites cibles.
Les chercheurs appellent leur protocole SABRE, pour l'analyse structurelle par diffusion Raman améliorée, et dire que cela pourrait aider les scientifiques qui étudient les interactions amyloïdes impliquées dans les maladies neurodégénératives, les actions neuroprotectrices des acides gras et la fonction des agents de chimiothérapie.
Les détails paraissent ce mois-ci dans le journal de l'American Chemical Society Lettres nano .
Leur méthode extrait l'emplacement de groupes chimiques spécifiques au sein des molécules en localisant leurs vibrations caractéristiques. Lorsqu'un laser active des plasmons dans les nanoparticules, il amplifie la lumière diffusée par vibration des molécules voisines, un phénomène appelé diffusion Raman améliorée en surface (SERS). L'amélioration est sensible à l'emplacement exact de la molécule par rapport à la nanoparticule.
"Les molécules peuvent vibrer de différentes manières, il faut donc attribuer un "centre de vibration" à chacun, " dit Hafner. " Si vous regardez une partie d'une molécule vibrer, vous pouvez visualiser où cela se produit, mais nous devions aussi trouver un moyen mathématique de le décrire."
Les spectres SERS sont notoirement difficiles à démêler, la méthode SABERS complète nécessite donc également des mesures spectrales non améliorées et des calculs théoriques à la fois de l'optique des nanotiges et des propriétés moléculaires, il a dit.
Hafner et son équipe ont testé leur technique sur trois structures :des molécules de tensioactifs qui viennent avec des nanotiges d'or, des molécules lipidiques qui forment des membranes sur les nanotiges d'or et le tryptophane, un acide aminé qui se dépose dans la membrane.
"Nous avons constaté que la couche de tensioactif est inclinée de 25 degrés, ce qui est intéressant car cela explique pourquoi d'autres mesures ont trouvé que la couche apparaît plus fine que prévu, " a déclaré Hafner.
Les lipides remplacent facilement les tensioactifs sur les nanotiges puisqu'ils se terminent par la même structure chimique. En comparant les vibrations de cette structure dans le groupe de tête lipidique à une double liaison dans la queue, SABERS a trouvé l'orientation et l'épaisseur correctes de la membrane bicouche lipidique. "C'est juste une chance cosmique qu'un lipide se termine par une structure parfaitement symétrique qui vibre et soit Raman active et aime s'asseoir sur une nanotige, " a déclaré Hafner.
Les chercheurs ont également utilisé SABERS pour localiser le tryptophane dans la bicouche lipidique. "C'est très lumineux, spectroscopiquement, et facile à voir, " dit-il. " Dans les structures biologiques réelles, le tryptophane n'est qu'un petit résidu attaché à une protéine beaucoup plus grosse. Cependant, le tryptophane aide à ancrer la protéine à la membrane, les chercheurs veulent donc savoir où il préfère s'asseoir."
Prochain, Hafner veut analyser des molécules plus grosses. "En principe, par des trucs spectroscopiques, nous pourrions l'appliquer à de plus grandes structures, et peut-être même trouver chaque résidu dans une protéine pour obtenir toute la structure. C'est futuriste, mais c'est là où nous pensons pouvoir aller avec, " il a dit.