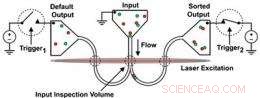
Un mélange de molécules d'ADN est poussé à travers un canal nanofluidique. Les molécules marquées avec une étiquette fluorescente déclenchent un champ électrique qui les détourne d'un côté. (Providex/Laboratoire Caighead)
(Phys.org) -- Les chercheurs en nanotechnologie de Cornell ont mis au point un nouvel outil pour étudier les changements épigénétiques de l'ADN qui peuvent provoquer le cancer et d'autres maladies :un dispositif fluidique à l'échelle nanométrique qui trie et collecte l'ADN, une molécule à la fois.
L'épigénétique fait référence aux modifications chimiques de l'ADN qui ne modifient pas le code génétique réel, mais peut influencer l'expression des gènes et peut être transmise lorsque les cellules se reproduisent. L'une des plus importantes est la méthylation de l'ADN, où les groupes méthyle - petites structures de carbone et d'hydrogène - sont ajoutés à l'ADN. Les biologistes étudient cela en précipitant chimiquement les molécules méthylées, mais ces méthodes nécessitent de gros échantillons et endommagent ou jettent souvent les molécules qu'elles sont censées trouver. La nanofluidique offre un moyen de sélectionner des molécules individuelles dans de minuscules échantillons et de les collecter pour une étude plus approfondie.
Le nouvel appareil, développé dans le laboratoire de Harold Craighead, le professeur d'ingénierie Charles W. Lake Jr., est décrit dans la première édition en ligne du 21 mai Actes de l'Académie nationale des sciences .
Le processus commence par une réaction biochimique qui attache une étiquette fluorescente aux molécules d'ADN méthylées. Ensuite, l'échantillon est conduit à travers un canal nanofluidique d'environ 250 nanomètres de diamètre - si petit que les molécules d'ADN passent une par une. Les lasers illuminent le flux et provoquent une fluorescence. Lorsqu'une molécule fluorescente passe, un détecteur déclenche un champ électrique pulsé qui pousse la molécule d'un côté juste avant que le canal ne se divise en Y. Les molécules méthylées descendent d'une branche, tout le reste en bas de l'autre.
« L'identité couleur devient un code-barres pour le traitement des molécules, " a expliqué Ben Cipriany, doctorat '12, chercheur principal sur le projet, faire une analogie avec les méthodes utilisées par la poste pour trier les colis sur un tapis roulant. « Finalement, nous pourrions utiliser plusieurs couleurs, chacun représentant une caractéristique épigénétique différente, " il ajouta.
L'appareil réagit si rapidement qu'il peut trier plus de 500 molécules par minute, les chercheurs ont dit. Le tri fluorescent n'est pas nouveau, ils ont noté, mais auparavant, cela n'a été fait qu'avec des matériaux plus gros, comme les nanoparticules ou les cellules.
"Nous avons créé une version miniaturisée qui trie les molécules individuelles et fonctionne avec une très petite quantité de matériau d'entrée, ", a déclaré Cipriany.
Pour tester leur méthode, les chercheurs ont observé une fluorescence dans chaque bras du Y. Ils ont également collecté le minuscule échantillon de molécules qui avaient été triées comme méthylées, amplifié à l'aide de la méthode PCR (réaction en chaîne polymérisée) familière aux chimistes organiques et analysé l'échantillon résultant. Les faux positifs étaient limités à environ 1 à 2 %, qui se compare avantageusement aux autres méthodes de tri, ils ont dit.
La sortie triée pourrait être dirigée vers un autre système microfluidique pour le séquençage automatisé des gènes, les chercheurs ont suggéré. La méthode pourrait également être adaptée à d'autres tâches de séparation de molécules, ils ont ajouté.
La recherche a été soutenue par les National Institutes of Health, Centre Cornell pour la génétique des invertébrés et l'Institut national du cancer. La nanofabication a été effectuée à l'installation scientifique et technologique Cornell NanoScale, financé par la National Science Foundation.
Cipriany, qui a commencé cette recherche dans le cadre de son doctorat. études à Cornell, est maintenant au centre de recherche et de développement d'IBM Semiconductor à Hopewell Junction, NEW YORK.