
Crédit :DeepMind
Les joueurs applaudissent, dire des mots comme Whoo, frapper des couteaux en plastique sur la table et profiter des meilleurs week-ends avec l'intelligence artificielle comme acte principal, grâce à l'IA déchaînée dans les jeux.
FILAIRE ROYAUME-UNI l'éditeur scientifique de, Matt Reynolds, a examiné l'impact de DeepMind sur les jalons de l'IA :« Il a dépassé les champions de Go, a battu les joueurs professionnels de StarCraft et a tourné son attention vers les échecs et le shogi."
Que les jeux continuent mais les choses sérieuses doivent briller sérieusement. En bref, nous pouvons admirer que libérer l'IA à des fins de découverte scientifique est devenu particulièrement bien vivant grâce aux recherches de DeepMind.
Les observateurs technologiques ont commenté cette semaine des documents de recherche montrant les forces de l'IA. "Au fur et à mesure que l'IA mûrit en tant que domaine (et manque de jeux vidéo à conquérir), probablement plus de ses réalisations ressembleront à celles-ci :des améliorations solides dans des domaines de recherche importants."
Un article de recherche publié dans La nature a fait l'actualité instantanée comme un article sur la façon dont l'IA peut être utilisée pour prédire le repliement des protéines. VentureBeat fait référence à des défis redoutables qui se rapprochent des objectifs.
L'article est "Prédiction améliorée de la structure des protéines en utilisant les potentiels de l'apprentissage en profondeur, " L'article a été publié en ligne le 15 décembre dans La nature .
Le système DeepMind s'appelle AlphaFold. Reynolds a fait une remarque intéressante à propos d'AlphaFold et de l'ensemble de données :« Le domaine du repliement des protéines est également bien configuré pour la formation d'agents artificiellement intelligents. un référentiel de la structure 3-D et de la constitution génétique de 150, 000 protéines, qui a été utilisé pour entraîner le système de prédiction de la structure des protéines de DeepMind, appelé AlphaFold."
L'article a exploré les réalisations de DeepMind jusqu'à présent dans l'utilisation de l'IA pour prédire le repliement des protéines. Le gros problème du repliement des protéines est que, comme Kelsey Piper dans Vox remarqué, c'est une « question cruciale pour le développement de nouveaux médicaments ».
Trois membres de l'équipe DeepMind, André Sénior, John Jumper et Demis Hassabis, a publié une entrée de blog le 15 janvier expliquant les structures des protéines, le défi du repliement des protéines et ce qu'ils voulaient accomplir.
« Comme le démontre le paradoxe de Levinthal, il faudrait plus de temps que l'âge de l'univers connu pour énumérer au hasard toutes les configurations possibles d'une protéine typique avant d'atteindre la véritable structure 3-D – pourtant les protéines elles-mêmes se replient spontanément, en quelques millisecondes. Prédire comment ces chaînes se replieront dans la structure 3-D complexe d'une protéine est ce que l'on appelle le "problème de repliement des protéines", un défi sur lequel les scientifiques travaillent depuis des décennies."
L'équipe a montré qu'elle pouvait entraîner un réseau de neurones à faire des prédictions précises "des distances entre les paires de résidus, qui transmettent plus d'informations sur la structure que les prédictions de contact."
Ils ont parlé d'optimisation via "un algorithme de descente de gradient simple pour générer des structures sans procédures d'échantillonnage complexes".
Le gros problème avec le gras des protéines est que, dit Piper, c'est une « question cruciale pour le développement de nouveaux médicaments ». Comment? Et, pourquoi les « formes » sont-elles si importantes ? "La forme que prennent les protéines prédit avec quelles autres substances elles vont interagir, la compréhension du repliement des protéines est donc cruciale pour la découverte de médicaments et pourrait même être utilisée pour développer de nouveaux procédés de fabrication, " dit Piper.
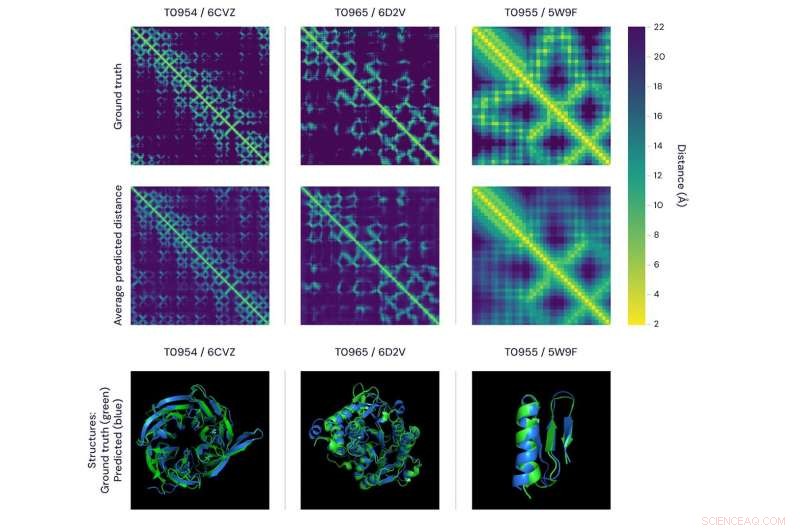
Deux façons de visualiser l'exactitude des prédictions d'alphafold. la figure du haut présente les matrices de distance pour trois protéines. La luminosité de chaque pixel représente la distance entre les acides aminés dans la séquence comprenant la protéine - plus le pixel est brillant, plus la paire est proche. Montré dans la rangée du haut sont les vrais, distances déterminées expérimentalement et, dans la rangée du bas, la moyenne des distributions de distance prédites par AlphaFold. Surtout, ceux-ci correspondent bien à l'échelle mondiale et locale. les panneaux du bas représentent la même comparaison en utilisant des modèles 3D, présentant les prédictions d'AlphaFold (bleu) par rapport aux données de vérité terrain (vert) pour les trois mêmes protéines. Crédit :DeepMind
Le blog DeepMind a déclaré que "la forme d'une protéine est censée dicter sa fonction. Une fois la forme d'une protéine comprise, son rôle au sein de la cellule peut être deviné, et les scientifiques peuvent développer des médicaments qui fonctionnent avec la forme unique de la protéine. »
AlphaFold pourrait aider les chercheurs à mieux comprendre la fonction et le dysfonctionnement des protéines.
Une citation remarquable dans VentureBeat du chef du groupe de bioinformatique de l'UCL David Jones, qui a conseillé l'équipe DeepMind sur certaines parties du projet :
"Les techniques expérimentales pour déterminer les structures des protéines sont longues et coûteuses, il y a donc une énorme demande pour de meilleurs algorithmes informatiques pour calculer les structures des protéines directement à partir des séquences de gènes qui les codent, et le travail de DeepMind sur l'application de l'IA à ce problème de longue date en biologie moléculaire est une avancée certaine."
Quels types de techniques expérimentales ont été longs et coûteux ? Le blog mentionnait la cryomicroscopie électronique, résonance magnétique nucléaire et cristallographie aux rayons X, chacun en fonction d'essais et d'erreurs, "ce qui peut prendre des années de travail, et coûtent des dizaines ou des centaines de milliers de dollars par structure protéique."
Kyle Wiggers dans VentureBeat :"Avec AlphaFold, L'équipe de recherche de DeepMind s'est concentrée sur le problème de la modélisation des formes cibles à partir de zéro sans s'appuyer sur des protéines résolues comme modèles."
Comment la nature définit le repliement des protéines :"Le repliement des protéines est le processus par lequel les protéines atteignent leur structure tertiaire fonctionnelle (native) mature, et commence souvent en co-traduction. Le repliement des protéines nécessite des chaperons et implique souvent l'établissement par étapes de structures secondaires et supersecondaires régulières, à savoir les hélices α et les feuilles , qui se replient rapidement, stabilisée par des liaisons hydrogène et des ponts disulfure, puis structure tertiaire."
De retour en juillet, avant la publication de l'article DeepMind, La nature avait déjà pris note que "la course pour relever l'un des plus grands défis de la biologie - prédire les structures 3-D des protéines à partir de leurs séquences d'acides aminés - s'intensifie, grâce à de nouvelles approches d'intelligence artificielle (IA).
L'article du blog DeepMind a déclaré qu'ils avaient rendu le code disponible (GitHub), et offert le lien, pour tous ceux qui souhaitent en savoir plus ou reproduire leurs résultats.
Dans leur article de blog, ils ont expliqué pourquoi leur système est important :« Notre système, AlphaFold... s'appuie sur des décennies de recherche antérieure utilisant de grands ensembles de données génomiques pour prédire la structure des protéines. Les modèles 3D de protéines générés par AlphaFold sont bien plus précis que tous ceux qui l'ont précédé."
Avancer, le blog a déclaré qu'un outil comme AlphaFold pourrait aider les chercheurs en maladies rares à prédire la forme d'une protéine d'intérêt rapidement et économiquement et pourrait éventuellement les aider à contribuer à la découverte efficace de médicaments, tout en réduisant les coûts associés à l'expérimentation.
La recherche sur les maladies rares n'est pas le seul domaine qui peut bénéficier de ce qu'ils ont accompli. Le blog parlait de polluants comme le plastique et le pétrole; les progrès des enzymes biodégradables permis par la conception des protéines pourraient aider à décomposer les déchets de manière plus respectueuse de notre environnement.
"En réalité, les chercheurs ont déjà commencé à concevoir des bactéries pour sécréter des protéines qui rendront les déchets biodégradables, et plus facile à traiter, " selon le blog.
© 2020 Réseau Science X