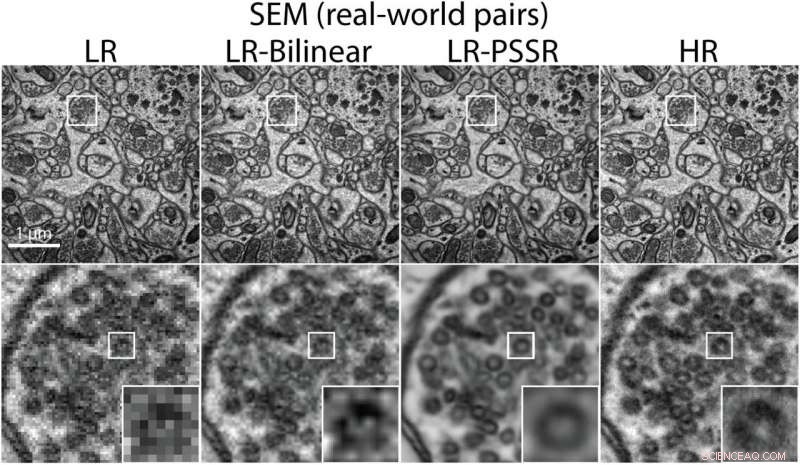
Une image montrant les versions côte à côte des captures au microscope électronique. Crédit :Institut Salk
Les descriptions des cellules du cerveau dans les manuels donnent une apparence simple aux neurones :un long axone central semblable à une épine avec des dendrites ramifiées. Pris individuellement, ceux-ci peuvent être faciles à identifier et à cartographier, mais dans un vrai cerveau, ils ressemblent plus à un tas noueux de poulpes, avec des centaines de membres entrelacés. Cela fait de la compréhension de leur comportement et de leurs interactions un défi majeur pour les neuroscientifiques.
L'imagerie microscopique est une façon pour les chercheurs de démêler notre fouillis neuronal. En prenant des photographies de couches très fines d'un cerveau et en les reconstituant sous forme tridimensionnelle, il est possible de déterminer où sont les structures et comment elles se rapportent.
Mais cela apporte ses propres défis. Obtenir des images haute résolution, et les capturer rapidement afin de couvrir une partie raisonnable du cerveau, est une tâche majeure.
Une partie du problème réside dans les compromis et les compromis que tout photographe connaît. Ouvrez l'ouverture assez longtemps pour laisser entrer beaucoup de lumière et tout mouvement provoquera un flou; prenez une image rapide pour éviter le flou et le sujet peut devenir sombre.
Mais d'autres problèmes sont spécifiques aux méthodes utilisées dans la reconstruction cérébrale. Pour un, l'imagerie cérébrale à haute résolution prend énormément de temps. Pour un autre, dans la technique largement utilisée appelée microscopie électronique à bloc série, un morceau de tissu est coupé en un bloc, la surface est imagée, une section mince est découpée et le bloc est à nouveau imagé ; le processus est répété jusqu'à la fin. Cependant, le faisceau d'électrons qui crée les images microscopiques peut en fait faire fondre l'échantillon, déformer le sujet qu'il essaie de capturer.
Manoir d'Uri, directeur du Waitt Advanced Biophotonics Core Facility du Salk Institute for Biological Studies à San Diego, est responsable de la gestion de nombreux microscopes à haute puissance utilisés par les chercheurs à travers le pays. Il est également chargé d'identifier et de déployer de nouveaux microscopes et de développer des solutions capables de résoudre les problèmes auxquels les technologies d'aujourd'hui sont confrontées.
"Si quelqu'un vient avec un problème et que nos instruments ne peuvent pas le faire, ou nous ne pouvons pas en trouver un qui peut, c'est mon travail de développer cette capacité, " dit le Manoir.
Conscient des enjeux d'imagerie auxquels sont confrontés les neuroscientifiques, il a décidé qu'une nouvelle approche était nécessaire. S'il avait atteint les limites physiques de la microscopie, Manoir raisonné, peut-être que de meilleurs logiciels et algorithmes pourraient fournir une solution.
"Il existe des approches mathématiques et informatiques sophistiquées qui ont été étudiées pendant des décennies pour supprimer le bruit sans supprimer le signal, " Manor a déclaré. "C'est là que j'ai commencé."
Travailler avec Linjing Fang, un spécialiste de l'analyse d'images chez Salk, ils ont élaboré une stratégie pour utiliser des GPU (unités de traitement graphique) pour accélérer le traitement des images microscopiques.
Ils ont commencé avec une astuce de traitement d'image appelée déconvolution qui avait été développée en partie par John Sedat, l'un des héros scientifiques de Manor et un mentor à Salk. L'approche a été utilisée par des astronomes qui voulaient résoudre des images d'étoiles et de planètes avec une résolution supérieure à celle qu'ils pouvaient obtenir directement à partir de télescopes.
"Si vous connaissez les propriétés optiques de votre système, alors vous pouvez déblurer vos images et obtenir deux fois la résolution de l'original, " il expliqua.
Ils pensaient que l'apprentissage en profondeur - une forme d'apprentissage automatique qui utilise plusieurs couches d'analyse pour extraire progressivement des caractéristiques de niveau supérieur à partir d'entrées brutes - pourrait être très utile pour augmenter la résolution des images microscopiques, un processus appelé super-résolution.
IRM, Imagerie par satellite, et les photographies ont toutes servi de cas de test pour développer approches de super-résolution, mais remarquablement peu avait été fait en microscopie. Peut-être, Pensée du manoir, la même chose pourrait être faite avec la microscopie.
La première étape de la formation d'un système d'apprentissage profond consiste à trouver un grand corpus de données. Pour ça, Manor a fait équipe avec Kristen Harris, professeur de neurosciences à l'Université du Texas à Austin et l'un des principaux experts en microscopie cérébrale.
"Ses protocoles sont utilisés dans le monde entier. Elle faisait de la science ouverte avant que ce ne soit cool, " a déclaré Manor. " Elle obtient des images incroyablement détaillées et collabore avec Salk depuis plusieurs années. "
Harris a offert à Manor autant de données qu'il en avait besoin pour la formation. Puis, en utilisant le supercalculateur Maverick du Texas Advanced Computing Center (TACC) et plusieurs jours de calcul en continu, il a créé des analogues à basse résolution des images de microscope à haute résolution et a formé un réseau d'apprentissage en profondeur sur ces paires d'images.
