
Une capture d'écran du site DIVE. Crédit :Gupta et al.
Les articles universitaires contiennent souvent des comptes rendus de nouvelles percées et de théories intéressantes liées à une variété de domaines. Cependant, la plupart de ces articles sont écrits en utilisant un jargon et un langage technique qui ne peuvent être compris que par des lecteurs familiarisés avec ce domaine d'étude particulier.
Les lecteurs non experts sont donc généralement incapables de comprendre les articles scientifiques, à moins qu'ils ne soient organisés et rendus plus accessibles par des tiers qui comprennent les concepts et les idées qu'ils contiennent. Avec ça en tête, une équipe de chercheurs du Texas Advanced Computing Center de l'Université du Texas à Austin (TACC), L'Oregon State University (OSU) et l'American Society of Plant Biologists (ASPB) ont entrepris de développer un outil capable d'extraire automatiquement les phrases et la terminologie importantes des documents de recherche afin de fournir des définitions utiles et d'améliorer leur lisibilité.
"Notre projet est motivé par le besoin d'améliorer la lisibilité des articles de revues, " Weijia Xu, qui dirigent l'équipe de TACC, a déclaré TechXplore. "C'est un effort commun entre les conservateurs biologiques, éditeurs de revues et informaticiens visant à développer un service Web capable de reconnaître et de permettre la conservation par les auteurs de la terminologie importante utilisée dans les publications de revues. La terminologie et les mots sont ensuite attachés à la fin de l'article de revue afin d'augmenter son accessibilité pour les lecteurs. »
Xu et ses collègues ont développé un cadre extensible qui peut être utilisé pour extraire des informations à partir de documents. Ils ont ensuite implémenté ce cadre au sein d'un service web appelé DIVE (Domain Information Vocabulary Extraction), en l'intégrant au pipeline de publication de revues de l'ASPB. Contrairement aux outils existants pour extraire les informations de domaine, leur cadre combine plusieurs approches, y compris l'extraction guidée par l'ontologie, extraction basée sur des règles, le traitement du langage naturel (NLP) et les techniques d'apprentissage en profondeur.

La vue d'ensemble de l'architecture du système proposée par les chercheurs. Crédit :Gupta et al.
« Les résultats atteints par les différents modèles sont ensuite stockés dans une base de données centralisée, " a expliqué Xu. " Nous avons également conçu un service Web qui permet aux utilisateurs de gérer les résultats d'extraction. Le service Web est intégré au pipeline de publication de production à l'ASPB."
Une fois la version préliminaire d'un article de revue soumise et entrée dans le pipeline de l'ASPB, le manuscrit est automatiquement envoyé à DIVE, qui le traite et produit une URL avec laquelle l'auteur pourra accéder aux résultats du traitement de DIVE. L'auteur de l'article est invité à visiter le lien fourni et à examiner les informations extraites avant de pouvoir soumettre officiellement l'article.
« L'auteur doit visiter le site DIVE pour examiner les résultats de l'extraction et approuver définitivement la liste des informations à inclure à la fin de son article, " dit Xu. " DIVE suit également les corrections de l'auteur pour améliorer les futures tâches d'extraction. Actuellement, aucun autre éditeur de revue n'a adopté une approche similaire et l'a intégrée à son pipeline de publications. »
Lors de ses analyses et lors de l'extraction des données clés des documents, le cadre développé par les chercheurs utilise plusieurs techniques. Cela lui permet de capturer plus d'informations que d'autres méthodes, tels que ABNER (A Biomedical Named Entity Recognizer), qui est un outil logiciel open source pour l'exploration de texte de biologie moléculaire qui ne peut extraire que des termes généraux (par exemple, des gènes et des protéines). Contrairement à DIVE, ABNER est uniquement basé sur des champs aléatoires conditionnels (CRF), une méthode de modélisation statistique couramment utilisée dans les applications de reconnaissance de formes et d'apprentissage automatique.
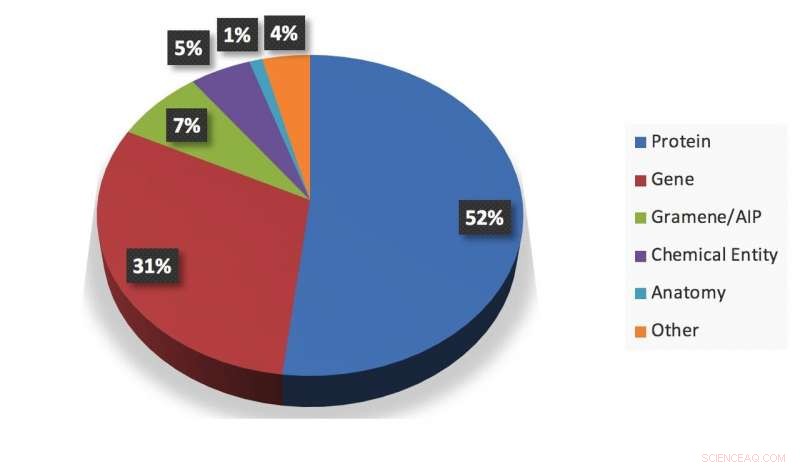
Un résumé visuel d'un instantané des informations extraites par le système. Crédit :Gupta et al.
"Une contribution majeure de notre projet est qu'il aide à construire des ensembles de données et des modèles qui peuvent déduire les intérêts de recherche des auteurs à partir de leurs publications, " Xu a déclaré. "Notre projet peut bénéficier à des communautés plus larges de chercheurs en biologie. Pour les auteurs, les extractions et l'inclusion des informations clés peuvent augmenter l'accessibilité de leurs articles."
Xu et son collègue Amit Gupta ont évalué leur framework et comparé ses performances à celles d'autres outils d'extraction d'informations, y compris ABNER. Leurs résultats ont révélé que l'utilisation de plusieurs approches, y compris l'apprentissage en profondeur, DIVE atteint des scores de précision plus élevés que les autres modèles pré-entraînés uniquement basés sur les CRF. De façon intéressante, le framework DIVE peut également être mis à jour en continu, car des modèles d'extraction supplémentaires peuvent y être ajoutés à tout moment.
L'application web DIVE permet non seulement aux lecteurs non experts de mieux comprendre les articles académiques, cela peut aussi les aider à identifier des articles alignés avec leurs intérêts. Des chercheurs, d'autre part, peut utiliser DIVE pour rester informé sur des domaines de recherche particuliers, ainsi que de se renseigner sur la nouvelle terminologie et les tendances liées à leur domaine d'intérêt. Finalement, les informations générées par l'application peuvent également guider les conservateurs de biologie dans leurs décisions et leurs processus de collecte de données.
« Nous poursuivons notre projet en explorant deux directions, " dit Xu. " D'un côté, nous étudions de nouvelles méthodes à intégrer à nos modèles d'extraction d'informations pour améliorer les performances. D'autre part, nous essayons également d'étendre notre service en l'offrant à d'autres communautés d'utilisateurs et éditeurs de revues."
© 2019 Réseau Science X