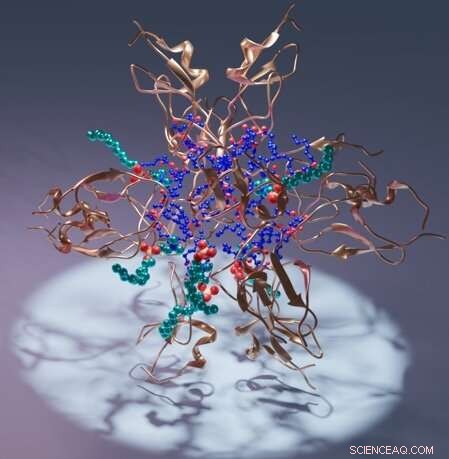
Rendu artistique d'un domaine de la protéine kinase C C1 (cuivre), de son ligand diacylglycérol (bleu) et de son détergent (cyan). Crédit :Sachin Katti.
Dans une avancée majeure pour la conception rationnelle de médicaments, une équipe Texas A&M AgriLife a décrit plusieurs structures protéiques d'un acteur crucial dans les processus cellulaires. Cette avancée pourrait apporter de nouvelles idées pour le traitement de maladies telles que la maladie d'Alzheimer, le sida, le cancer et autres.
Plus précisément, les travaux décrivent le domaine C1 de la protéine kinase C, PKC, qui aide à réguler l'activité de la protéine dans les organismes. Dans les structures, le domaine C1 s'enroule autour de différentes molécules d'intérêt thérapeutique intense, fournissant le premier guide fiable à résolution atomique pour la conception de candidats-médicaments.
Publié le 16 mai dans Nature Communications , la recherche a été dirigée par Tatyana Igumenova, Ph.D., professeure agrégée au Département de biochimie et de biophysique du Texas A&M College of Agriculture and Life Sciences. L'auteur principal du projet est Sachin Katti, Ph.D., un boursier postdoctoral travaillant avec Igumenova.
L'étude a impliqué une collaboration avec Inna Krieger, Ph.D., professeure adjointe de recherche, et James Sacchettini, Ph.D., professeur, tous deux au Département de biochimie et de biophysique.
L'une des structures protéiques les plus recherchées
Une cellule saine répond aux signaux chimiques de manière précise et complexe. Recevoir les apports chimiques de l'environnement de la cellule et les transmettre aux systèmes de contrôle centraux au sein du noyau cellulaire est la tâche de protéines spécialisées telles que PKC.
Une activité PKC inappropriée apparaît dans de nombreuses maladies humaines. En conséquence, il existe un grand intérêt à trouver des moyens d'affiner l'activité de la PKC avec des médicaments. La conception de tels médicaments offrira de nouvelles approches pour le traitement de la maladie d'Alzheimer, du sida, du cancer et plus encore.
"La protéine kinase C est l'une des protéines les plus étudiées en biologie cellulaire et en pharmacologie", a déclaré Igumenova. "Un obstacle majeur a été le manque d'informations structurelles précises pour guider les efforts de conception de médicaments."
Une complication pour la conception de médicaments est que la famille PKC compte 11 membres. Différents membres de la famille des PKC peuvent avoir des effets physiologiques opposés, de sorte qu'un candidat-médicament retenu doit être sélectif quant à la PKC qu'il cible.
Pour ce faire, les candidats-médicaments doivent s'adapter à une PKC cible comme la clé d'une serrure. Mais déterminer la structure 3D d'un "sur-commutateur" PKC - le domaine C1 - lié aux activateurs PKC n'a pas été facile.
Les structures protéiques sont généralement résolues à l'aide de la cristallographie aux rayons X. La technique consiste à utiliser des rayons X pour déterminer la position des atomes dans un cristal. Pour cette méthode, les chercheurs doivent créer des conditions dans lesquelles la protéine d'intérêt cristallise. Pourtant, des efforts intenses dans de nombreux laboratoires de recherche au cours des trois dernières décennies n'ont pas réussi à produire des cristaux de domaines C1 liés à des ligands pertinents. En raison de ce manque de progrès, plusieurs chercheurs ont déclaré la tâche impossible, a déclaré Igumenova.

Crystals of a domain of protein kinase C spontaneously formed in Katti’s NMR sample tube. Credit:Sachin Katti.
Solving a 30-year problem
Accepting the problem as challenging, Katti and Igumenova decided instead to study the molecules in solution using nuclear magnetic resonance, NMR, spectroscopy. This involved finding the right components to mimic cell membranes, where the C1 domain would encounter ligands.
"Then, one fine day, Sachin discovered crystals forming in an old NMR tube," Igumenova said. "I give all the credit to Sachin, who basically said, 'I'm going to go and test them and see if they are actually the protein.' And he was right. It gave us confidence that crystallization is possible."
In turn, Katti gives credence to the insights obtained from NMR, and a bit of luck.
"I think that's the beauty of doing research where you have to use multiple approaches," he said. "You never know when one approach is going to be useful for doing something with other approaches."
Insights from NMR and X-ray crystallography
The new protein structures, along with the team's NMR results, have already yielded interesting information. One long-standing mystery has been how C1 domains can accommodate ligands that have very different chemical structures, Igumenova said.
"Our previous NMR work indicated that the loops of the C1 domain that bind ligands are very dynamic," Igumenova said. "This C1 domain is like a PAC-man. It binds the membrane, and then it searches for a ligand. Once it finds the ligand, it latches on."
In addition, the structure shows that the ligand-binding groove has a "water-loving," or hydrophilic, surface at the bottom, and "water-repelling," or hydrophobic, surface at the top.
"If you think about a lipid molecule, the head group is hydrophilic and the tail is hydrophobic," Igumenova said. "So, when C1 domains bind lipid ligands, the patterns match."
The team's results include the structure of a C1 domain bound to its natural ligand, diacylglycerol. In addition, the team describes several other structures of C1 that include different compounds of pharmacological interest.
The work also provides a method for testing different drug candidates, Katti said.
"If you want to study fish, you want to study them in water," Katti said. "Now we know how to create a membrane-like environment where these very hydrophobic compounds can be tested for C1 binding."
Next, Katti and Igumenova plan to explore C1 domains from other PKC family members.
"It's important for us to focus on C1 domains because they have inherent differences that can be exploited to achieve selectivity," Igumenova said. "What we are finding now is that not all C1 domains are created equal." Findings reveal new mechanism of activation for ALK