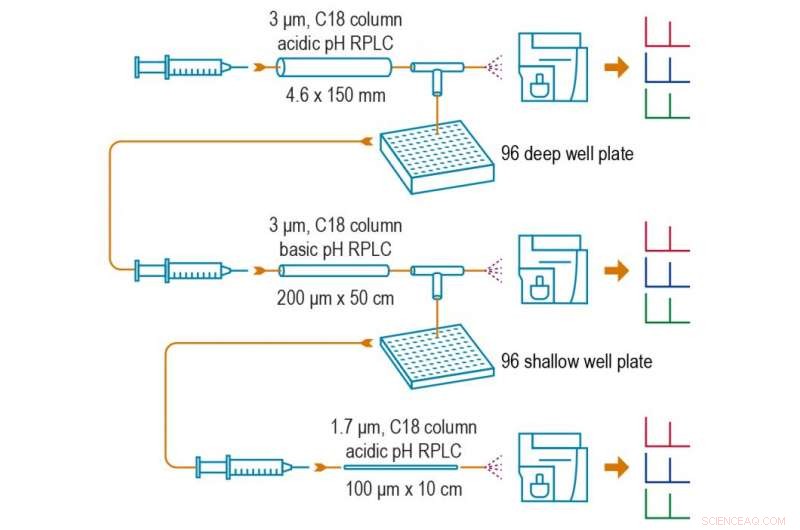
La méthode SRM en plongée profonde du PNNL offre une nouvelle façon de préparer et d'analyser des échantillons de sang, et permet de détecter des protéines qui existent à de très faibles concentrations - aussi peu que 10 picogrammes par millilitre d'un échantillon de sang. La méthode a un processus en trois étapes :1) Couper l'échantillon initial en 96 fractions et isoler la fraction avec la protéine d'intérêt, 2) Séparez uniquement cette fraction en 96 autres fractions et isolez à nouveau la fraction contenant la protéine d'intérêt, et 3) Injecter l'échantillon enrichi résultant dans un spectromètre de masse pour analyser les protéines présentes. Crédit :Laboratoire national du Nord-Ouest du Pacifique
Les gens pourraient commencer à recevoir un traitement vital contre le cancer et d'autres maladies beaucoup plus tôt à l'aide d'une nouvelle méthode d'analyse des protéines sanguines.
Un article rédigé par des chercheurs du Pacific Northwest National Laboratory décrit une nouvelle façon de préparer et d'analyser des échantillons de sang qui améliore considérablement la capacité des chercheurs à détecter les protéines qui existent à de très faibles concentrations - aussi peu que 10 picogrammes par millilitre de sang. Si petit, les concentrations difficiles à détecter sont courantes avec les protéines indicatrices de maladie, qui sont aussi appelés biomarqueurs.
De nombreux chercheurs identifient des protéines en utilisant des anticorps, Des protéines en forme de Y qui s'accrochent aux protéines qu'elles sont spécifiquement conçues pour cibler. Mais de nombreuses protéines n'ont pas d'anticorps correspondants ou les anticorps qu'elles possèdent ne sont pas assez bons pour trouver des protéines lorsque peu d'entre elles sont présentes.
Les chercheurs utilisent depuis longtemps des anticorps de cette façon, mais la spectrométrie de masse offre une alternative intéressante et plus efficace pour identifier les protéines. Pour analyser une protéine donnée dans le sang ou d'autres échantillons biologiques, les chercheurs utilisent une approche de spectrométrie de masse appelée surveillance de réaction sélectionnée, également connu sous le nom de SRM. SRM recherche spécifiquement des peptides uniques qui indiquent la présence d'une protéine ciblée.
En 2012, Les scientifiques du PNNL ont amélioré le SRM standard lorsqu'ils ont développé une nouvelle méthode appelée PRISM-SRM, qui sépare un échantillon en 96 fractions et détermine laquelle de ces fractions contient les protéines ciblées. Cela permet aux chercheurs d'analyser un échantillon enrichi avec des protéines plus ciblées via le spectromètre de masse. Par conséquent, il peut détecter des protéines qui existent à des concentrations comprises entre 1 nanogramme et 100 picogrammes par millilitre dans le sang. Le niveau de sensibilité de 100 picogrammes se produit lorsque les protéines courantes qui ne sont pas liées à la maladie sont éliminées par un processus appelé épuisement, tandis qu'une sensibilité de 1 nanogramme se produit lorsque les protéines communes ne sont pas épuisées.
Mais ils voulaient toujours détecter des protéines qui sont présentes en quantités encore plus faibles. Pour analyser un échantillon avec encore plus des protéines souhaitées à l'aide d'un spectromètre de masse, une équipe de chercheurs du PNNL dirigée par Tao Liu a créé un nouveau processus qui commence par de plus grandes quantités d'échantillons dès le départ. Après avoir coupé l'échantillon initial en 96 fractions, les scientifiques ont isolé la fraction avec la protéine ciblée de la même manière qu'ils l'avaient fait avec PRISM. Prochain, ils ont séparé juste cette fraction en 96 autres - et isolé à nouveau la fraction contenant la protéine d'intérêt.
Appelé SRM en plongée profonde, cette nouvelle approche permet de cibler les protéines présentes dans l'échantillon sanguin d'origine à des concentrations aussi faibles que 10 picogrammes par millilitre - et sans avoir besoin d'anticorps ni d'épuisement initial. Cela augmente la sensibilité de cette méthode d'environ 100, 000 fois par rapport à l'analyse SRM conventionnelle et d'environ 100 fois par rapport à PRISM-SRM.
Le SRM en plongée profonde pourrait être utilisé par les chercheurs pour identifier les protéines indicatrices de maladie dans n'importe quel échantillon biologique ou clinique avant qu'elles ne deviennent plus abondantes - y compris potentiellement diagnostiquer le cancer avant qu'une tumeur ne soit détectable par d'autres moyens. Une telle détection précoce peut augmenter les chances de sauver la vie d'un patient.