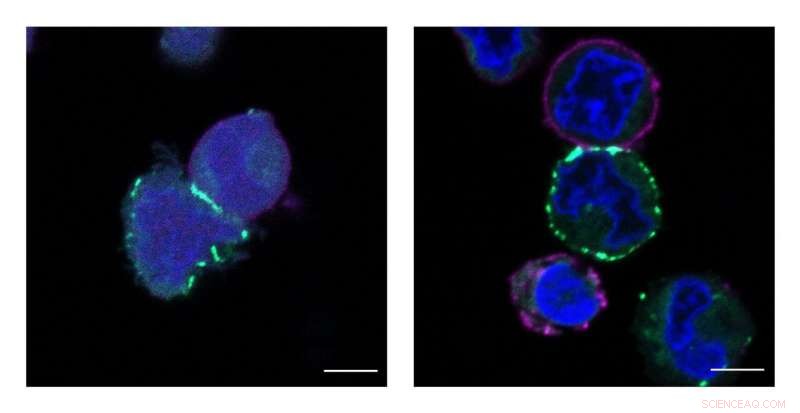
Le groupe MacMillan de Princeton a développé une nouvelle technologie appelée μMap qui identifie les voisins les plus proches des protéines sur les surfaces cellulaires. Ces images montrent des cellules étiquetées, ou surligné en vert, par le catalyseur μMap. Crédit :Tamara Reyes-Robles/Centre scientifique exploratoire Merck
Lorsque les entreprises partenaires de la Princeton Catalysis Initiative se sont entretenues il y a deux ans avec David MacMillan, ils lui ont présenté un défi biologique au cœur des potentiels médicaments anticancéreux et autres thérapeutiques :quelles protéines à la surface d'une cellule se touchent ?
Ce qu'ils voulaient était analogue à un projecteur éclairant une grotte sombre – quelque chose pour éclairer métaphoriquement une protéine et ses plus proches voisins sur la membrane cellulaire. Grand, molécules complexes, les protéines sont l'étoffe de la vie, le pivot même sur lequel tout tourne autour de nous - la façon dont nous pensons, la façon dont nous grandissons, les maladies que nous contractons. Les protéines sont capables de le déterminer en envoyant des messages à leurs voisins. Mais alors que les scientifiques pouvaient auparavant dire qui se trouvait à l'intérieur de la grotte, ils ne pouvaient pas dire qui se tenait à côté de qui, et manquaient donc de connaissances importantes sur ces communications essentielles de protéine à protéine.
Le groupe MacMillan du Département de chimie a annoncé dans le présent numéro de Science qu'ils ont développé ce projecteur.
La technologie de rupture, nommé μMap par l'équipe de chercheurs de Princeton et de scientifiques de Merck, utilise un photocatalyseur, une molécule qui, lorsqu'il est activé par la lumière, stimule une réaction chimique pour identifier les relations spatiales à la surface des cellules. Le catalyseur génère un marqueur qui marque les protéines et leurs voisins moléculaires, qui à son tour permet la cartographie précise de leur micro-environnement.
La technologie pourrait avoir un impact sur la protéomique, génomique et neurosciences, pour ne citer que quelques-uns des domaines les plus évidents. Mais les applications de la biologie fondamentale sont si vastes que MacMillan, qui est James S. McDonnell Distinguished University Professor of Chemistry de Princeton, est avide de mettre la technologie « entre les mains de tous » pour voir ce que les scientifiques d'autres domaines peuvent proposer.
« Pour les technologies dont nous disposons actuellement, le problème n'est pas de savoir si vous pouvez marquer des choses, " a-t-il dit. " Le problème est que vous pouvez étiqueter des milliers de choses et donc vous ne pouvez pas dire ce qui se passe là-bas et ce qui est juste à côté. Cela s'avère être vraiment, vraiment important parce que les molécules, les protéines ou les enzymes qui se signalent sont généralement juste à côté les unes des autres. Bien, l'état de l'art ne vous dit pas ce qui est proche."
Ils ont donc proposé une approche radicalement nouvelle.
« Nous avons fait des expériences critiques et nous avons immédiatement pu montrer que nous étiquetions les choses à une très courte distance, " a déclaré MacMillan. "Nous savons maintenant exactement ce qu'il y a dans le quartier. Et cela n'a jamais été fait auparavant. Pour la biologie, ce sera comme allumer l'interrupteur et soudainement tout voir."
Les scientifiques du Merck Exploratory Science Center (MESC) Rob Oslund et Olugbeminiyi Fadeyi, co-auteurs d'articles basés à Cambridge, Massachusetts, a déclaré que la technologie pourrait inspirer de vastes nouveaux développements en biologie. "Compte tenu du rôle important de comprendre les interactions protéiques au sein des micro-environnements cellulaires, " Oslund a dit, "cette technologie a le potentiel d'être un outil révolutionnaire pour les laboratoires universitaires et industriels des sciences de la vie du monde entier."
La Carte, micro-carte prononcée, identifie les voisins dans un rayon de 1 à 10 nanomètres autour d'une protéine particulière. (Pour référence, un cheveu humain vaut environ 100, 000 nanomètres de diamètre.) La résolution à ce niveau identifie les 10 ou 15 molécules les plus proches.
Jacob Geri, chercheur postdoctoral au Merck Center for Catalysis de l'Université de Princeton et co-premier auteur sur le Science article avec l'étudiant diplômé James Oakley et la scientifique du MESC Tamara Reyes-Robles, ladite Map le fait en utilisant la lumière bleue pour alimenter une réaction catalytique.
Voici comment cela fonctionne :le catalyseur, dans ce cas, un composé organométallique - est sélectivement attaché à l'un quelconque d'environ 40, 000 protéines à la surface d'une cellule, où il agit comme une sorte d'antenne. Lumière bleue, qui a une énergie photonique très élevée, sert de déclencheur. Quand brillait sur la cellule, cette lumière bleue est captée par l'antenne, qui convertit son énergie photonique en énergie chimique. Cette énergie latente ne refroidit pas; il ne diffuse pas; il n'erre pas sans but le long de la membrane cellulaire en peignant tout ce qu'il rencontre. Il s'assoit juste.
Sur la base d'un article publié il y a une quarantaine d'années, Le groupe de MacMillan a eu l'idée d'employer l'utilisation d'une molécule organique appelée diazirine qui est particulièrement réceptive à cette énergie latente. Lorsqu'une diazirine se déplace très près du catalyseur, à moins de 0,1 nanomètre, l'énergie chimique est transférée à la diazirine. La diazirine réagit à son tour si violemment qu'elle libère un sous-produit et devient ce qu'on appelle un carbène, une espèce "en colère" qui s'attache aux protéines voisines.
"Le catalyseur transfère tellement d'énergie que la molécule se déchire pour exposer un atome de carbone incroyablement instable, qui s'en tiendra alors à tout ce qu'il peut, " expliqua Geri.
Le catalyseur peut effectuer cette réaction chimique plusieurs fois, donc le processus se répète pour toutes les molécules localisées, protéines et enzymes. Parce que les carbènes ont une durée de vie si courte (quelques nanosecondes seulement), leur réaction permet une instantané en temps réel de toutes les molécules contiguës. Ensuite, les chercheurs peuvent assembler une carte précise du micro-environnement, ce que les scientifiques recherchaient en technologie.
"Une grande partie du mécanisme de la maladie passe par la façon dont ces cellules se parlent, et ils ne peuvent parler que s'ils se touchent, " dit Geri. " C'est pourquoi la surface de la cellule est si importante. S'ils se touchent, ils peuvent communiquer."
Il a ajouté:"Nous pouvons maintenant comprendre ce qui fait que cette communication se produise ou ce qui fait que cette communication change. Cela a vraiment été une expérience incroyable, working on this."
MacMillan's group chose two categories of human cells to investigate. One was a class of proteins that had known interactions, selected as a kind of control group to prove that their interactions could be captured by μMap. The second group was "more interesting, " said Geri. It centered on proteins called PD-L1 and PD-1, which are associated with the body's immune system and its response to cancer cells.
Normalement, sick cells like cancer cells would present as molecular interlopers that need to be cleared by the immune system. But cancer cells are deceptive, said MacMillan. They send out a "don't kill me" signal through a cloaking mechanism involving the PD-L1 and PD-1 axis. Since cancer therapies are successful partly based on their capacity to block that signal, scientists want to know more about how it is transmitted. Mapping the precise neighborhood is an essential early step. When researchers put the μMap catalyst on PD-L1 and PD-1, the molecules in their micro-environment are tagged. Protein-protein interactions that had previously been hypothesized could now be directly observed. And several correlations were detected that had never been conceived of.
"Maintenant, we don't do the cancer biology, " said MacMillan. "But we've invented this tool that can give you a lot of information about these cancer cells. We think that by using this information, you can start to target those proteins as a way to also remove interfering signals. And if you can remove those signals, you make your immune system better at going after these cancer cells."
Soon after MacMillan arrived at Princeton, he began driving research in harnessing blue LED light to perform previously impossible feats of chemistry. Merck became involved in 2006, with a seed donation toward MacMillan's research. The company has since donated additional monies, and in 2019 they announced a 10-year funding commitment towards the Princeton Catalysis Initiative, which fosters interdisciplinary collaborations to accelerate the discovery of new research areas.
"Our collaboration created a novel cellular chemistry approach leveraging photoredox catalysis to activate diazirines, an important class of organic molecules, in a temporal-controlled manner, " said Merck's Fadeyi. "Because of the routine use of diazirines within chemical biology and biology, this method will be in high demand not only for protein labeling, but for identifying the binding targets of other biomolecules to elucidate their functional roles."
He added:"The collaboration was successful due to the close interactions between Merck scientists and Dave's lab."
MacMillan likewise lauded the discovery as proof of the value of collaboration across academic and industry lines, like those envisioned when the Princeton Catalysis Initiative first came into being in 2018.
"As chemists, we don't know any good questions in biology—zero, " dit-il. " Alors, you're taking these people who know everything about biology, and they have this problem that they're trying to solve. And it's ultimately a great problem for a chemistry group. À la fois, it's not a problem that a chemistry group would ever think about because they don't know biology. You have these two different areas and you put them together and you start to realize there all these great things you can do.
"This is what I love about the social science of science, " he added. "It is absolutely a beautiful example of how it took a village to solve a problem."