Les chimistes du Boston College ont mis au point un nouveau technologie dérivée de bactéries pour incorporer des acides aminés non canoniques dans des protéines d'une large classe d'organismes, y compris les humains. Crédit :Biologie chimique cellulaire
Les protéines, les machines moléculaires qui pilotent les processus sous-jacents à la biologie, sont constituées de seulement 20 blocs de construction canoniques appelés acides aminés. Pendant près de deux décennies, les scientifiques ont cherché des méthodes pour concevoir de nouveaux acides aminés pour construire des protéines.
Une équipe de chimistes du Boston College a développé une technologie pour incorporer avec précision une gamme d'acides aminés non canoniques utiles dans des protéines fabriquées chez les eucaryotes, la vaste classe d'organismes supérieurs qui comprend les humains, l'équipe a rapporté dans le journal Biologie Chimique Cellulaire .
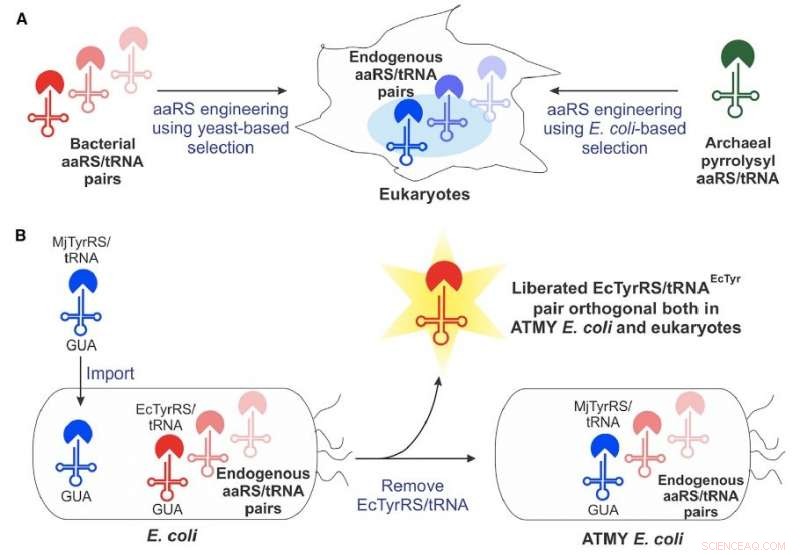
Il y a environ 15 ans, les scientifiques ont d'abord vu le potentiel d'un machinerie génétique dérivée des bactéries - connue sous le nom de paire aminoacyl-ARNt synthétase/ARNt - pour incorporer des acides aminés non canoniques dans des protéines produites dans des cellules eucaryotes. Mais la méthode a fait face à un certain nombre de restrictions techniques qui ont limité son développement à grande échelle.
L'équipe du Boston College a surmonté bon nombre de ces limitations en développant une nouvelle souche de bactérie E. coli qui permet une ingénierie facile de la paire aminoacyl-ARNt synthétase/ARNt dérivée des bactéries, selon le professeur adjoint de chimie Abhishek Chatterjee, qui a mené le projet. Cette nouvelle approche a permis l'incorporation de divers acides aminés non canoniques, dont la p-boronophénylalanine, en protéines produites dans des cellules humaines ainsi que dans la souche modifiée de E. coli .
Chatterjee a déclaré que l'équipe était surprise par la facilité de la nouvelle approche, qui est décrit dans le nouveau rapport « Ressusciter la paire bactérienne tyrosyl-ARNt synthétase/ARNt pour étendre le code génétique des deux E. coli et eucaryotes."
"Créer ce roman E. coli souche nécessitait de substituer sa paire native aminoacyl-ARNt synthétase/ARNt par une contrepartie d'un organisme différent, ce que nous pensions être très difficile, " dit-il. "Mais cela s'est avéré tout à fait faisable. Cela ouvre cette technologie complète."
Chatterjee a déclaré que l'équipe cherchait à créer une nouvelle méthode pour concevoir et surveiller les fonctions des protéines afin d'élargir la compréhension scientifique des processus qui guident les fonctions des protéines dans nos cellules.
"Des milliers de protéines sont codées dans le génome qui font de nous ce que nous sommes, mais nous savons très peu de choses sur ce processus, " dit Chatterjee. " Dans les cellules humaines, il y en a une vingtaine environ, 000 gènes codant pour des protéines. Ce qu'ils font et comment ils le font reste difficile à étudier. L'un des problèmes majeurs est que si vous voulez savoir ce qu'ils font, il faut les espionner. Vous devez attacher une sonde qui peut rendre compte de ce qui se passe."
L'introduction de telles sondes s'est avérée difficile, car le processus endommage souvent la protéine cible.
Dans n'importe quelle cellule, les protéines sont constituées de 20 acides aminés, un groupe fixe guidé dans l'ordre par des instructions génétiques.
"L'idée est que nous pouvons introduire un nouveau bloc de construction dans les protéines que la nature n'a pas - au-delà des 20 acides aminés canoniques que la nature utilise, " a déclaré Chatterjee. " Si nous pouvons le faire, nous avons la capacité d'introduire très spécifiquement une grande variété de fonctionnalités non naturelles dans n'importe quel site de pratiquement n'importe quelle protéine."
L'avantage immédiat serait d'aider les chercheurs qui perçoivent encore les mystères de la biologie cellulaire et de la fonction des protéines.
"Vous pouvez créer une protéine avec un acide aminé non canonique dans n'importe quel site choisi, le charger avec des sondes très petites et donner un signal optique qui indique où il va, " a déclaré Chatterjee. " Cela pourrait vous permettre de manipuler le fonctionnement de la protéine. Vous pourriez introduire des limites, donc quoi que fasse la protéine, il n'en peut plus. Et vous pouvez retirer la sonde en utilisant un signal externe tel que la lumière. Cette technologie ouvre de nombreuses nouvelles façons de commencer à sonder et à concevoir la fonction des protéines, ce qui serait très difficile autrement."