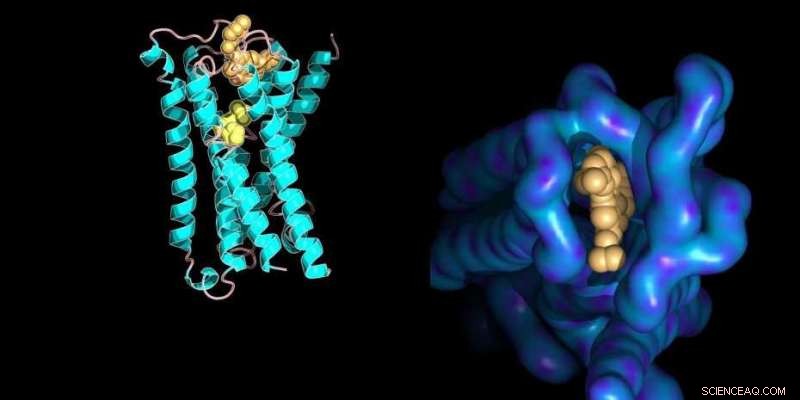
Modèle moléculaire d'un récepteur membranaire protéique lié à ses petites molécules ligands avec les détails structurels du site de liaison allostérique (à droite). Crédit :Ilaria Piazza / ETH Zurich / Entrée de la base de données PDB 4MQT
Des chercheurs de l'ETH Zurich ont utilisé une nouvelle approche pour découvrir des interactions jusqu'alors inconnues entre les protéines et les petites molécules métaboliques dans les cellules bactériennes. La technique peut également être utilisée pour tester l'effet des médicaments.
La science a maintenant une nouvelle "omique" - une interaction, qui traite des interactions entre les protéines et les petites molécules. Ayant précédemment porté sur la génomique ou la protéomique, les biologistes systémiques peuvent désormais ajouter l'interactomique protéine-métabolite au mélange. La génomique s'intéresse à l'analyse systématique de tous les gènes d'un organisme, tandis que la protéomique traite de l'ensemble des protéines au sein d'une unité biologique.
Avec l'interactomique protéine-métabolite, le groupe dirigé par Paola Picotti, Professeur de Biologie des systèmes moléculaires à l'ETH Zurich, a maintenant ajouté un autre niveau aux "omics" existants. Les chercheurs ont récemment publié une étude dans la revue Cell, dans lequel ils ont systématiquement analysé et quantifié les interactions de toutes les protéines avec des métabolites (petites molécules métaboliques) au niveau du protéome entier pour la première fois, établir les relations entre eux.
Différents sites de coupe
Les chercheurs ont montré combien de protéines et d'enzymes présentes dans une cellule bactérienne d'E. coli interagissent avec les métabolites. Faire cela, ils ont utilisé une approche connue sous le nom de protéolyse limitée (LiP), combinée à une analyse par spectrométrie de masse.
Les chercheurs ont extrait le fluide cellulaire, qui contient des protéines, à partir des cellules bactériennes. Ils ont ensuite ajouté un métabolite à chaque échantillon et lui ont permis d'interagir avec les protéines. Finalement, ils coupent les protéines en plus petits morceaux (peptides) à l'aide de "ciseaux moléculaires". Au total, les chercheurs ont ainsi testé 20 métabolites différents et leurs interactions avec les protéines.
Lorsqu'une protéine interagit avec un métabolite, qu'il se dépose dans le site actif de la protéine ou qu'il se fixe sur un autre site, la structure des protéines est altérée. Les "ciseaux moléculaires" le coupent ensuite à différents endroits de la structure d'origine, résultant en un ensemble différent de peptides.
A l'aide du spectromètre de masse, les chercheurs ont mesuré toutes les pièces présentes dans l'échantillon et ont introduit les données obtenues dans un ordinateur pour reconstruire les différences structurelles et les changements, et où dans la protéine ceux-ci sont localisés.
Des centaines de nouvelles interactions découvertes
Connaissance de l'interactome protéine-métabolite (les interactions entre protéines et métabolites et les réseaux moléculaires (de signaux) qui leur appartiennent), était auparavant très limité par rapport à ce que l'on savait des interactions entre différentes protéines ou entre protéines et ADN ou ARN. L'étude a maintenant fortement augmenté cette connaissance.
En utilisant cette approche avec E. coli, Picotti et son équipe ont découvert environ 1, 650 interactions protéine-métabolite différentes, dont plus de 1, 400 étaient auparavant inconnus. Des milliers de sites de liaison sur des protéines auxquelles les métabolites peuvent se fixer ont également été découverts. « Bien que le métabolisme d'E. coli et des molécules associées soit déjà très bien connu, nous avons réussi à découvrir de nombreuses nouvelles interactions et les sites de liaison correspondants, " dit Picotti. Cela prouve l'énorme potentiel de la méthode :" Les données que nous produisons avec cette technique permettront d'identifier de nouveaux mécanismes de régulation, enzymes inconnues et de nouvelles réactions métaboliques dans la cellule."
Le changement structurel régule l'activité
Dans leur étude, les chercheurs montrent également que les petites molécules métaboliques préfèrent se lier (et donc réguler) à des protéines dont la concentration est plus ou moins constante dans le temps. Cela suggère que la liaison des métabolites aux protéines et les changements dans les concentrations de protéines sont deux voies complémentaires par lesquelles les cellules régulent l'activité des protéines.
Les protéines peuvent être activées ou inactivées relativement rapidement par un changement structurel médié par des métabolites. "Ce type de changement structurel peut être inversé plus rapidement, " explique Picotti. Du point de vue de la cellule, cela a souvent du sens, car utiliser la voie impliquant un changement de concentration signifie que la cellule doit démanteler ou reconstruire des protéines, qui lui coûte plus de temps, énergie et ressources.
Picotti et ses collègues ont également pu montrer que de nombreuses enzymes sont moins exigeantes qu'on ne le pensait :elles peuvent apparemment se lier à plusieurs métabolites différents et les modifier chimiquement. Jusqu'à maintenant, il a été supposé que les enzymes étaient pour la plupart spécifiques à quelques molécules très similaires.
L'industrie pharmaceutique est très intéressée par la nouvelle approche, car il peut être utilisé pour tester l'interaction de médicaments avec des protéines cellulaires et identifier les cibles d'un médicament. Les chercheurs pourraient étudier à quelles protéines et à quels sites ce médicament se lie, comment il modifie leur structure et influence ainsi leur activité. Cela faciliterait et accélérerait les tests et le développement de nouveaux médicaments.