
Hiroaki Kiyokawa, MARYLAND, Doctorat, professeur de Pharmacologie et de Pathologie, était co-auteur principal d'études publiées dans Communication Nature et Avancées scientifiques . Crédit :Université Northwestern
Les chercheurs de Northwestern Medicine ont développé une technique pour cataloguer comment les cellules éliminent les protéines inutiles, un processus qui a des implications pour le cancer et les maladies du spectre autistique, selon les résultats publiés dans Communication Nature .
Défauts d'ubiquitination, ou comment les cellules se débarrassent des protéines inutiles, ont été impliqués dans diverses maladies, mais les scientifiques manquaient auparavant d'une méthode fiable pour suivre les enzymes impliquées dans ce processus d'élimination, selon Hiroaki Kiyokawa, MARYLAND, Doctorat., professeur de pharmacologie et co-auteur principal de l'étude.
Pour lutter contre cela, une équipe collaborative dirigée par Kiyokawa et Jun Yin, Doctorat., professeur agrégé de chimie à la Georgia State University, transfert d'ubiquitine orthogonal développé, une méthode de suivi des enzymes qui déclenchent l'élimination de quelles protéines. Cette technique peut permettre aux scientifiques de mieux comprendre les mécanismes de la maladie résultant d'une ubiquitination anormale, menant, espérons-le, à une meilleure thérapie, dit Kiyokawa.
L'ubiquitination signale la destruction des protéines et est facilitée par trois classes d'enzymes; E1, Enzymes E2 et E3. L'ubiquitine est liée aux trois enzymes en séquence, mais les enzymes E1 et E2 sont de simples enzymes amorces, les cellules de mammifères n'ont donc qu'un nombre limité de variétés E1 et E2. D'autre part, il existe des centaines d'enzymes E3, suggérant que chaque E3 a sa propre cible protéique unique.
"La relation entre les enzymes E3 et la protéine cible est comme un verrou et une clé, " dit Kiyokawa, également professeur de pathologie et membre du Robert. H. Lurie Comprehensive Cancer Center de l'Université Northwestern.
La diversité des protéines cibles et les interactions faibles signifient qu'il peut être difficile d'identifier clairement quelle enzyme E3 marque quelle protéine. Cependant, le transfert orthogonal d'ubiquitine (OUT) est une nouvelle solution au problème, selon Kiyokawa.
En SORTIE, les scientifiques ont conçu une voie d'ubiquitine artificielle qui imite le système natif mais ne répond qu'à l'ubiquitine mutée, qui a une étiquette spéciale qui permet aux scientifiques d'identifier la protéine qu'il cible finalement à la fin du cycle.
"Il y a deux voies dans les mêmes cellules, " dit Kiyokawa. " L'original est comme une autoroute, et le sentier aménagé est un détour."
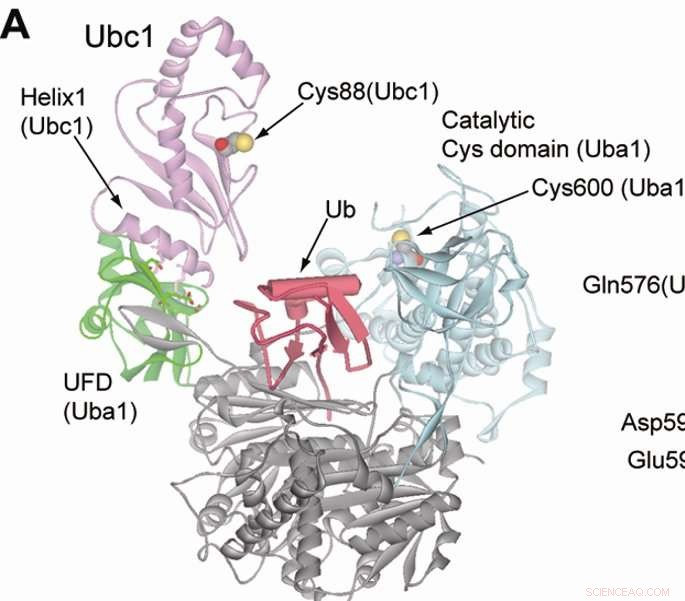
Illustration montrant l'ubiquitine passant d'une enzyme E1 à une enzyme E2. Crédit :Université Northwestern
Dans l'étude actuelle, les chercheurs ont construit une voie artificielle pour une enzyme E3 appelée E6AP, et introduit l'ubiquitine mutée et les voies dans les cellules. Après un écran protéomique, les scientifiques ont pu identifier les cibles protéiques spécifiques de l'E6AP.
"Cette étude n'est qu'une vitrine de la technologie, mais nous pensons que nous pouvons appliquer cela à bon nombre des 600 protéines E3 sans trop de tracas, " a déclaré Kiyokawa. " On sait qu'un certain nombre d'enzymes E3 sont surexprimées ou mutées dans les maladies neuronales, il y a donc un large bassin d'opportunités de recherche. »
Cependant, Il a été démontré que l'E6AP en particulier cible les protéines suppressives de tumeur lorsqu'elles sont mal activées par le virus du papillome humain, contribuant au risque accru de cancer auquel sont confrontés les patients infectés. En outre, le locus du gène qui code pour E6AP est connu pour être muté dans le syndrome d'Angelman, un trouble du spectre autistique.
"Nous ne savons toujours pas pourquoi E6AP doit être muté pour montrer les symptômes du syndrome d'Angelman, mais l'investigation de cibles spécifiques peut nous en apprendre davantage sur les mécanismes physiopathologiques de la maladie autistique, " dit Kiyokawa.
Une enquête plus approfondie sur des cibles spécifiques est déjà en cours - Kiyokawa et ses collègues ont récemment publié une autre étude impliquant OUT, cette fois dans Avancées scientifiques .
Ils ont trouvé une enzyme E3 différente, ÉBRÉCHER, était impliqué dans la dégradation de CDK4, une enzyme qui joue un rôle clé dans la prolifération cellulaire.
"Cela semble être un mécanisme qui peut arrêter le cycle cellulaire sous stress, " dit Kiyokawa. " Si ce règlement est enfreint, l'organisme peut être plus sensible au cancer."
Encore, il ne suffit pas d'identifier simplement ces cibles d'ubiquitination, selon Kiyokawa.
"Nous devons apprendre pourquoi ces cibles nouvellement définies sont sous la régulation des enzymes E3 si nous souhaitons trouver de nouvelles cibles thérapeutiques, " a-t-il dit. " Ce n'est qu'alors que nous pourrons développer des médicaments ou des traitements. "