
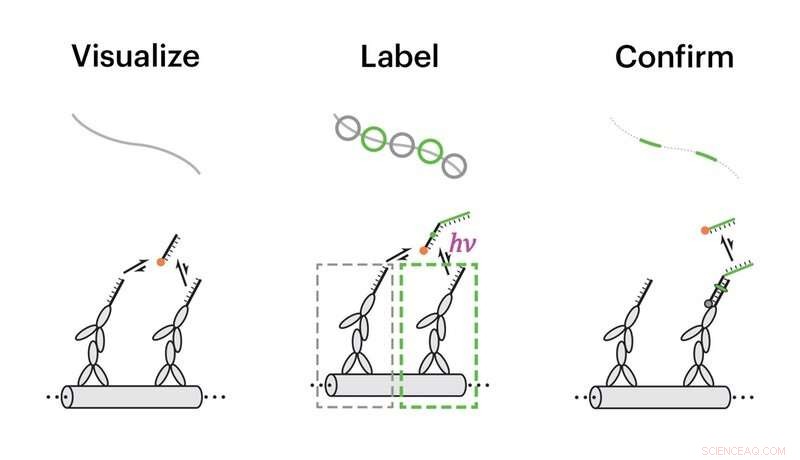
L'équipe a démontré les capacités d'Action-PAINT sur des nanostructures d'ADN synthétique exposant des sites d'accueil identiques à proximité pour les brins de l'imageur. Dans un premier temps, les sites d'amarrage ont été visualisés par microscopie super-résolution (à gauche), alors, à l'aide d'un progiciel spécial, les sites d'amarrage individuels sélectionnés manuellement ont été étiquetés en réticulant les brins de l'imageur avec un rayonnement UV (au milieu), et enfin, les événements de marquage réussis ont été vérifiés par un tour supplémentaire de microscopie à super-résolution (à droite). Crédit :Institut Wyss de l'Université Harvard
Pour comprendre comment les molécules individuelles jouent leur rôle dans les processus biologiques à l'intérieur des cellules dans lesquelles elles sont synthétisées, les chercheurs ont développé des méthodes de microscopie à super-résolution pour les visualiser au niveau d'une seule molécule. Cependant, pour enquêter sur leurs fonctions, finalement, ils aimeraient aussi pouvoir les modifier individuellement à cette haute résolution. Alors que la visualisation de molécules individuelles a fait de grands progrès ces dernières années, jusqu'à présent, il est resté difficile de les modifier directement de manière contrôlée, mode molécule à molécule.
Maintenant, signalé dans Chimie de la nature , des chercheurs du Wyss Institute for Biologically Inspired Engineering de Harvard et de la Harvard Medical School (HMS), ont développé "Action-PAINT, " une méthode qui combine l'approche de microscopie de super-résolution DNA-PAINT en temps réel de l'équipe avec une stratégie de marquage de molécule unique à un emplacement souhaité dans des nanostructures synthétiques ou des cellules intactes. Cette approche pourrait être développée davantage pour permettre aux chercheurs de stimuler ou d'inhiber la fonctions de molécules individuelles et étudier les conséquences pour les processus biologiques normaux et liés aux maladies en temps réel et en super-résolution.
"Les méthodes d'imagerie à super-résolution nous ont permis de" voir ce qui était auparavant invisible ". En couplant notre méthode de microscopie super-résolution DNA-PAINT à une approche de réticulation, nous pouvons désormais également « toucher ce qui était auparavant inaccessible » en attachant une poignée physique à des molécules individuellement observables juste au moment de la visualisation, " a déclaré Peng Yin, membre du corps professoral du Wyss Institute, Doctorat., qui a dirigé l'étude. Yin est également co-responsable de l'Initiative de robotique moléculaire de l'Institut, et professeur de biologie des systèmes à HMS.
L'équipe de Yin a développé une approche en deux étapes qui visualise d'abord des protéines uniques ou d'autres molécules à super-résolution, puis transfère un marqueur moléculaire vers le site cible souhaité. En option, les chercheurs peuvent confirmer les transferts réussis avec une série supplémentaire d'imagerie à super-résolution.
La première étape de l'imagerie d'une molécule unique repose sur la méthode DNA-PAINT qui a permis à l'équipe de déterminer dans un premier temps la localisation précise de molécules ou de caractéristiques moléculaires spatialement séparées par des distances bien inférieures à la limite de diffraction de la lumière, et donc invisible pour la plupart des microscopes. Les chercheurs ont d'abord attaché un court « brin d'amarrage » d'ADN à la cible qui sert de site de liaison pour un « brin d'imageur » complémentaire portant un colorant fluorescent. Parce que le brin de l'imageur se lie avec un taux d'activation/désactivation programmable, il produit des événements "clignotants" définis qui peuvent être observés à l'aide de microscopes standard. Pour attacher une étiquette physique à la cible, les chercheurs ont réalisé leur première étape d'imagerie en super-résolution avec un brin d'imageur légèrement plus complexe qui comprenait également un réticulant photo-inductible, qui est capable de lier chimiquement l'amarrage et le brin de l'imageur l'un à l'autre lorsqu'ils sont exposés au rayonnement UV, et une séquence reporter supplémentaire.
Pour marquer des protéines cibles individuelles dans des cellules intactes avec Action-PAINT, les brins d'amarrage sont attachés à des molécules d'anticorps qui se lient à des protéines cibles avec une spécificité élevée. Tous les événements de liaison d'anticorps au niveau des protéines cibles dans une zone définie sont visualisés avec des brins d'imagerie fluorescents complémentaires à l'aide d'une microscopie à super-résolution (à gauche), et les protéines cibles dans les zones triées sur le volet représentées par des quadrangles verts (au milieu) sont marquées par des événements de réticulation. Finalement, les événements de réticulation réussis sont confirmés à l'aide de brins d'imagerie secondaire et d'imagerie à super-résolution (à droite). Crédit :Institut Wyss de l'Université Harvard
« Un élément essentiel de notre nouvelle méthode est le contrôle précis du laser de réticulation, qui fonctionne en synchronisation avec la séquence de clignotement de super-résolution. Cela convertit vraiment notre microscopie super-résolution DNA-PAINT d'une méthode d'imagerie passive à une méthode active, permettant une interaction en temps réel entre le chercheur et les cibles moléculaires individuelles, " a déclaré le co-premier et co-auteur Mingjie Dai.
Pour activer cette nouvelle fonctionnalité, l'équipe a développé un progiciel qui leur a permis de cartographier d'abord les emplacements précis de toutes les cibles moléculaires dans une zone d'intérêt à l'aide de la méthode DNA-PAINT, puis synchroniser étroitement le clignotement d'un UV avec les événements de clignotement ultérieurs. "De cette façon, nous pourrions lier chimiquement le brin de l'imageur à des cibles moléculaires sélectionnées sur mesure, un par un, avec une résolution moléculaire - comme un peintre pose sa couleur, patch par patch dans le style pointillisme, " dit Dai, Doctorat., un ancien post-doctorant dans l'équipe de Yin, qui est actuellement chargé de recherche au département de biologie des systèmes et de développement technologique de HMS au Wyss Institute.
Démontrer l'efficacité et la sélectivité de leur approche, l'équipe a d'abord utilisé des nanostructures d'ADN synthétiques qui ont exposé des sites d'amarrage pour les brins de l'imageur dans des motifs définis. "Mettre Action-PAINT au travail, nous avons commencé par démontrer son efficacité en marquant des cibles à molécule unique à seulement 30 à 70 nm de leurs voisins identiques avec des efficacités élevées sur la cible, " a déclaré le co-premier auteur Ninning Liu, Doctorat., " puis a encore validé notre méthode en cellules fixes, où nous avons sélectionné et marqué les protéines des microtubules le long des filaments du cytosquelette, avec différents modèles personnalisés. » Liu est stagiaire postdoctoral dans l'équipe de Yin.
Les auteurs envisagent qu'Action-PAINT pourrait être développé davantage en un outil largement applicable qui, par exemple, pourrait aider à modifier directement les activités des récepteurs membranaires simples à la surface des cellules d'où ils dirigent le comportement cellulaire, ou de canaux ioniques qui contrôlent la fonction des cellules neuronales. En outre, les méthodes pourraient permettre le transfert de poignées moléculaires à des protéines uniques qui pourraient permettre leur extraction et leur purification avec d'autres protéines auxquelles elles se lient naturellement.
« Action-PAINT ajoute un autre niveau de fonctionnalité aux capacités développées par l'équipe de Peng Yin qui peuvent permettre à la fois la cartographie des interactions protéiques fonctionnelles basées sur la position moléculaire à l'intérieur des cellules individuelles, et l'analyse biochimique de ces interactions une fois ces molécules isolées, qui pourraient à l'avenir aider à identifier et/ou valider de nouvelles cibles médicamenteuses, " a déclaré le directeur fondateur du Wyss Institute, Donald Ingber, MARYLAND., doctorat