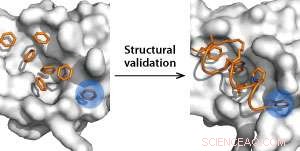
Détection de poches de liaison utilisant des molécules de benzène comme sondes (à gauche) et validation structurelle à l'aide de peptides agrafés (à droite). Crédit :Y.S. Tan et al.
À la recherche de nouvelles thérapies contre le cancer, Les chercheurs d'A*STAR ont mis au point une stratégie informatique qui déterre tous les sites de liaison ou « poches » auparavant inconnus sur les cibles médicamenteuses.
Des traitements contre le cancer plus efficaces sont susceptibles d'émerger du pipeline de développement de médicaments. La découverte de médicaments anticancéreux repose sur l'identification et la caractérisation des poches de liaison dans les protéines cibles. Typiquement, cette évaluation utilise des techniques informatiques qui reposent sur des structures protéiques statiques. Cependant, les protéines ont une flexibilité inhérente qui provoque une tendance à changer de forme au contact des médicaments. Certaines poches de reliure restent indétectables à moins qu'elles n'interagissent avec une substance appropriée et, donc, sont manqués par les simulations conventionnelles. Ces poches cachées, cependant, sont généralement des sites hydrofuges ou hydrophobes qui ne s'ouvrent que lorsqu'il y a des substances à faible polarité.
Pour y remédier, Yaw Sing Tan et Chandra Verma du Bioinformatics Institute ont développé une méthode basée sur des sondes appelée dynamique moléculaire de la cartographie des ligands (LMMD). Ils ont utilisé cette technique pour rechercher des poches de liaison cachées dans la protéine cible anticancéreuse MDM2. Les prédictions résultantes ont été validées expérimentalement par des collaborateurs de longue date du laboratoire p53 d'A*STAR et de l'Institut des sciences chimiques et de l'ingénierie ainsi que des biologistes structurels de l'Université de Newcastle, ROYAUME-UNI.
Tan explique qu'il avait initialement conçu cette méthode basée sur une sonde pour une autre protéine cible et l'avait utilisée avec succès pour trouver une poche de liaison cachée qui restait fermée dans les simulations conventionnelles. « Nous avons ensuite décidé d'appliquer cette approche à MDM2 pour voir si nous pouvions découvrir des sites de liaison auparavant inconnus qui pourraient améliorer la puissance des inhibiteurs de MDM2 existants, " il ajoute.
En utilisant des molécules de benzène comme sondes de détection de poches hydrophobes, les chercheurs ont identifié par ordinateur deux nouveaux sites de liaison sur MDM2. "Nous étions ravis de voir que ces sites se trouvent très près de la poche de liaison de la protéine suppresseur de tumeur p53, " dit Tan.
Par ailleurs, les chercheurs s'attendent à ce que les sites nouvellement découverts conduisent à des peptides agrafés plus puissants. Par conséquent, ils ont créé des peptides agrafés à partir d'analogues connus pour se lier étroitement à MDM2 et réactiver p53, et déterminé l'affinité de ces peptides pour MDM2. Leurs simulations ont montré que les peptides se liaient plus fortement à MDM2 que p53 dans les poches et correspondaient aux expériences de cristallographie biophysique et aux rayons X.
"Cette méthode pourrait être utilisée pour interroger d'autres cibles protéiques anticancéreuses afin de découvrir de nouveaux sites de liaison qui pourraient être ciblés pour l'inhibition, " dit Tan. L'équipe travaille maintenant à étendre la portée des sondes LMMD à d'autres types de ligands.