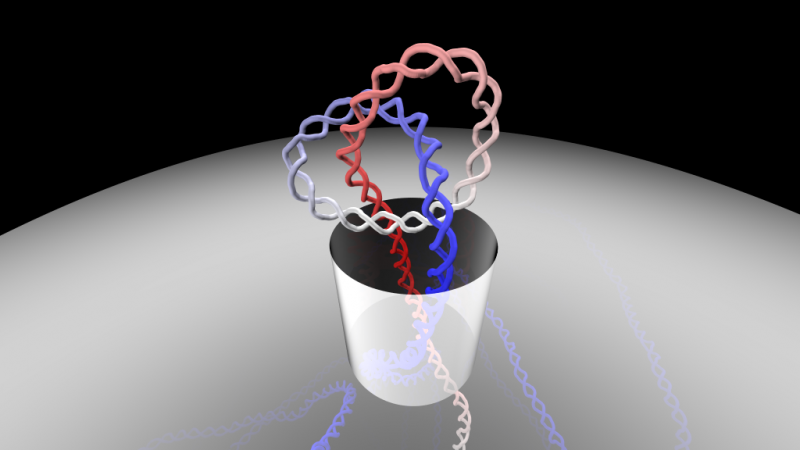
Comment de longs filaments d'ADN, qui ont une structure alambiquée et fortement nouée, parviennent à passer à travers les minuscules pores de divers systèmes biologiques ? C'est la question passionnante posée par Antonio Suma et Cristian Micheletti, des chercheurs de l'École internationale d'études avancées (SISSA) de Trieste qui ont utilisé des simulations informatiques pour étudier les options disponibles pour le matériel génétique dans de telles situations. L'étude vient d'être publiée dans PNAS , le journal de l'Académie nationale des sciences des États-Unis. Crédit :Antonio Suma, SISSA
Quiconque a déjà navigué sur un voilier sait que faire un nœud est le meilleur moyen d'attacher une corde à un crochet et d'éviter le glissement. De la même manière, les nœuds dans les fils à coudre les empêchent de glisser à travers deux morceaux de tissu. Comment, alors, peut de longs filaments d'ADN, qui ont une structure alambiquée et fortement nouée, parviennent à passer à travers les minuscules pores de divers systèmes biologiques ? C'est la question passionnante posée par Antonio Suma et Cristian Micheletti, chercheurs de l'Ecole Internationale d'Etudes Avancées (SISSA) de Trieste, qui a utilisé des simulations informatiques pour étudier la dynamique de la molécule dans de telles situations. L'étude vient d'être publiée dans PNAS , le journal de l'Académie nationale des sciences des États-Unis.
"Notre étude informatique met en lumière les dernières avancées expérimentales sur la manipulation de l'ADN noué, et ajoute des éléments intéressants et inattendus, " explique Micheletti. " Nous avons d'abord observé comment des filaments d'ADN noués traversaient de minuscules pores d'un diamètre d'environ 10 nanomètres (10 milliardièmes de mètre). Le comportement observé dans nos simulations était en bon accord avec les mesures expérimentales obtenues par une équipe de recherche internationale dirigée par Cees Dekker, qui ont été publiés il y a quelques mois seulement dans Biotechnologie naturelle . Ces expériences avancées et sophistiquées ont marqué un tournant dans la compréhension du nouage de l'ADN. Cependant, les expériences actuelles ne peuvent pas détecter comment les nœuds d'ADN passent réellement à travers le pore étroit.
"En réalité, le phénomène se produit sur une toute petite échelle spatiale inaccessible aux microscopes. C'est pourquoi notre groupe a eu recours à ce que le grand biophysicien allemand Klaus Schulten a appelé « le microscope computationnel, ' C'est, simulations informatiques."
Suma et Micheletti expliquent :« Les simulations ont révélé que le passage du nœud peut se produire de deux manières distinctes :L'une où le nœud est serré, et l'autre où le nœud est plus délocalisé. Dans les deux cas, le nœud ne passe pas seulement par le pore, mais il le fait en très peu de temps."
De plus, le nœud passe généralement dans les étapes finales de la translocation, lorsque la plupart du brin d'ADN est déjà passé. "Mais il y a quelque chose de plus qui est contre-intuitif, " précisent les auteurs. " La taille du nœud, qu'elles soient petites ou grandes, ne semble pas beaucoup affecter le temps d'obstruction des pores. Cette dernière dépend plutôt de la vitesse de translocation, lequel, à son tour, dépend de la position initiale du nœud le long du filament." Ces résultats, disent les chercheurs, devrait aider à la conception d'expériences futures sondant le nouage spontané de l'ADN, un lieu encore largement inexploré, en particulier en ce qui concerne la taille des nœuds d'ADN.
Faire progresser notre compréhension actuelle des nœuds dans les molécules biologiques est important pour clarifier leurs implications dans les contextes biologiques ainsi que dans les contextes applicatifs, comme le séquençage de l'ADN à l'aide de nanopores. Suma et Micheletti espèrent que les directions prometteuses suggérées par leur étude peuvent conduire à un profilage plus détaillé et plus précis de l'intrication dans l'ADN, ARN et protéines.