Les biologistes de l'Université Monash ont mis en lumière les mécanismes moléculaires complexes responsables de l'inactivation des gènes induite par des répétitions étendues dans une étude internationale publiée aujourd'hui dans Nature Plants. .
Ce phénomène a été lié à un certain nombre de maladies héréditaires, dont l'ataxie de Friedreich chez l'homme, et provoque des anomalies de croissance chez des plantes telles qu'Arabidopsis thaliana.
La recherche visait à comprendre le mécanisme par lequel les répétitions élargies provoquent le silençage épigénétique, une procédure essentielle pour contrôler l'expression des gènes.
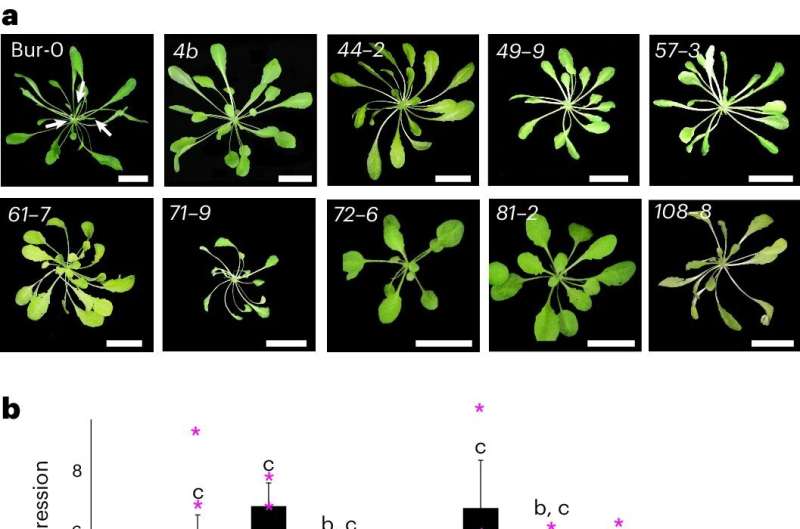
Les chercheurs ont découvert de nouveaux composants nécessaires à ce processus de mise au silence en utilisant un modèle végétal qui présente les symptômes de défauts de croissance à des températures plus élevées mais pas à des températures plus basses.
La protéase SUMO FUG1, le lecteur d'histone AL3 et la protéine chromodomaine LHP1 ont été identifiés comme les trois acteurs les plus importants, selon l'étude.
"Ces protéines se réunissent pour créer un module essentiel requis pour le silençage épigénétique induit par une expansion répétée", a déclaré l'auteur principal de l'étude, le Dr Sridevi Sureshkumar, qui dirige le département de génétique du groupe de recherche principal de l'école des sciences biologiques de l'université Monash.
"Notre recherche révèle le rôle crucial que jouent ces protéines dans l'orchestration de l'inactivation génétique déclenchée par des répétitions étendues", a déclaré le Dr Sureshkumar.
"La connaissance de ces systèmes contribue non seulement à l'avancement de notre compréhension de la biologie végétale, mais offre également un aperçu des maladies qui affectent les humains", a-t-elle déclaré.
Au cours de la recherche, des méthodes modernes de criblage génétique et des tests à deux hybrides sur levure ont été utilisés afin de déterminer que FUG1, une protéase SUMO non caractérisée, participe de manière significative au silençage épigénétique. Après une analyse plus approfondie, il a été démontré que FUG1 interagit avec AL3, qui est un lecteur d'histone connu pour se lier à des marques d'histone particulières liées à l'expression efficace des gènes.
De plus, les chercheurs ont découvert que la protéine AL3 interagit avec LHP1, une protéine chromodomaine jouant un rôle dans la dissémination des marques histones restrictives. L'inversion du silençage génique et la suppression des symptômes associés à l'expansion répétée ont tous deux été provoqués par la perte de fonction de l'un de ces composants au cours de l'expérience.
"Ces résultats mettent en évidence l'importance des modificateurs post-traductionnels et des lecteurs d'histone dans la régulation épigénétique", a déclaré le Dr Sureshkumar.
"Notre étude ouvre la voie à des recherches plus approfondies sur le rôle de ces protéines dans divers processus biologiques et maladies humaines", a-t-elle déclaré.
"Les résultats présentent non seulement des conséquences potentielles sur la santé humaine, mais contribuent également à notre compréhension de la biologie végétale, qui est déjà avancée."
Le Dr Sureshkumar, qui a dirigé cette étude internationale impliquant des institutions du Royaume-Uni, de Chine, du Canada, d'Inde et d'Australie, a déclaré que la collaboration multinationale les avait aidés à progresser dans divers aspects de cette recherche.
Le Dr Sureshkumar a déclaré que cette recherche pourrait potentiellement ouvrir la voie au développement de nouvelles techniques thérapeutiques ciblant la dérégulation épigénétique chez les personnes souffrant de maladies héréditaires.
Plus d'informations : Sridevi Sureshkumar et al, la protéase SUMO FUG1, le lecteur d'histone AL3 et la protéine chromodomaine LHP1 font partie intégrante de la répétition du silençage génique induit par l'expansion chez Arabidopsis thaliana, Nature Plants (2024). DOI :10.1038/s41477-024-01672-5
Informations sur le journal : Plantes naturelles
Fourni par l'Université Monash