
Un nouvel article publié aujourd'hui (24 avril) dans la revue Nature par une équipe internationale de 279 scientifiques dirigée par les Royal Botanic Gardens, Kew présente la compréhension la plus récente de l'arbre de vie des plantes à fleurs.
Utilisant 1,8 milliard de lettres de code génétique provenant de plus de 9 500 espèces couvrant près de 8 000 genres de plantes à fleurs connus (environ 60 %), cette incroyable réalisation jette un nouvel éclairage sur l’histoire évolutive des plantes à fleurs et leur ascension vers la domination écologique sur Terre.
Les auteurs de l'étude pensent que les données aideront les futures tentatives visant à identifier de nouvelles espèces, à affiner la classification des plantes, à découvrir de nouveaux composés médicinaux et à conserver les plantes face au changement climatique et à la perte de biodiversité.
Cette étape majeure pour la science végétale, dirigée par Kew et impliquant 138 organisations à l'échelle internationale, a été construite sur 15 fois plus de données que n'importe quelle étude comparable sur l'arbre de vie des plantes à fleurs. Parmi les espèces séquencées pour cette étude, plus de 800 n'ont jamais vu leur ADN séquencé auparavant.
La quantité considérable de données révélées par cette recherche, dont le traitement prendrait 18 ans à un seul ordinateur, constitue un grand pas en avant vers la construction d'un arbre de vie pour les 330 000 espèces connues de plantes à fleurs - une entreprise massive de la Tree of Life Initiative de Kew.
Le Dr Alexandre Zuntini, chercheur à RBG Kew, déclare :« Analyser cette quantité de données sans précédent pour décoder les informations cachées dans des millions de séquences d'ADN représentait un énorme défi. Mais cela offrait également une opportunité unique de réévaluer et d'étendre nos connaissances sur le sujet. plantez un arbre de vie, ouvrant une nouvelle fenêtre pour explorer la complexité de l'évolution des plantes. "
Déverrouiller des spécimens d'herbier historiques pour des recherches de pointe
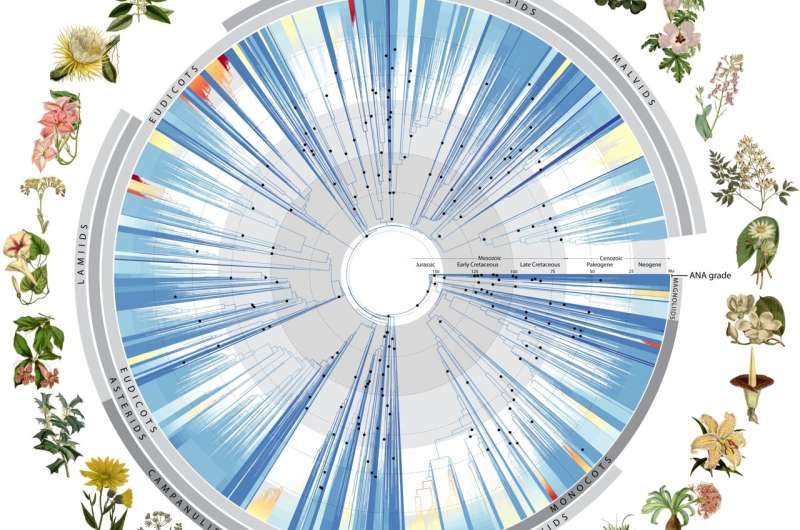
L’arbre de vie des plantes à fleurs, tout comme notre propre arbre généalogique, nous permet de comprendre les relations entre les différentes espèces. L'arbre de vie est découvert en comparant les séquences d'ADN entre différentes espèces pour identifier les changements (mutations) qui s'accumulent au fil du temps comme un enregistrement fossile moléculaire.
Notre compréhension de l’arbre de vie s’améliore rapidement parallèlement aux progrès de la technologie de séquençage de l’ADN. Pour cette étude, de nouvelles techniques génomiques ont été développées pour capturer magnétiquement des centaines de gènes et des centaines de milliers de lettres de code génétique de chaque échantillon, des ordres de grandeur supérieurs aux méthodes précédentes.
L’un des principaux avantages de l’approche de l’équipe est qu’elle permet de séquencer une grande diversité de matériel végétal, ancien et nouveau, même lorsque l’ADN est gravement endommagé. Les vastes trésors de matériel végétal séché des collections d'herbiers du monde, qui comprennent près de 400 millions de spécimens scientifiques de plantes, peuvent désormais être étudiés génétiquement.
Grâce à de tels spécimens, l'équipe a réussi à séquencer un spécimen de sandwort (Arenaria globiflora) collecté il y a près de 200 ans au Népal et, malgré la mauvaise qualité de son ADN, a pu le placer dans l'arbre de vie.
L'équipe a même analysé des plantes disparues, comme l'olivier de Guadalupe (Hesperelaea palmeri), qui n'a pas été observée vivante depuis 1875. En fait, 511 des espèces séquencées sont déjà en danger d'extinction, selon la Liste rouge de l'UICN, dont trois autres comme Hesperelaea qui sont déjà éteints.
Le professeur William Baker, responsable principal de la recherche chez Tree of Life, déclare :« À bien des égards, cette nouvelle approche nous a permis de collaborer avec les botanistes du passé en exploitant la richesse des données enfermées dans les spécimens d'herbiers historiques, dont certains ont été collectés dès le début du 19e siècle.
"Nos illustres prédécesseurs tels que Charles Darwin ou Joseph Hooker n'auraient pas pu prévoir l'importance que ces spécimens prendraient aujourd'hui dans la recherche génomique. L'ADN n'a même pas été découvert de leur vivant !
"Notre travail montre à quel point ces incroyables musées botaniques sont importants pour les études révolutionnaires sur la vie sur Terre. Qui sait quelles autres opportunités scientifiques non découvertes s'y trouvent ?"
Sur l’ensemble des 9 506 espèces séquencées, plus de 3 400 provenaient de matériel provenant de 163 herbiers répartis dans 48 pays. Du matériel supplémentaire provenant de collections de plantes du monde entier (par exemple, banques d'ADN, graines, collections vivantes) a été essentiel pour combler les principales lacunes des connaissances et apporter un nouvel éclairage sur l'histoire de l'évolution des plantes à fleurs. L'équipe a également bénéficié de données accessibles au public sur plus de 1 900 espèces, soulignant la valeur de l'approche scientifique ouverte pour la future recherche génomique.
Les plantes à fleurs représentent à elles seules environ 90 % de toute la vie végétale connue sur terre et se trouvent pratiquement partout sur la planète, des tropiques les plus humides aux affleurements rocheux de la péninsule Antarctique. Et pourtant, notre compréhension de la façon dont ces plantes ont fini par dominer la scène peu après leur origine a dérouté les scientifiques pendant des générations, y compris Charles Darwin.
Les plantes à fleurs sont apparues il y a plus de 140 millions d'années, après quoi elles ont rapidement dépassé les autres plantes vasculaires, y compris leurs plus proches parents vivants :les gymnospermes (plantes non fleuries qui ont des graines nues, comme les cycadales, les conifères et le ginkgo).
Darwin était intrigué par l’apparition apparemment soudaine d’une telle diversité dans les archives fossiles. Dans une lettre de 1879 adressée à Joseph Dalton Hooker, son proche confident et directeur de RBG Kew, il écrivait :« Le développement rapide, autant que nous puissions en juger, de toutes les plantes supérieures au cours des temps géologiques récents est un mystère abominable. »
En utilisant 200 fossiles, les auteurs ont adapté leur arbre de vie au temps, révélant ainsi l’évolution des plantes à fleurs au fil des temps géologiques. Ils ont découvert que les plantes à floraison précoce explosaient effectivement en diversité, donnant naissance à plus de 80 % des principales lignées qui existent aujourd'hui peu après leur origine.
Cependant, cette tendance a ensuite décliné à un rythme plus régulier au cours des 100 millions d'années suivantes jusqu'à une nouvelle poussée de diversification il y a environ 40 millions d'années, coïncidant avec une baisse mondiale des températures. Ces nouvelles découvertes auraient fasciné Darwin et aideront sûrement les scientifiques d'aujourd'hui à relever les défis de comprendre comment et pourquoi les espèces se diversifient.

Une collaboration véritablement mondiale
Assembler un arbre de vie aussi étendu aurait été impossible sans la collaboration des scientifiques de Kew avec de nombreux partenaires à travers le monde. Au total, 279 auteurs ont participé à la recherche, représentant de nombreuses nationalités différentes et provenant de 138 organisations dans 27 pays. Parmi eux, le consortium Genomics for Australian Plants (GAP) qui a été l'un des premiers à adopter les techniques de l'équipe et qui a travaillé en étroite collaboration avec Kew pour maximiser le nombre d'espèces de plantes australiennes dans l'arbre.
Des collaborateurs internationaux ont également partagé leur expertise botanique unique, ainsi que de nombreux échantillons de plantes précieuses du monde entier qui ne pourraient être obtenus sans leur aide. La nature globale de l'arbre est en grande partie le résultat de ce merveilleux partenariat.
Le Dr Mabel Lum, responsable de programme chez Bioplatforms Australia et du consortium GAP, déclare :« Nous sommes fiers d'être un partenaire et collaborateur majeur dans les efforts de RBG Kew visant à construire une infrastructure de recherche mondiale pour faire progresser notre compréhension de l'arbre de vie des plantes à fleurs. Cette collaboration fructueuse a souligné notre engagement à favoriser l'innovation et la collaboration dans la recherche scientifique, fournissant ainsi un tremplin pour de futures découvertes qui contribueront à façonner notre compréhension du monde naturel pour les générations à venir."

Faire bon usage de l'arbre de vie végétal
L’arbre de vie, une plante à fleurs, présente un énorme potentiel dans la recherche sur la biodiversité. En effet, tout comme on peut prédire les propriétés d’un élément en fonction de sa position dans le tableau périodique, la localisation d’une espèce dans l’arbre de vie permet de prédire ses propriétés. Les nouvelles données seront donc inestimables pour améliorer de nombreux domaines scientifiques et au-delà.
Pour ce faire, l'arbre et toutes les données qui le sous-tendent ont été rendus ouvertement et librement accessibles au public et à la communauté scientifique, notamment via le Kew Tree of Life Explorer. Les auteurs de l'étude estiment qu'un tel libre accès est essentiel pour démocratiser l'accès aux données scientifiques à travers le monde.
Le libre accès aidera également les scientifiques à tirer le meilleur parti des données, par exemple en les combinant avec l’intelligence artificielle pour prédire quelles espèces végétales peuvent contenir des molécules au potentiel médicinal. De la même manière, l’arbre de vie peut être utilisé pour mieux comprendre et prédire comment les ravageurs et les maladies affecteront les plantes du Royaume-Uni à l’avenir. En fin de compte, notent les auteurs, les applications de ces données dépendront de l'ingéniosité des scientifiques qui y accéderont.
Le Dr Melanie-Jayne Howes, responsable principale de la recherche à RBG Kew, qui n'est pas l'auteur de l'étude mais qui utilisera les données dans ses recherches, déclare :« Les produits chimiques végétaux ont inspiré de nombreux médicaments pharmaceutiques, mais ont encore un grand potentiel inexploité. contribuer à la découverte de futurs médicaments. Le défi est de savoir lesquelles étudier scientifiquement dans la recherche de nouveaux médicaments parmi environ 330 000 espèces de plantes à fleurs.
"À Kew, nous appliquons l'IA pour prédire quelles espèces végétales contiennent des produits chimiques ayant un potentiel pharmaceutique contre le paludisme. La disponibilité de ce nouvel ensemble de données vaste offre des opportunités passionnantes pour améliorer ces prédictions et ainsi accélérer la découverte de médicaments à partir de plantes contre le paludisme et d'autres maladies."

Espèces remarquables dans l'arbre de vie des plantes à fleurs
Plus d'informations : Zuntini, A. R., Carruthers, T. et al, Phylogénomique et essor des angiospermes, Nature (2024). www.nature.com/articles/s41586-024-07324-0
Informations sur le journal : Nature
Fourni par les Jardins botaniques royaux de Kew