Une étude a exploité le génome de la pêche, créant des réseaux de co-expression génique (GCN) à grande échelle qui prédisent les fonctions des gènes et rationalisent le processus de sélection des pêches. Cette approche innovante aborde la tâche complexe d'identification des gènes liés aux caractères de sélection souhaitables chez les pêches.
La génétique des pêches est confrontée à des défis tels que de longues périodes intergénérationnelles et des systèmes de transformation génétique limités, ce qui rend l'identification et la validation des gènes complexes. Les approches traditionnelles ne parviennent souvent pas à prédire les fonctions des gènes en raison de ces limitations.
Les réseaux de co-expression génique (GCN), qui cartographient les relations entre les expressions géniques, offrent une solution prometteuse. En tirant parti du principe « coupable par association », les GCN peuvent prédire les fonctions des gènes sur la base de modèles d'expression partagés. En raison de ces défis, il existe un besoin urgent d'outils innovants tels que les GCN pour améliorer notre compréhension de la génétique des pêches.
Des chercheurs de l'Institut de Recerca i Tecnologia Agroalimentàries (IRTA) et du Centre de Recerca en Agrigenòmica (CRAG) ont publié une étude dans la revue Horticulture Research le 2 janvier 2024. L'étude présente un nouvel outil qui exploite les GCN pour prédire les fonctions des gènes dans les pêches, révolutionnant potentiellement la sélection fruitière.
L'étude a créé quatre GCN à partir de 604 bibliothèques Illumina RNA-Seq et a évalué leurs performances à l'aide d'un algorithme d'apprentissage automatique basé sur le principe du « coupable par association ». Parmi les réseaux, COO300 a montré les meilleures performances, englobant 21 956 gènes. Pour valider COO300, les chercheurs ont mené deux études de cas.
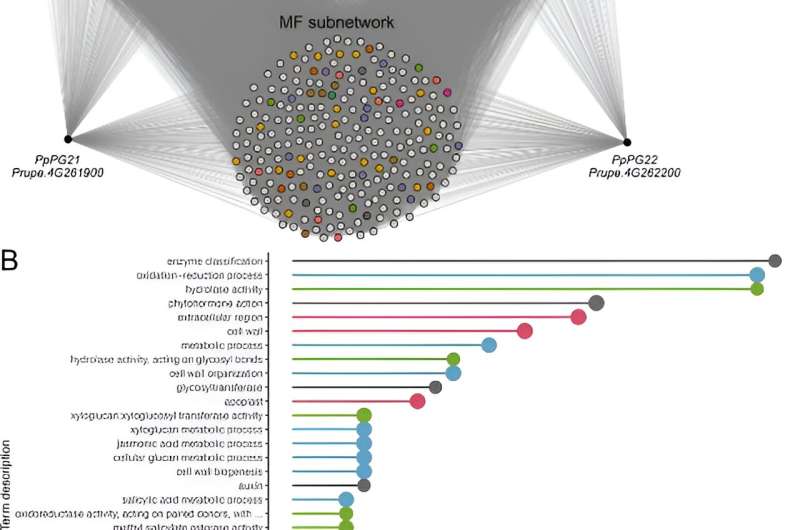
Dans la première étude de cas, les gènes PpPG21 et PpPG22, impliqués dans le ramollissement des pêches, ont été analysés. Les gènes co-exprimés ont formé le réseau de chair fondante (MF), enrichi de termes liés à l'organisation de la paroi cellulaire et à la signalisation des phytohormones. Le réseau MF comprenait des gènes associés au désassemblage de la paroi cellulaire et aux phytohormones liées à la maturation, démontrant la capacité du COO300 à prédire avec précision les fonctions des gènes.
Dans la deuxième étude de cas, le facteur de transcription PpMYB10.1, qui régule l’accumulation d’anthocyanes, a été examiné. Le réseau de couleur des fruits (FC) a identifié des gènes impliqués dans le métabolisme et la régulation des anthocyanes. En comparant le réseau FC avec les orthologues de la vigne, les chercheurs ont identifié des réseaux de régulation conservés, validant ainsi les capacités prédictives de COO300.
Le Dr Iban Eduardo, chercheur principal au CRAG, a déclaré :« Le développement du réseau COO300 représente une avancée significative dans la génétique des pêches. En prédisant avec précision les fonctions des gènes, cet outil améliore non seulement notre compréhension de la biologie des pêches, mais ouvre également la voie à des stratégies de sélection plus ciblées et plus efficaces."
L’étude offre aux sélectionneurs un outil puissant pour valoriser les variétés de pêches. En décodant les fonctions des gènes, le PeachGCN v1.0 et ses scripts, accessibles à tous, devraient faire progresser la saveur, la longévité et la nutrition des pêches. Cette connaissance génomique est un signe avant-coureur de changement, signalant une évolution vers une sélection basée sur les données dans l'agriculture.
Plus d'informations : Felipe Pérez de los Cobos et al, Exploration des réseaux de coexpression génétique à grande échelle chez le pêcher (Prunus persica L.) :un nouvel outil pour prédire la fonction des gènes, Recherche horticole (2024). DOI : 10.1093/hr/uhad294
Informations sur le journal : Recherche horticole
Fourni par l'Université agricole de NanJing