De nouvelles découvertes réalisées par des chercheurs de l'Université de Buffalo révèlent que l'édition de l'ARN pourrait jouer un rôle plus important dans la biologie humaine et dans le développement de maladies humaines qu'on ne le pense généralement.
Dans un article publié dans Communications Biology Le 4 mai, les auteurs de l'étude concluent que les travaux fournissent la preuve d'une compréhension plus nuancée du fonctionnement de la biologie moléculaire, notamment en termes de réponse des protéines et des gènes aux défis environnementaux.
Les sites d’édition d’ARN provoquent des modifications des polymorphismes mononucléotidiques (SNP) – le type de variation génétique le plus courant chez l’homme – dans les gènes transcrits qui peuvent conduire au codage de différentes protéines. L'expression de ces différentes protéines peut à son tour être un facteur dans un large éventail de maladies humaines.
"Des phénomènes externes peuvent modifier votre biologie et modifier la façon dont vous réagissez aux défis auxquels vous êtes confrontés, qu'il s'agisse de maladies infectieuses ou de changement climatique", explique Peter L. Elkin, MD, auteur correspondant, professeur et directeur du Département d'informatique biomédicale. à la Jacobs School of Medicine and Biomedical Sciences de l'UB.
Avec ce type de défis, poursuit-il, l'interféron est régulé positivement, ce qui stimule la libération des enzymes APOBEC, une famille d'enzymes qui modifient l'ARN.
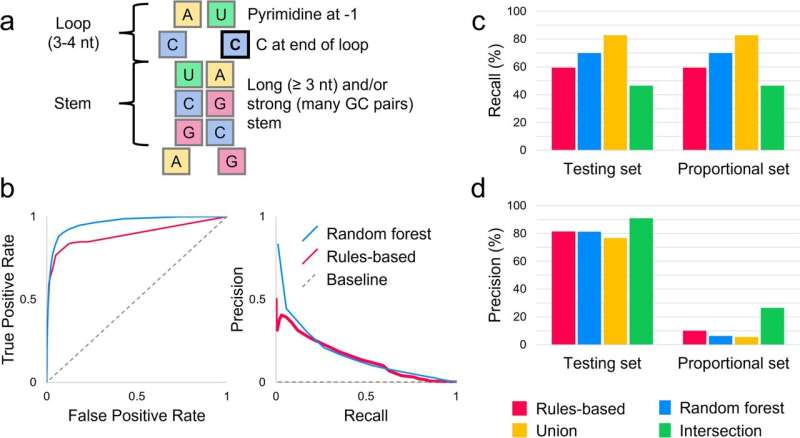
La recherche se concentre sur cette famille d’enzymes. L'équipe d'Elkin a découvert que 4,5 % des SNP entraînant un type spécifique de changement dans l'ADN sont des sites probables pour l'édition de l'ARN. Si ce type d'édition d'ARN se produit ne serait-ce qu'une fraction de ces sites, affirment les auteurs, cela pourrait avoir des effets significatifs sur la santé humaine.
Des travaux antérieurs ont montré une certaine relation entre l'édition d'ARN médiée par APOBEC et certaines maladies auto-immunes et neurologiques. Elkin ajoute qu'environ 70 % des maladies neurologiques génétiques impliquent au moins un site d'édition d'ARN.
"Nous avons découvert que certaines zones spécifiques du génome sont sensibles à ce type de changements", dit-il, ce qui suggère que les sites d'édition d'ARN pourraient donc avoir un effet significatif sur les maladies génétiques.
Les chercheurs ont été encore plus intrigués lorsque leurs collaborateurs du Roswell Park Comprehensive Cancer Center ont découvert que les sites d'édition de l'ARN étaient impliqués dans des cancers particuliers.
"J'ai commencé à penser que les sites d'édition d'ARN pourraient jouer un rôle plus important dans la maladie qu'on ne le pensait auparavant", explique Elkin.
Pour découvrir tous les sites d'édition d'ARN dans le génome, l'équipe a développé un outil informatique appelé RNAsee, qu'elle a validé grâce à l'apprentissage automatique.
L'outil a révélé que 22,7 % des sites d'édition potentiels trouvés par RNAsee étaient étiquetés comme probablement pathogènes ou pathogènes, contre seulement 9,2 % étiquetés comme probablement bénins ou bénins.
Les auteurs écrivent que cette découverte démontre que le type d'édition d'ARN qu'ils ont étudié "a une possibilité substantielle d'influencer négativement la santé humaine".
"Ce travail ajoute une autre dimension à notre compréhension de la façon dont notre protéome se développe", explique Elkin, faisant référence à l'ensemble complet des protéines exprimées par les humains.
La prochaine étape du travail, ajoute-t-il, concernerait les chercheurs qui travaillent sur la génétique de maladies spécifiques pour lesquelles des sites d'édition d'ARN ont été découverts, pour donner suite à ces découvertes.
"J'espère que les chercheurs poursuivront leurs expériences fondamentales sur les gènes d'intérêt là où nous avons trouvé des sites d'édition d'ARN", déclare Elkin.
Plus d'informations : Melissa Van Norden et al, Les implications de l'édition d'ARN C-to-U médiée par APOBEC3 pour les maladies humaines, Biologie des communications (2024). DOI :10.1038/s42003-024-06239-w
Informations sur le journal : Biologie des communications
Fourni par l'Université de Buffalo