Lorsque l’on tente de mesurer des structures moléculaires avec une précision nanométrique, chaque bruit apparaît dans les données :quelqu’un qui passe devant le microscope, de minuscules vibrations dans le bâtiment et même la circulation à l’extérieur. Une nouvelle technique de traitement supprime le bruit des données du microscope optique en temps réel, permettant aux scientifiques de suivre des molécules individuelles plus de 10 fois plus précisément qu'auparavant.
Une équipe de chercheurs en bioingénierie de l'Université de l'Illinois à Urbana-Champaign a introduit un algorithme appelé maximisation adaptative des intersections, ou AIM, qui supprime le bruit haute fréquence des données du microscope optique à super-résolution beaucoup plus rapidement que les méthodes standard et donne des résolutions d'image beaucoup plus élevées. .
L’algorithme permettra aux scientifiques d’étudier les systèmes chimiques et biologiques beaucoup plus facilement et avec précision qu’auparavant. Cette recherche a été publiée dans la revue Science Advances.
"Au début, nous voulions simplement développer un algorithme rapide car notre laboratoire produisait trop de données pour que les algorithmes traditionnels puissent les gérer, mais nous avons découvert que l'AIM pouvait également atteindre une précision inférieure au nanomètre, ce qui est du jamais vu dans notre domaine", a déclaré Hongqiang Ma. , professeur-chercheur en bio-ingénierie et auteur principal de l'étude. "De plus, il ne nécessite pas une immense puissance de calcul comme les outils traditionnels. Il peut fonctionner sur un ordinateur portable. Nous voulons en faire un outil plug-and-play pour tous les utilisateurs de microscopes."
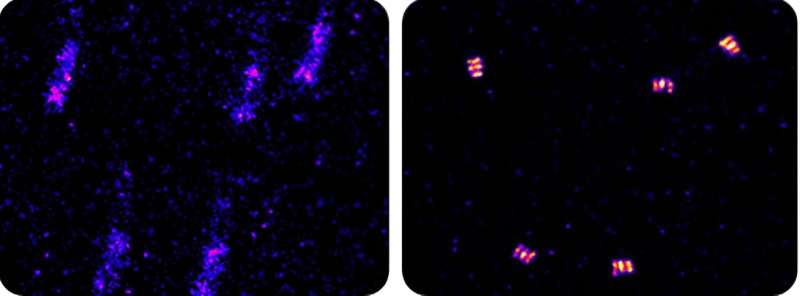
Au cours des dernières décennies, la technique de microscopie de localisation d'une seule molécule a permis aux scientifiques de visualiser des structures à l'échelle moléculaire, surpassant ainsi ce que l'on pensait être une limitation fondamentale des microscopes optiques. Cependant, elle est limitée en pratique par un bruit incontrôlable, ou « dérive », qui brouille les images et empêche la microscopie à super-résolution d'atteindre sa résolution la plus élevée.
"La localisation d'une seule molécule utilise en fait un instrument assez simple, mais la partie délicate qui a un impact réel sur la résolution de l'image est la dérive", a déclaré Yang Liu, professeur de bio-ingénierie et responsable du projet. "De nombreux chercheurs ne suppriment que la dérive des basses fréquences. La suppression de la dérive des hautes fréquences (les vibrations infimes causées par le bruit ambiant) nécessite beaucoup de calculs et nécessite beaucoup de temps et de ressources."
Les méthodes standard pour supprimer la dérive sont basées sur les corrélations mathématiques entre les images. Selon Liu, les microscopes de son laboratoire génèrent un si grand volume de données d'images que les méthodes de corrélation d'images prennent des jours, même avec des ressources de calcul intensif.
AIM compare également les images adjacentes, mais il procède en plaçant chaque point de données au centre d'un cercle (défini par la précision de localisation) et en recherchant des points à l'intérieur de ce cercle dans d'autres images. Les points qui se chevauchent dans le « rayon d'intersection » sont condensés en une seule localisation. Ensuite, le processus est répété une fois de plus avec les points condensés. Ces étapes utilisent des ressources informatiques minimales et sont plus rapides que le temps d’acquisition d’une caméra de microscope. Ainsi, des images corrigées de la dérive peuvent être produites en temps réel.
Les chercheurs ont testé AIM en utilisant à la fois des données simulées et des structures appelées origami ADN qui présentent des caractéristiques bien définies. L'algorithme a réussi à localiser les structures et le degré de précision, inférieur à 1 nanomètre, s'est avéré bien supérieur aux méthodes standard de corrélation d'images, environ 10 nanomètres.
Le laboratoire de Liu intégrera l'AIM aux techniques de microscopie à haut débit en cours de développement pour améliorer la détection des maladies. Cependant, Liu pense également que l’algorithme trouvera des applications dans la biologie et la bio-ingénierie. "C'est un outil rapide et facile à utiliser, et nous voulons le rendre largement accessible à l'ensemble de la communauté", a-t-elle déclaré. "Nous rendons notre logiciel accessible au public. Nous voulons que les gens obtiennent une meilleure résolution d'image grâce à ce simple post-traitement."
Plus d'informations : Hongqiang Ma et al, Vers une nanoscopie à haut débit sans dérive grâce à la maximisation adaptative des intersections, Science Advances (2024). DOI :10.1126/sciadv.adm7765
Informations sur le journal : Progrès scientifiques
Fourni par le Grainger College of Engineering de l'Université de l'Illinois