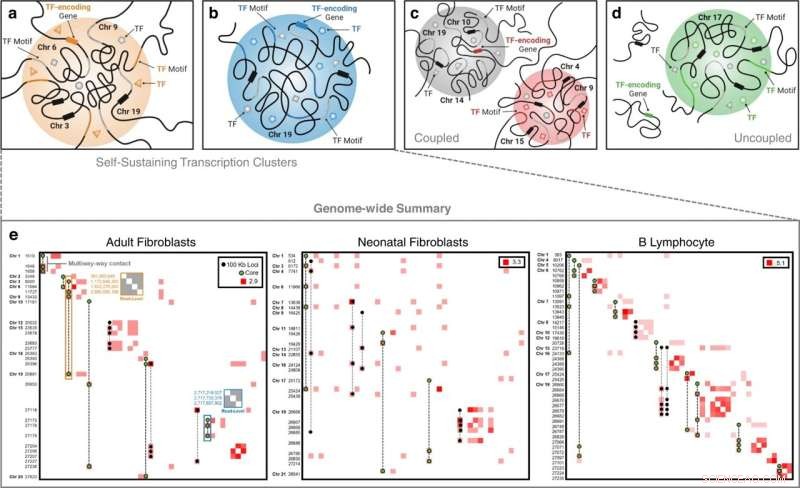
Classes de clusters de transcription. Dans un cluster de transcription auto-entretenu, un TF et le gène codant pour ce TF sont tous deux présents. Les exemples inter- et intra-chromosomiques en (a) et (b), respectivement, illustrent ce phénomène où en a on voit le TF d'intérêt (triangle orange) circuler au niveau du cluster, son motif de liaison présent sur la chromatine (partie orange ), et son gène correspondant exprimé (rectangle orange sur le chromosome 6). Les formes grises représentent des TF supplémentaires avec des motifs de liaison (partie grise de la chromatine) au niveau du cluster. Les rectangles noirs sur les chromosomes 3, 9 et 19 représentent des gènes supplémentaires présents dans le cluster. c Une classe de clusters de transcription indépendante des analogues où nous observons une liaison TF (carré rouge) à un cluster de transcription (cluster rouge) et son gène correspondant exprimé dans un cluster de transcription séparé (cluster gris), mais pas dans le même cluster. d Une classe de clusters de transcription indépendante des analogues où nous observons une liaison TF (cercle vert) à un cluster de transcription (cluster vert) et son gène correspondant exprimé mais pas dans un cluster de transcription. e Clusters de transcription auto-entretenus spécifiques à un type de cellule à l'échelle du génome extraits de données de contact multivoies et décomposés en matrices de contact Hi-C à une résolution de 100 kb. Les fréquences de contact sont transformées en log pour une meilleure visualisation. Les fréquences le long de la diagonale indiquent une interaction entre deux ou plusieurs locus multidirectionnels uniques qui relèvent du même bac de 100 kb. Les étiquettes d'axe sont des coordonnées bin non contiguës de 100 kb dans l'ordre chromosomique. Les contacts multidirectionnels qui composent les clusters de transcription auto-entretenus sont superposés. Les contacts multivoies avec des loci de couleur verte représentent des grappes de transcription « principales » - des grappes de transcription contenant un régulateur maître et son analogue de gène. Un exemple de carte de contact au niveau de la lecture pour le cluster de transcription auto-entretenu inter-chromosomique FOXO3 est indiqué par la case en surbrillance orange dans la matrice de contact des fibroblastes adultes et une carte de contact au niveau de la lecture pour le cluster de transcription auto-entretenu intra-chromosomique ZNF320 est indiqué par la case en surbrillance bleue. Les valeurs le long de l'axe gauche de ces matrices de contact au niveau de la lecture sont les positions des paires de bases des locus de contact dans le génome. Crédit :Nature Communications (2022). DOI :10.1038/s41467-022-32980-z
La troisième loi de l'écrivain de science-fiction Arthur C. Clarke dit que "toute technologie suffisamment avancée est indiscernable de la magie".
Indika Rajapakse, Ph.D., est une croyante. L'ingénieur et mathématicien se retrouve désormais biologiste. Et il pense que la beauté du mélange de ces trois disciplines est cruciale pour comprendre le fonctionnement des cellules.
Son dernier développement est une nouvelle technique mathématique pour commencer à comprendre comment le noyau d'une cellule est organisé. La technique, que Rajapakse et ses collaborateurs ont testée sur plusieurs types de cellules, a révélé ce que les chercheurs ont appelé des grappes de transcription auto-entretenues, un sous-ensemble de protéines qui jouent un rôle clé dans le maintien de l'identité cellulaire.
Ils espèrent que cette compréhension exposera les vulnérabilités qui peuvent être ciblées pour reprogrammer une cellule afin d'arrêter le cancer ou d'autres maladies.
"De plus en plus de biologistes du cancer pensent que l'organisation du génome joue un rôle énorme dans la compréhension de la division cellulaire incontrôlable et si nous pouvons reprogrammer une cellule cancéreuse. Cela signifie que nous devons comprendre plus en détail ce qui se passe dans le noyau", a déclaré Rajapakse, professeur agrégé de calcul. médecine et bioinformatique, mathématiques et génie biomédical à l'Université du Michigan. Il est également membre du U-M Rogel Cancer Center.
Rajapakse est l'auteur principal de l'article, publié dans Nature Communications . Le projet a été dirigé par un trio d'étudiants diplômés avec une équipe interdisciplinaire de chercheurs.
L'équipe a amélioré une technologie plus ancienne pour examiner la chromatine, appelée Hi-C, qui cartographie les parties du génome proches les unes des autres. Il peut identifier les translocations chromosomiques, comme celles qui se produisent dans certains cancers. Sa limitation, cependant, est qu'il ne voit que ces régions génomiques adjacentes.
La nouvelle technologie, appelée Pore-C, utilise beaucoup plus de données pour visualiser comment toutes les pièces du noyau d'une cellule interagissent. Les chercheurs ont utilisé une technique mathématique appelée hypergraphes. Pensez :diagramme de Venn en trois dimensions. Il permet aux chercheurs de voir non seulement des paires de régions génomiques qui interagissent, mais la totalité des relations complexes et qui se chevauchent à l'échelle du génome au sein des cellules.
"Cette relation multidimensionnelle que nous pouvons comprendre sans ambiguïté. Cela nous donne un moyen plus détaillé de comprendre les principes organisationnels à l'intérieur du noyau. Si vous comprenez cela, vous pouvez également comprendre où ces principes organisationnels s'écartent, comme dans le cancer", a déclaré Rajapakse. "C'est comme mettre trois mondes ensemble - la technologie, les mathématiques et la biologie - pour étudier plus en détail à l'intérieur du noyau."
Les chercheurs ont testé leur approche sur des fibroblastes néonataux, des fibroblastes adultes biopsiés et des lymphocytes B. Ils ont identifié des organisations de clusters de transcription spécifiques à chaque type cellulaire. Ils ont également découvert ce qu'ils ont appelé des clusters de transcription auto-entretenus, qui servent de signatures transcriptionnelles clés pour un type de cellule.
Rajapakse décrit cela comme la première étape d'une vision plus large.
"Mon objectif est de construire ce type d'image sur le cycle cellulaire pour comprendre comment une cellule passe par différentes étapes. Le cancer est une division cellulaire incontrôlable", a déclaré Rajapakse. "Si nous comprenons comment une cellule normale change avec le temps, nous pouvons commencer à examiner les systèmes contrôlés et non contrôlés et trouver des moyens de reprogrammer ce système." Dévoiler les mystères de la structure du génome dans le noyau de la cellule humaine à l'aide d'une simulation informatique 3D