
Crédit :Université d'État de Pennsylvanie
Votre corps en regorge - 100 000 milliards de microbes dans votre intestin, poumons, bouche, et la peau. Votre maison en grouille - dans les toilettes et les éviers, sur les tables et les chaises, dans le tapis, et sur votre chien. Même le sol sur lequel tu te tiens regorge d'innombrables bactéries, champignons, protozoaires, algues, et des virus, tous microscopiques, font tous partie d'une communauté d'organismes interagissant les uns avec les autres et avec l'environnement. Ces communautés et les environnements avec lesquels elles interagissent sont appelés « microbiomes, " et notre compréhension croissante d'eux change la façon dont nous traitons les maladies, cultiver, et créer des produits du quotidien.
Cela change notre vision de la nature.
Les scientifiques de Penn State et du College of Agricultural Sciences sont à la pointe de la recherche sur les microbiomes. Ils sondent les tripes humaines et animales pour apprendre comment les micro-organismes influencent la santé, et ils explorent les sols pour découvrir comment les microbes profitent aux cultures. Grâce à ce travail, les scientifiques acquièrent une appréciation de la complexité de la vie microbienne sur Terre. Et ils explorent également les avantages potentiels et les défis que présentent ces créatures.
Le voyage humain dans le royaume du microscopique a commencé en 1657, quand Antoni van Leeuwenhoek, un drapier vivant à Delft, Hollande, a révélé qu'il avait découvert, en regardant à travers son microscope simple qu'il s'est lui-même créé, minuscules "animalcules" vivant dans l'eau du lac.
Dans une lettre à la Royal Society of London nouvellement formée, van Leeuwenhoek a écrit que ces animalcules étaient « si petits, à mes yeux, que je jugeais que même si 100 de ces tout petits animaux étaient étendus les uns contre les autres, ils ne pouvaient pas atteindre la longueur d'un grain de sable grossier.. . c'était merveilleux à voir."
La découverte des micro-organismes par Van Leeuwenhoek a constitué un changement sans précédent dans la compréhension humaine du monde naturel. Tout à coup, la vie sur Terre est devenue à la fois considérablement plus complexe et extrêmement merveilleuse. Et encore, plus de 350 ans plus tard, nous ne savons pas qui sont beaucoup de ces créatures et ce qu'elles font.
"Selon à qui vous parlez, nous ne savons peut-être rien sur 99% des micro-organismes dans l'environnement, " dit Carolee Bull, professeur et chef du Département de phytopathologie et de microbiologie environnementale. Mais, Elle ajoute, un peu comme le changement qui s'est produit après que van Leeuwenhoek a découvert des microbes, une révolution scientifique des temps modernes promet de révéler les identités et les activités des communautés de micro-organismes qui ont un si grand impact sur nos vies.
Cette fois, l'outil qui rend tout cela possible est le séquençage à haut débit, également connu sous le nom de séquençage de nouvelle génération. Disponible pour la première fois en l'an 2000 et largement utilisé depuis la dernière décennie, la technologie peut déterminer l'ordre des nucléotides - l'As, Ts, Gs, et Cs—pour des centaines de milliers de molécules d'ADN d'une myriade d'espèces simultanément. La technologie permet d'apprendre les identités de chaque espèce présente dans un petit échantillon de, par exemple, l'eau de l'étang. Séquençage simple de l'ADN, d'autre part, est beaucoup plus limité dans ses capacités.

Les champignons sont retirés du lit, qui est composé de trois couches :le compost, le boîtier, et le champignon. Scientifique invité, Les recherches de Fabricio Rocha Vieira portent sur des matériaux alternatifs pour la couche d'enveloppe. Sa principale question est de savoir comment les matériaux alternatifs modifient l'interaction des microbes avec le champignon. Comprendre cela a des implications sur les rendements des cultures et aussi sur la sécurité alimentaire. Crédit :Michelle Bixby
"Le concept de microbiomes n'est pas particulièrement nouveau, " dit Bull. " Mais avec le séquençage à haut débit, la boîte noire de la microbiologie s'éclaire enfin, " dit Bull. "Cela nous montre que les microbes ne travaillent pas seuls dans le vide. Au lieu, ils font partie d'une communauté dans laquelle l'environnement, d'autres organismes, et les microbes s'influencent et se répondent."
Protéger la qualité de l'eau et restaurer les sols
Le microbiome du sol est un sujet d'étude compliqué. Une cuillère à café de terre contient probablement un milliard de cellules bactériennes individuelles, peut-être 500, 000 fragments de champignons, des milliers de protozoaires, et qui sait combien de virus, dit Mary Ann Bruns, professeur agrégé de science et gestion des écosystèmes. Et des bactéries seules, Elle ajoute, il peut en exister 10, 000 à 20, 000 espèces différentes.
Bruns utilise le séquençage à haut débit, entre autres outils, pour taquiner cette "soupe à l'ADN, " comme elle l'appelle, qui est contenu dans le sol. Ses recherches sur les microbes responsables du cycle de l'azote à l'échelle du terrain s'inscrivent dans le cadre plus large de la réduction du transport des nutriments vers les zones mortes côtières. "Globalement, la moitié de l'azote contenu dans les engrais appliqués aux cultures n'est pas absorbé par les cultures, " dit-elle. " Au lieu de cela, il s'infiltre dans les eaux souterraines ou s'écoule dans les sédiments. Une grande partie de cet azote finit par se retrouver dans le golfe du Mexique et la baie de Chesapeake, où il bouleverse les écosystèmes. Je suis intéressé par la façon dont nous pouvons arrêter ce processus à la source, comment nous pouvons réduire le gaspillage de nos méthodes d'application et de gestion de l'azote."
La clé, elle dit, consiste à trouver des communautés microbiennes plantes-sol qui conservent l'azote, car elles sont responsables d'une grande partie du cycle des nutriments dans le sol. Par exemple, les microbes convertissent l'ammonium en nitrate, qui est la forme d'azote la plus facilement perdue. "Certains microbes sont responsables de nombreuses réactions biochimiques dans le sol qui entraînent une faible efficacité, " dit Bruns. " Dans l'agriculture traditionnelle, nous avons contrecarré ce problème de perte d'azote en ajoutant un montant d'assurance. C'est moins cher et plus facile d'en ajouter plus que d'essayer de comprendre comment éviter les pertes en premier lieu."
La bonne nouvelle est que si certains microbes favorisent la perte d'azote du sol, d'autres espèces sont capables de fixer l'azote de l'atmosphère et de le maintenir en place. Grâce au séquençage à haut débit, Bruns et l'un de ses étudiants diplômés ont caractérisé un mélange de deux souches étroitement apparentées de cyanobactéries et de plusieurs espèces de bactéries non photosynthétiques qui forment rapidement des biofilms sur les sols pour réduire l'érosion et le ruissellement. Ce "consortium, " elle dit, pourrait être ajouté au sol agricole pour fixer le carbone et l'azote et aider les nutriments à rester en place, réduisant ainsi le besoin d'applications supplémentaires d'azote et protégeant les environnements en aval de la pollution par l'azote.
Bruns dit qu'elle aimerait voir les entreprises s'intéresser à ces produits et les développer à des fins agricoles. Par exemple, de tels produits microbiens pourraient être appliqués, avec les amendements, pour récupérer les terres endommagées, un autre domaine où la recherche sur le microbiome fournit des informations. Bruns et son associée postdoctorale Claudia Rojas ont utilisé le séquençage de nouvelle génération pour montrer une augmentation des champignons mycorhiziens et des rhizobiums bénéfiques dans les terrains miniers revégétalisés après avoir ajouté des quantités modérées de compost et de chaux.
Terrence Bell, maître de conférences en phytopathologie et microbiologie environnementale, s'intéresse également à la création de consortiums microbiens qui peuvent être ajoutés aux sols pour améliorer leur fonction. Il se concentre sur la restauration des sols qui ont perdu leur activité microbienne en raison de la surutilisation d'engrais et de produits chimiques. "Certains sols agricoles sont épuisés des micro-organismes essentiels nécessaires à la croissance des cultures parce qu'ils ont été traités avec des engrais chimiques pendant si longtemps, " il dit.
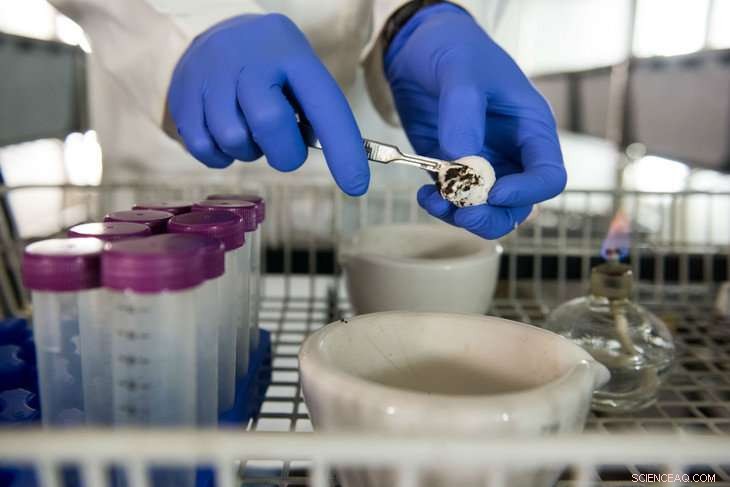
Les champignons sont tranchés pour collecter des échantillons à tester. Crédit :Michelle Bixby
Grâce à des études expérimentales, Bell et l'un de ses étudiants de premier cycle ont démontré que c'était le cas. Ils ont appliqué différents types d'amendements nutritifs aux sols et ont constaté que les sols exposés à de grandes quantités de phosphore chimique ont subi des changements dans la composition du microbiome, y compris une réduction de la diversité microbienne, qui semblait avoir un impact sur la croissance des cultures.
"Notre prochaine étape consiste à déterminer si nous pouvons contrer ce problème en réintroduisant la diversité microbienne dans ces environnements, " dit Bell.
Comme pour les recherches de Bruns, la tâche nécessite l'utilisation des dernières techniques de séquençage. « Cela dépend vraiment des outils disponibles, et le fait que cela ne fait encore que moins d'une décennie que le séquençage à haut débit a été largement utilisé dans notre domaine, " dit Bell. Avec ces technologies, "nous nous concentrons sur les fonctions, " ajoute Bruns. " La plupart des organismes ne sont ni pathogènes ni pathogènes, ils sont juste là à attendre les bonnes conditions pour devenir actifs. Notre objectif est de le comprendre et d'utiliser les connaissances à notre avantage."
Vérification de l'intestin
L'environnement de l'intestin humain comprend des dizaines de milliards de micro-organismes individuels, pesant collectivement près de 4,5 livres. Beaucoup de ces microbes sont impliqués dans la réduction du risque de cancer, dépression, obésité, et même l'autisme. En réalité, Les « bonnes » bactéries deviennent si populaires pour leurs effets positifs sur la santé que la nouvelle industrie des probiotiques a généré plus de 35 milliards de dollars de bénéfices en 2015. Mais certaines espèces sont liées à des problèmes.
Pensez à l'obésité. Aux États-Unis seulement, 34 percent of adults and 15 to 20 percent of children and adolescents are obese. Dans les années récentes, researchers and clinicians have been turning to the gut microbiome to try to better understand this problem. Fecal matter is 50 percent bacteria. Microorganisms must be playing an important metabolic role.
Andrew Patterson, associate professor of veterinary and biomedical sciences, has learned a great deal about how bacteria influence obesity and the metabolic diseases associated with obesity, namely type II diabetes and non-alcoholic fatty liver disease. Par exemple, in his studies, he has noticed that mice given tempol, a drug typically used to protect cells against radiation damage, weigh significantly less than mice not given the drug. To investigate further, he and his team designed an experiment in which they fed mice a high-fat diet and gave them the drug tempol. They found that these mice gained significantly less weight than mice that were fed a high-fat diet but were not given tempol.
Patterson determined that the tempol was likely reducing the amounts of Lactobacillus and Clostridium bacteria in the mice guts. And when these bacteria decreased, a specific bile acid—known as tauro-beta muricholic acid—increased. "For some reason the bacteria metabolize bile acids either as a protective mechanism, or as a way of scavenging off nutrients for growth, " il dit.

Mushroom slices are dropped into test tubes to be frozen in order to remove all moisture. Credit:Michelle Bixby
Secreted from the liver into the intestine, bile acids are responsible for digesting dietary fats and oils. "If you have a disorder in these types of processes then you have a really hard time digesting fat, " says Patterson.
To determine what was going on between the Lactobacillus and bile acids, Patterson turned to metabolomics—the study of the chemical fingerprints that are left behind after cellular processes take place. He learned that when the bile acid tauro-beta muricholic acid increases, it turns off the farnesoid X receptor (FXR), which is responsible for regulating the metabolism of bile acids, graisses, and glucose in the body. "FXR is there to say, 'Hey, there's enough bile acid in the intestine, shut off synthesis in the liver, or there's not enough bile acid so synthesis needs to be turned on, '" says Patterson.
The revelation about FXR's involvement led Patterson and his colleagues at the Hershey Medical Center and the National Cancer Institute to design their own anti-obesity drug that specifically targets FXR. In less than two years, they created a pill, modeled after tauro-beta muricholic acid but made from glycine-beta muricholic acid, that caused mice to gain significantly less weight and have less insulin resistance when fed a high-fat diet than mice in the untreated control group.
Patterson received a RAIN grant from the college to help commercialize his product. He also formed a company, called Heliome Biotech, Inc., to commercialize the drug, along with any others that may arise. But Patterson cautions that although his drug has the potential to help patients, it isn't a cure-all. "I don't think this is going to be the magic pill that allows you to eat a tub of ice cream every day and not see any metabolic problems later in life, " he says. "You have to adopt a healthy lifestyle as well."
Lactobacillusisn't the only bacteria with strains that can cause problems.E. coli, trop, which is common and generally benign throughout the human gut, includes at least one very dangerous strain—O157:H7. This bacterium enters the body through contaminated foods.
"One of the things that makes O157:H7 so terrible is when it gets in your intestines it produces a powerful toxin called Shiga toxin that is responsible for a lot of the serious symptoms of disease, from mild diarrhea to severe kidney damage, " says Edward Dudley, associate professor of food science. "The question is what's the difference between the people who only get diarrhea and those who get terribly sick?"
Dudley believes our gut microbiomes might hold the key to this question. "Could it be that different microorganisms in our gut—what I carry versus what you carry—could cause our reactions to O157:H7 to be different? After all, when O157:H7 enters our intestines, it's not existing there by itself; it's interacting with hundreds of microorganisms that are found in our gut. We are asking whether any of these organisms that O157:H7 finds itself setting up shop with do anything that may either increase or decrease the amount of toxin that the organism produces."
Those suspicions were confirmed when Chun Chen, un ancien Ph.D. student of Dudley's, grew O157:H7 in the laboratory together with a variety of strains of common gut E. coli. "When grown together with O157:H7 some of them dramatically increase the amount of toxin that O157:H7 produces, suggesting what E. coli strains are in your intestines might actually play a role in dictating the course of the disease, " says Dudley.
Edward Dudley, associate professor of food science in the College of Agricultural Sciences, and Chun Chen, un ancien Ph.D. student of Dudley's, grew O157:H7 in the laboratory together with a variety of strains of common gut E. coli to confirm suspicions over whether or not the different microorganisms within in our gut could cause different reactions to this dangerous strain of bacteria. Their study suggests what E. coli strains are present in a person's intestines might actually play a role in dictating the course of the disease. Crédit :iStock
Another one of Dudley's former students, Kakolie Goswami, along with a colleague from the University of Michigan, repeated the experiment using sterile mice that contained no bacteria. They inoculated those mice with O157:H7 and also with a nonpathogenic strain of E. coli that he'd previously shown in the laboratory to amplify Shiga toxin. They found the same result:the mice with the O157:H7 and the other strain of E. coli did worse than the mice with only O157:H7.
According to Dudley, antibiotics can't be used to treat O157:H7 the way they can with other bacteria. En réalité, antibiotics actually increase the amount of Shiga toxin that the pathogen produces. Par conséquent, doctors simply monitor patients and treat their symptoms. "By knowing which E. coli strains a patient carries, doctors could predict the course of the disease in patients and be prepared with various treatments, " says Dudley. He adds that it also may be possible to use certain strains of E. coli as a probiotic that patients could ingest that would block the ability of O157:H7 to produce large quantities of toxin.
"Depuis des décennies, we studied various bacteria growing as pure cultures in the lab, alors qu'en réalité, whether it's human health or in the soils or oceans, no bacteria, except a very tiny number of them, exist by themselves, " says Dudley. "They are always in a community of other organisms. Now we're really beginning to pick apart just how an organism of interest behaves differently when it's in the presence of other organisms. There is much to learn."
En effet, microbiome research is an open book with the potential to transform our lives. "We know now that microbiomes are driving more than we ever thought, " says Bull. "There is a wealth of information that we have only just begun to tap."
Bull notes that it's important for public institutions, like Penn State, to be involved in this type of research. "As a land-grant institution, Penn State is mandated to create knowledge for the greater good, " she says. "For example, if you talk to a farmer, he or she will tell you, 'This is my best soil; anything I grow here will be fine, ' but a hundred yards away, he or she might say, 'This is not a great spot, '" says Bull. "Farmers know the difference because they've seen the yields, but they do not know why one spot is great for their crops and the other is not. It's our job as scientists to do the research and give them the answer for that."