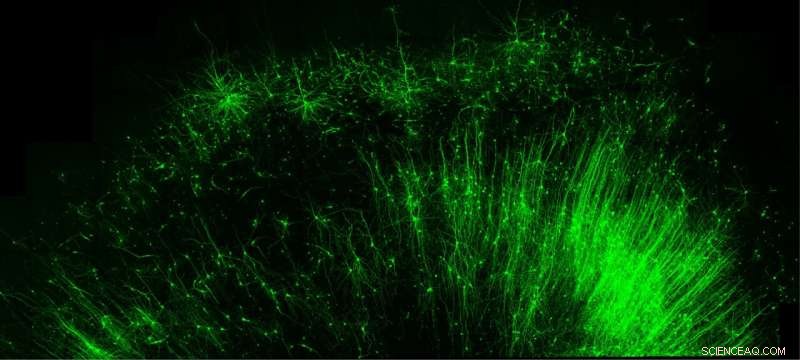
Marquer et illuminer uniquement les cellules « frein » inhibitrices (vertes) dans le tissu cérébral humain n'est qu'une des nombreuses choses que le nouvel outil de l'Université Duke, CellREADR, peut faire. Crédit :Derek Southwell, Duke University
Les chercheurs de l'Université Duke ont développé un outil d'édition basé sur l'ARN qui cible les cellules individuelles plutôt que les gènes. Il est capable de cibler précisément tout type de cellule et d'ajouter sélectivement toute protéine d'intérêt.
Les chercheurs ont déclaré que l'outil pourrait permettre de modifier des cellules et des fonctions cellulaires très spécifiques pour gérer la maladie.
À l'aide d'une sonde à base d'ARN, une équipe dirigée par le neurobiologiste Z. Josh Huang, Ph.D. et le chercheur postdoctoral Yongjun Qian, Ph.D. démontré qu'ils peuvent introduire dans les cellules des marqueurs fluorescents pour étiqueter des types spécifiques de tissus cérébraux ; un interrupteur marche/arrêt sensible à la lumière pour faire taire ou activer les neurones de leur choix ; et même une enzyme d'autodestruction pour effacer précisément certaines cellules mais pas d'autres. L'œuvre paraît le 5 octobre dans Nature .
Leur système de surveillance et de contrôle sélectif des cellules repose sur l'enzyme ADAR, présente dans les cellules de chaque animal. Bien que CellREADR (Accès cellulaire par détection d'ARN par ADAR endogène) n'en soit qu'à ses débuts, les applications possibles semblent infinies, a déclaré Huang, tout comme son potentiel de travail dans tout le règne animal.
"Nous sommes ravis car cela fournit une technologie simplifiée, évolutive et généralisable pour surveiller et manipuler tous les types de cellules chez n'importe quel animal", a déclaré Huang. "Nous pourrions en fait modifier des types spécifiques de fonction cellulaire pour gérer les maladies, quelle que soit leur prédisposition génétique initiale", a-t-il déclaré. "Ce n'est pas possible avec les thérapies ou les médicaments actuels."
CellREADR est une chaîne d'ARN personnalisable composée de trois sections principales :un capteur, un panneau d'arrêt et un ensemble de plans.
Tout d'abord, l'équipe de recherche décide du type de cellule spécifique qu'elle souhaite étudier et identifie un ARN cible qui est uniquement produit par ce type de cellule. La remarquable spécificité tissulaire de l'outil repose sur le fait que chaque type de cellule fabrique un ARN signature qui n'est pas observé dans d'autres types de cellules.
Une séquence de capteur est ensuite conçue comme brin complémentaire de l'ARN cible. Tout comme les barreaux de l'ADN sont constitués de molécules complémentaires qui sont intrinsèquement attirées les unes vers les autres, l'ARN a le même potentiel magnétique pour se lier à un autre morceau d'ARN s'il a des molécules correspondantes.
Une fois qu'un capteur s'est introduit dans une cellule et a trouvé sa séquence d'ARN cible, les deux morceaux fusionnent pour créer un morceau d'ARN double brin. Ce nouveau mashup d'ARN déclenche l'enzyme ADAR pour inspecter la nouvelle création, puis modifier un seul nucléotide de son code.
The ADAR enzyme is a cell-defense mechanism designed to edit double-strand RNA when it occurs, and is believed to be found in all animal cells.
Knowing this, Qian designed CellREADR's stop sign using the same specific nucleotide ADAR edits in double-stranded RNA. The stop sign, which prevents the protein blueprints from being built, is only removed once CellREADR's sensor docks to its target RNA sequence, making it highly specific for a given cell type.
Once ADAR removes the stop sign, the blueprints can be read by cellular machinery that builds the new protein inside the target cell.
In their paper, Huang and his team put CellREADR through its paces. "I remember two years ago when Yongjun built the first iteration of CellREADR and tested it in a mouse brain," Huang said. "To my amazement, it worked spectacularly on his first try."
The team's careful planning and design paid off as they were then able to demonstrate CellREADR accurately labelled specific brain cell populations in living mice, as well as effectively added activity monitors and control switches where directed. It also worked well in rats, and in human brain tissue collected from epilepsy surgeries.
"With CellREADR, we can pick and choose populations to study and really begin to investigate the full range of cell types present in the human brain," said co-author Derek Southwell, M.D., Ph.D., a neurosurgeon and assistant professor in the department of neurosurgery at Duke.
Southwell hopes CellREADR will improve his and others' understanding of the wiring diagram for human brain circuits and the cells within them, and in doing so, help advance new therapies for neurological disorders, such as a promising new method to treat drug-resistant epilepsy he is piloting.
Huang and Qian are especially hopeful about CellREADR's potential as a "programmable RNA medicine" to possibly cure diseases—since that's what drew them both to science in the first place. They have applied for a patent on the technology.
"When I majored in pharmacology as an undergraduate, I was very naïve," Qian said. "I thought you could do a lot of things, like cure cancer, but actually it's very difficult. However, now I think, yes maybe we can do it." New technology identifies molecular properties of cells and maps their location within tissues