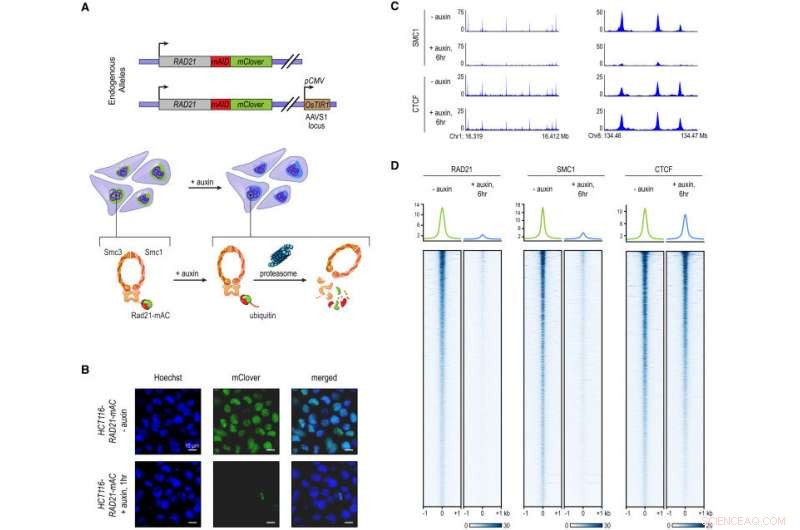
Le marquage de RAD21 endogène avec un Degron inductible aux Perte de Cohésine presque complète. (A) Dans les cellules HCT-116-RAD21-mAC, les deux allèles RAD21 sont marqués avec des degrons inductibles par l'auxine et un reporter mClover, et le gène OsTIR1 est intégré au locus AAVS1. Le traitement à l'auxine entraîne une dégradation protéasomale de RAD21. (B) Imagerie de cellules vivantes après coloration Hoechst 33342 pour marquer les noyaux. La fluorescence nucléaire de mClover correspondant à RAD21 marquée a été perdue après 1 h de traitement auxine. (C) SMC1 et CTCF ChIP-seq signal avec et sans traitement auxine. (D) RAD21, SMC1, et CTCF ChIP-seq signal (gauche, milieu, à droite) sur tous les pics appelés pour chacune des protéines dans les cellules RAD21-mAC non traitées. En haut :Enrichissements moyens pour chaque protéine. Après dégradation de RAD21, le complexe de cohésine ne se lie plus à la chromatine. La liaison CTCF n'est pas affectée. Crédit: Cellule . DOI :10.1016/j.cell.2017.09.026
Une équipe multi-institutionnelle couvrant le Baylor College of Medicine, Université du riz, L'Université de Stanford et le Broad Institute du MIT et de Harvard ont créé la première carte 4D haute résolution du repliement du génome, le suivi d'un génome humain entier au fur et à mesure qu'il se replie au fil du temps. Le rapport, qui peuvent conduire à de nouvelles façons de comprendre les maladies génétiques, apparaît sur la couverture de Cellule .
Établir des connexions
Depuis des décennies, les chercheurs ont soupçonné que lorsqu'une cellule humaine répond à un stimulus, Les éléments d'ADN éloignés les uns des autres dans le génome se retrouvent rapidement, formant des boucles le long du chromosome. En réarrangeant ces éléments d'ADN dans l'espace, la cellule est capable de changer quels gènes sont actifs.
En 2014, la même équipe de scientifiques a montré qu'il était possible de cartographier ces boucles. Mais les premières cartes étaient statiques, sans la possibilité de regarder les boucles changer. Il n'était pas clair si, dans l'espace encombré du noyau, Les éléments de l'ADN pourraient se trouver suffisamment rapidement pour contrôler les réponses cellulaires.
"Avant, nous pourrions faire des cartes de la façon dont le génome s'est replié lorsqu'il était dans un état particulier, mais le problème avec une image statique est que si rien ne change jamais, il est difficile de comprendre comment les choses fonctionnent, " a déclaré Suhas Rao, premier auteur de la nouvelle étude. "Notre approche actuelle ressemble plus à la réalisation d'un film ; nous pouvons regarder les plis disparaître et réapparaître."
Un anneau pour les gouverner tous
Pour suivre le processus de pliage au fil du temps, l'équipe de recherche a commencé par perturber la cohésine, un complexe protéique en forme d'anneau qui était situé aux limites de presque toutes les boucles connues. En 2015, l'équipe a proposé que la cohésine crée des boucles d'ADN dans le noyau cellulaire par un processus d'extrusion.
"L'extrusion fonctionne comme le réglage de la longueur de la sangle sur un sac à dos, " a expliqué le Dr Erez Lieberman Aiden, directeur du Center for Genome Architecture du Baylor College of Medicine et auteur principal de la nouvelle étude. « Lorsque vous passez la sangle de chaque côté, il forme une boucle. L'ADN semble faire la même chose, sauf que les anneaux de cohésion semblent jouer le rôle d'ajusteur."
Aiden a déclaré qu'une prédiction cruciale du modèle 2015 est que toutes les boucles devraient disparaître en l'absence de cohésion. Dans la nouvelle recherche, Aiden, Rao et ses collègues ont testé cette hypothèse.
« Nous avons constaté que lorsque nous avons perturbé la cohésion, des milliers de boucles ont disparu, " dit Rao, étudiant en médecine à l'Université de Stanford, Hertz Fellow et membre du laboratoire Aiden. "Puis, quand on remet la cohésion, toutes ces boucles sont revenues – souvent en quelques minutes. C'est précisément ce que vous prédriez à partir du modèle d'extrusion, et cela suggère que la vitesse à laquelle la cohésine se déplace le long de l'ADN est parmi les plus rapides pour n'importe quelle protéine humaine connue."
Boucles contre groupes
Mais tout ne s'est pas passé comme prévu par les chercheurs. Dans certains cas, boucles ont fait exactement le contraire de ce que les chercheurs avaient prévu.
« Alors que nous regardions des milliers de boucles à travers le génome s'affaiblir, nous avons remarqué un motif amusant, " dit Aiden, également un boursier McNair, Hertz Fellow et chercheur principal au Centre de physique biologique théorique de l'Université Rice. "Il y avait quelques boucles étranges qui devenaient en fait plus fortes. Ensuite, alors que nous remettons la cohésion, la plupart des boucles ont été complètement récupérées - mais ces boucles étranges ont encore fait le contraire - elles ont disparu !"
En scrutant l'évolution des cartes au fil du temps, l'équipe s'est rendu compte que l'extrusion n'était pas le seul mécanisme réunissant des éléments d'ADN. Un deuxième mécanisme, appelé compartimentation, n'impliquait pas de cohésion.
"Le deuxième mécanisme que nous avons observé est assez différent de l'extrusion, " a expliqué Rao. " L'extrusion a tendance à réunir deux éléments d'ADN à la fois, et seulement s'ils se trouvent sur le même chromosome. Cet autre mécanisme peut relier entre eux de grands groupes d'éléments, même s'ils reposent sur des chromosomes différents. Et cela semble être aussi rapide que l'extrusion."
Directeur de l'Institut Large Eric Lander, un co-auteur de l'étude, mentionné, "Nous commençons à comprendre les règles selon lesquelles les éléments de l'ADN se rassemblent dans le noyau. Maintenant que nous pouvons suivre les éléments à mesure qu'ils se déplacent dans le temps, les mécanismes sous-jacents commencent à devenir plus clairs."