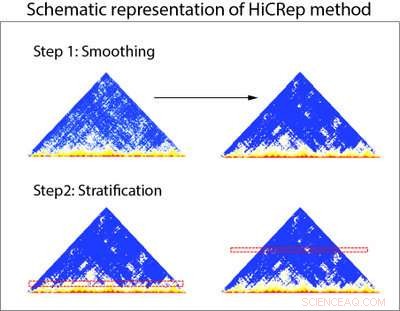
Représentation schématique de la méthode HiCRep. HiCRep utilise deux étapes pour évaluer avec précision la reproductibilité des données des expériences Hi-C. Étape 1 :Les données des expériences Hi-C (représentées dans des graphiques triangulaires) sont d'abord lissées afin de permettre aux chercheurs de voir plus clairement les tendances des données. Étape 2 :Les données sont stratifiées en fonction de la distance pour tenir compte de la surabondance d'interactions à proximité dans les données Hi-C. Crédit :Laboratoire Li, Université d'État de Pennsylvanie
Une nouvelle méthode statistique pour évaluer la reproductibilité des données de Hi-C - un outil de pointe pour étudier le fonctionnement du génome en trois dimensions à l'intérieur d'une cellule - contribuera à garantir la fiabilité des données de ces études « big data ».
"Hi-C capture les interactions physiques entre différentes régions du génome, " dit Qunhua Li, professeur adjoint de statistiques à Penn State et auteur principal de l'article. "Ces interactions jouent un rôle dans la détermination de ce qui fait d'une cellule musculaire une cellule musculaire au lieu d'une cellule nerveuse ou cancéreuse. Cependant, les mesures standard pour évaluer la reproductibilité des données ne peuvent souvent pas dire si deux échantillons proviennent du même type cellulaire ou de types cellulaires complètement indépendants. Cela rend difficile de juger si les données sont reproductibles. Nous avons développé une nouvelle méthode pour évaluer avec précision la reproductibilité des données Hi-C, ce qui permettra aux chercheurs d'interpréter avec plus de confiance la biologie à partir des données. »
La nouvelle méthode, appelé HiCRep, développé par une équipe de chercheurs de Penn State et de l'Université de Washington, est le premier à rendre compte d'une caractéristique unique des données Hi-C :les interactions entre les régions du génome qui sont proches les unes des autres sont beaucoup plus susceptibles de se produire par hasard et donc de créer de fausses, ou fausse, similitude entre des échantillons non apparentés. Un article décrivant la nouvelle méthode paraît dans la revue Recherche sur le génome .
« Avec la quantité massive de données produites dans les études sur le génome entier, il est essentiel d'assurer la qualité des données, " a déclaré Li. "Avec des technologies à haut débit comme Hi-C, nous sommes en mesure d'acquérir de nouvelles connaissances sur le fonctionnement du génome à l'intérieur d'une cellule, mais seulement si les données sont fiables et reproductibles."
À l'intérieur du noyau d'une cellule se trouve une quantité massive de matériel génétique sous forme de chromosomes, des molécules extrêmement longues constituées d'ADN et de protéines. Les chromosomes, qui contiennent des gènes et les séquences d'ADN régulatrices qui contrôlent quand et où les gènes sont utilisés, sont organisés et conditionnés dans une structure appelée chromatine. Le destin de la cellule, qu'elle devienne une cellule musculaire ou nerveuse, par exemple, dépend, au moins en partie, sur quelles parties de la structure de la chromatine sont accessibles pour l'expression des gènes, quelles parties sont fermées, et comment ces régions interagissent. HiC identifie ces interactions en verrouillant ensemble les régions d'interaction du génome, les isoler, puis les séquencer pour découvrir d'où ils viennent dans le génome.
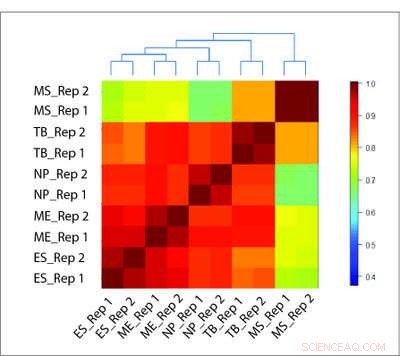
La méthode HiCRep est capable de reconstituer avec précision la relation biologique entre différents types cellulaires, là où les autres méthodes échouent. Crédit :Laboratoire Li, Université d'État de Pennsylvanie
"C'est un peu comme un bol de spaghetti géant dans lequel chaque endroit où les nouilles touchent pourrait être une interaction biologiquement importante, " dit Li. " Hi-C trouve toutes ces interactions, mais la grande majorité d'entre eux se situent entre des régions du génome très proches les unes des autres sur les chromosomes et n'ont pas de fonctions biologiques spécifiques. Une conséquence de ceci est que la force des signaux dépend fortement de la distance entre les régions d'interaction. Cela rend extrêmement difficile les mesures de reproductibilité couramment utilisées, tels que les coefficients de corrélation, pour différencier les données Hi-C car ce modèle peut sembler très similaire même entre des types de cellules très différents. Notre nouvelle méthode prend en compte cette caractéristique de Hi-C et nous permet de distinguer de manière fiable différents types de cellules. »
"Cela nous réapprend une leçon statistique de base qui est souvent négligée sur le terrain, " dit Li. " Assez souvent, la corrélation est traitée comme un indicateur de reproductibilité dans de nombreuses disciplines scientifiques, mais en fait ce n'est pas la même chose. La corrélation concerne la force avec laquelle deux objets sont liés. Deux objets non pertinents peuvent avoir une forte corrélation en étant liés à un facteur commun. C'est le cas ici. La distance est le facteur commun caché dans les données Hi-C qui détermine la corrélation, faire en sorte que la corrélation ne reflète pas les informations d'intérêt. Ironiquement, alors que ce phénomène, connu sous le nom d'effet de confusion en termes statistiques, est abordé dans chaque cours de statistique élémentaire, il est encore assez frappant de voir combien de fois il est négligé dans la pratique, même parmi les scientifiques bien formés.
Les chercheurs ont conçu HiCRep pour tenir compte systématiquement de cette caractéristique dépendante de la distance des données Hi-C. Pour y parvenir, les chercheurs lissent d'abord les données pour leur permettre de voir plus clairement les tendances dans les données. Ils ont ensuite développé une nouvelle mesure de similarité capable de distinguer plus facilement les données de différents types de cellules en stratifiant les interactions en fonction de la distance entre les deux régions. "C'est comme étudier l'effet d'un traitement médicamenteux pour une population d'âges très différents. La stratification par âge nous aide à nous concentrer sur l'effet médicamenteux. Pour notre cas, la stratification par distance nous aide à nous concentrer sur la véritable relation entre les échantillons. »
Pour tester leur méthode, l'équipe de recherche a évalué les données Hi-C de plusieurs types cellulaires différents à l'aide de HiCRep et de deux méthodes traditionnelles. Là où les méthodes traditionnelles se sont heurtées à de fausses corrélations basées sur l'excès d'interactions proches, HiCRep a pu différencier de manière fiable les types de cellules. En outre, HiCRep pourrait quantifier la quantité de différence entre les types de cellules et reconstruire avec précision quelles cellules étaient plus étroitement liées les unes aux autres.