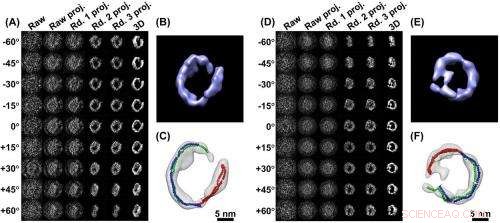
Images 3-D d'une seule particule (A) une série d'images d'une particule de protéine ApoA-1, prises sous différents angles comme indiqué. Une succession de quatre enrichissements informatiques (projections) clarifie le signal. Dans la colonne de droite se trouve l'image 3D compilée à partir des données clarifiées. B) est un gros plan de l'image 3D reconstruite. C) L'analyse montre comment la structure des particules est formée par trois protéines ApoA-1 (rouge, vert, modèles en forme de nouilles bleues)
(PhysOrg.com) -- Quand Gang Ren fait tourner les commandes de son microscope cryoélectronique, il compare cela au réglage fin du changement de vitesse et des freins d'un vélo de course. Mais cette machine du Lawrence Berkeley National Laboratory (Berkeley Lab) du département américain de l'Énergie (DOE) est un peu plus complexe. Il coûte près de 1,5 million de dollars, fonctionne à la température glaciale de l'azote liquide, et cela permet aux scientifiques de voir ce que personne n'a vu auparavant.
A la Fonderie Moléculaire, Le célèbre centre de recherche en nanotechnologie de Berkeley Lab, Ren a poussé son microscope Zeiss Libra 120 Cryo-Tem à des résolutions jamais envisagées par ses fabricants allemands, produire des instantanés détaillés de molécules individuelles. Aujourd'hui, lui et son collègue Lei Zhang rapportent les premières images 3D d'une protéine individuelle jamais obtenues avec suffisamment de clarté pour déterminer sa structure.
Les scientifiques créent régulièrement des modèles de protéines en utilisant la diffraction des rayons X, résonance magnétique nucléaire, et l'imagerie conventionnelle au microscope cryoélectronique (cryoEM). Mais ces modèles nécessitent une « moyenne » informatique des données provenant de l'analyse de milliers, voire des millions de molécules similaires, car il est si difficile de résoudre les caractéristiques d'une seule particule. Ren et Zhang ont fait exactement cela, générer des modèles détaillés à l'aide d'images au microscope électronique d'une seule protéine.
Il appelle sa technique « tomographie électronique à particules individuelles, » ou IPET. Le travail est décrit dans le numéro du 24 janvier de PLoS Un , la revue scientifique open source, dans un article intitulé « IPET and FETR :Experimental Approach for Studying Molecular Structure Dynamics by Cryo-Electron Tomography of a Single-Molecule Structure ».
Les images 3D rapportées dans l'article incluent celles d'un seul anticorps IgG et de l'apolipoprotéine A-1 (ApoA-1), une protéine impliquée dans le métabolisme humain. L'objectif de Ren est de produire des images 3D individuelles de protéines médicalement importantes, comme le HDL, le « bon cholestérol » protecteur cardiaque dont la structure a échappé aux efforts de légions de scientifiques armés d'outils de modélisation de protéines bien plus puissants. « Nous sommes bien partis, " dit Ren.
Ren a les références de quelqu'un qui sait ce qu'il peut faire. Il a été recruté pour travailler au Berkeley Lab en août 2010 de l'Université de Californie à San Francisco, où il avait utilisé un microscope cryoélectronique et des techniques de calcul de moyenne plus conventionnelles pour discerner la structure 3-D du LDL – le «mauvais cholestérol» considéré comme un facteur de risque majeur de maladie cardiaque.
Ses images de protéines uniques sont un peu floues, même après avoir été nettoyés par un filtrage informatique complexe, mais très instructif pour l'observateur entraîné. Ces particules individuelles sont extraordinairement minuscules, obligeant Ren à se concentrer sur un point de moins de 20 nanomètres. Il a rapporté des images de protéines aussi petites que 70 kDa. C'est des kilodaltons, une échelle lilliputienne (exprimée en unités de masse) réservée à la mesure des atomes, molécules, et des fragments d'ADN. C'est un moyen plus utile de dimensionner des objets mous comme des protéines qui peuvent être agglutinées, filandreux, ou disquette.
Contrairement aux images sculpturales des modèles de protéines, une suite de ces photographies peut donner une idée de ces particules dans toute leur souplesse à l'échelle nanométrique. Dans la structure complexe de ces protéines se cachent les secrets de leur fonction, et peut-être des clés de médicaments qui bloquent les mauvais et favorisent les bons. Avec un filtrage informatique supplémentaire, un modèle de protéine à contraste élevé peut être généré à partir des images et animé pour montrer ses parties mobiles en 3D.
« Cela vous permet de voir la personnalité de chaque protéine, '' dit Ren. "C'est une preuve de concept pour quelque chose que les gens pensaient impossible."
En observant la structure de protéines individuelles, il est possible de comprendre leur flexibilité, pièces mobiles. "Cela ouvre une porte pour l'étude de la dynamique des protéines, " dit Ren. « Les anticorps, par exemple, ne sont pas solides. Ils sont très souples, très dynamique.
Comment Ren a-t-il réussi à tirer autant de polyvalence de sa Balance 120 ? "Ce n'est pas un modèle très haut de gamme, '' concède-t-il. Cela a beaucoup à voir avec les accessoires qu'il boulonne sur la machine, et avec son talent artistique et sa patience. Il a équipé le microscope d'un 300 $, 000 caméra CCD, un puissant logiciel de traitement d'images, agents de contraste spéciaux, et un dispositif appelé « filtre d'énergie » qui passe au crible les données numérisées de la caméra et élimine les signaux faibles. Connaissant parfaitement sa machine sur mesure, il emploie aussi un élément d'huile de coude, travailler de longues heures pour extraire les images puissantes d'un torrent de bruit numérique.

Le scientifique du personnel Gang Ren (debout) et son collègue postdoctoral Lei Zhang peuvent vérifier des images de protéines individuelles à partir de leur microscope cryoélectronique à la fonderie moléculaire de Berkeley Lab.
Les angles multiples utilisés pour créer le portrait 3D aident à résoudre l'image moléculaire faible. « Toutes les images sont bruitées, ", explique Ren. « En physique, le bruit est incohérent entre les images, mais le signal — l'objet ou la protéine — est cohérent. En utilisant cette approche, nous trouvons que la partie cohérente (le signal) peut être améliorée, tandis que la partie incohérente (le bruit) sera considérablement réduite.
Les microscopes électroniques concentrent des flux d'électrons plutôt que de la lumière pour voir des choses incroyablement minuscules. La courte longueur d'onde d'un faisceau d'électrons permet une résolution et un grossissement beaucoup plus élevés que la lumière visible. De puissants microscopes électroniques sont utilisés depuis des décennies pour sonder les matériaux à l'échelle atomique; et juste à côté de la fonderie moléculaire se trouve le Centre national de microscopie électronique de Berkeley Lab, qui abrite les microscopes les plus puissants au monde. Le microscope TEAM 0.5 peut distinguer des objets aussi petits que le rayon d'un atome d'hydrogène. Mais ces microscopes lourds obtiennent cette résolution à l'échelle atomique avec des impulsions d'énergie qui effaceraient la plupart des protéines biologiques molles. Les microscopes électroniques à haute puissance sont principalement utilisés pour sonder la structure atomique de fortes, matériaux solides, comme le graphène - un réseau de carbone d'un seul atome d'épaisseur.
Le laboratoire de Ren est spécialisé dans la cryoEM, qui examine des objets congelés à -180 °C (-292 °F). Un bain d'azote liquide congèle les échantillons si rapidement qu'aucun cristal de glace ne se forme. « C'est amorphe, comme le verre, '' dit Ren. Les échantillons de protéines sont congelés sur un disque de la taille d'un ongle de bébé, rempli de minuscules puits de 2 microns de diamètre. Le disque est inséré dans le microscope sur un support rotatif qui peut incliner l'échantillon jusqu'à 140 ° à l'intérieur d'un vide - des angles de caméra suffisants pour produire une perspective 3D. « Le défi est de l'isoler de l'air, et de le tourner sans vibrations, même les vibrations du bouillonnement de l'azote liquide, '' dit Ren.
La température extrêmement basse fixe les échantillons et les empêche de se dessécher dans le vide nécessaire au balayage électronique. Il crée des conditions favorables à l'imagerie à des doses d'électrons beaucoup plus faibles - suffisamment faibles pour garder une seule protéine molle intacte tandis que plus de 100 images sont prises sur une période d'une à deux heures.