
Deux colonies bactériennes qui ont formé des anneaux violets à cause du forçage génétique que les chercheurs y ont implanté. Un nouveau modèle d'apprentissage automatique accélère considérablement cette science en prédisant l'interaction de dizaines de variables biologiques. Crédit :Lingchong You, université de Duke
Les ingénieurs biomédicaux de l'Université Duke ont conçu une approche d'apprentissage automatique pour modéliser les interactions entre des variables complexes dans des bactéries modifiées qui seraient autrement trop lourdes à prévoir. Leurs algorithmes sont généralisables à de nombreux types de systèmes biologiques.
Dans la nouvelle étude, les chercheurs ont formé un réseau neuronal pour prédire les motifs circulaires qui seraient créés par un circuit biologique intégré dans une culture bactérienne. Le système a fonctionné 30, 000 fois plus rapide que le modèle de calcul existant.
Pour améliorer encore la précision, l'équipe a conçu une méthode pour recycler le modèle d'apprentissage automatique plusieurs fois afin de comparer leurs réponses. Ensuite, ils l'ont utilisé pour résoudre un deuxième système biologique exigeant en calcul d'une manière différente, montrant que l'algorithme peut fonctionner pour des défis disparates.
Les résultats paraissent en ligne le 25 septembre dans la revue Communication Nature .
"Ce travail a été inspiré par Google montrant que les réseaux de neurones pouvaient apprendre à battre un humain dans le jeu de société Go, " dit Lingchong You, professeur de génie biomédical à Duke.
"Même si le jeu a des règles simples, il y a beaucoup trop de possibilités pour qu'un ordinateur calcule la meilleure option suivante de manière déterministe, " Vous avez dit. " Je me suis demandé si une telle approche pouvait être utile pour faire face à certains aspects de la complexité biologique auxquels nous sommes confrontés. "
Le défi auquel vous et son associé postdoctoral Shangying Wang étaient confrontés était de déterminer quel ensemble de paramètres pourrait produire un modèle spécifique dans une culture bactérienne suivant un circuit génétique modifié.

Une colonie bactérienne génétiquement modifiée pour inclure un circuit génique forme un anneau violet au fur et à mesure de sa croissance. Les chercheurs utilisent l'apprentissage automatique pour découvrir les interactions entre des dizaines de variables qui affectent les propriétés de l'anneau telles que son épaisseur, à quelle vitesse il se forme et le nombre d'anneaux qui se forment. Crédit :Lingchong You, université de Duke
Dans des travaux antérieurs, Votre laboratoire a programmé des bactéries pour produire des protéines qui, selon les spécificités de la croissance de la culture, interagissent les uns avec les autres pour former des anneaux. En contrôlant des variables telles que la taille de l'environnement de croissance et la quantité de nutriments fournis, les chercheurs ont découvert qu'ils pouvaient contrôler l'épaisseur de l'anneau, combien de temps il a fallu pour apparaître et d'autres caractéristiques.
En changeant un nombre quelconque de dizaines de variables potentielles, les chercheurs ont découvert qu'ils pouvaient faire plus, comme provoquer la formation de deux ou même trois anneaux. Mais comme une seule simulation informatique a pris cinq minutes, il est devenu impossible de rechercher un grand espace de conception pour un résultat spécifique.
Pour leur étude, le système se composait de 13 variables bactériennes telles que les taux de croissance, la diffusion, dégradation des protéines et mouvement cellulaire. Le simple calcul de six valeurs par paramètre prendrait à un seul ordinateur plus de 600 ans. L'exécuter sur un cluster d'ordinateurs parallèle avec des centaines de nœuds peut réduire ce temps d'exécution à plusieurs mois, mais l'apprentissage automatique peut le réduire à des heures.
"Le modèle que nous utilisons est lent car il doit prendre en compte des étapes intermédiaires dans le temps à un rythme suffisamment faible pour être précis, " a dit You. " Mais nous ne nous soucions pas toujours des étapes intermédiaires. Nous voulons juste les résultats finaux pour certaines applications. Et nous pouvons (revenir en arrière) déterminer les étapes intermédiaires si nous trouvons les résultats finaux intéressants."
Pour passer aux résultats finaux, Wang s'est tourné vers un modèle d'apprentissage automatique appelé réseau de neurones profonds qui peut effectivement faire des prédictions de plusieurs ordres de grandeur plus rapidement que le modèle d'origine. Le réseau prend en entrée des variables de modèle, attribue initialement des poids et des biais aléatoires, et crache une prédiction du modèle que la colonie bactérienne formera, en sautant complètement les étapes intermédiaires menant au motif final.
Bien que le résultat initial ne soit pas du tout proche de la bonne réponse, les poids et les biais peuvent être modifiés à chaque fois que de nouvelles données d'entraînement sont introduites dans le réseau. Compte tenu d'un ensemble « d'entraînement » suffisamment grand, le réseau de neurones finira par apprendre à faire des prédictions précises presque à chaque fois.
Pour gérer les quelques cas où l'apprentissage automatique se trompe, Vous et Wang avez trouvé un moyen de vérifier rapidement leur travail. Pour chaque réseau de neurones, le processus d'apprentissage comporte un élément d'aléatoire. En d'autres termes, il n'apprendra jamais deux fois de la même manière, même s'il est entraîné sur le même ensemble de réponses.
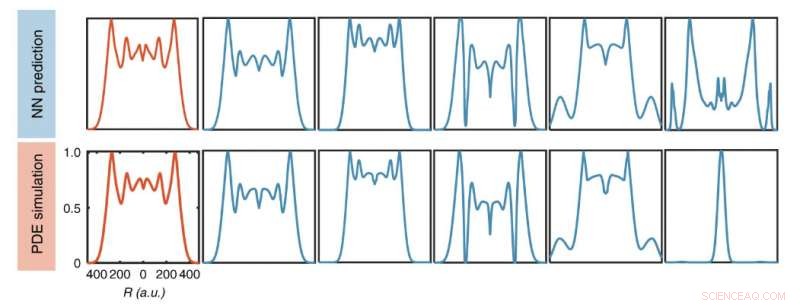
Chacun de ces graphiques représente une section transversale d'une colonie bactérienne. Les pics prédisent où la colonie produira des protéines violettes qui forment des anneaux en raison d'un circuit génétique artificiel. Les graphiques du haut ont été créés par un algorithme d'apprentissage automatique, tandis que ceux du bas ont été créés par une simulation plus approfondie. Ils correspondent très bien - sauf pour le dernier. Crédit :Université Duke
Les chercheurs ont formé quatre réseaux de neurones distincts et comparé leurs réponses pour chaque instance. Ils ont découvert que lorsque les réseaux de neurones entraînés font des prédictions similaires, ces prédictions étaient proches de la bonne réponse.
"Nous avons découvert que nous n'avions pas à valider chaque réponse avec le modèle de calcul standard plus lent, " a dit You. " Nous avons essentiellement utilisé la 'sagesse de la foule' à la place. "
Avec le modèle d'apprentissage automatique formé et corroboré, les chercheurs ont décidé de l'utiliser pour faire de nouvelles découvertes sur leur circuit biologique. Dans les 100 premiers, 000 simulations de données utilisées pour entraîner le réseau de neurones, un seul a produit une colonie bactérienne avec trois anneaux. Mais avec la vitesse du réseau de neurones, Vous et Wang avez non seulement pu trouver beaucoup plus de triplés, mais déterminer quelles variables étaient cruciales pour les produire.
"Le réseau neuronal a été capable de trouver des modèles et des interactions entre les variables qui auraient été autrement impossibles à découvrir, " dit Wang.
En guise de conclusion à leur étude, Vous et Wang avez essayé leur approche sur un système biologique qui fonctionne de manière aléatoire. La résolution de tels systèmes nécessite qu'un modèle informatique répète les mêmes paramètres plusieurs fois pour trouver le résultat le plus probable. Bien que ce soit une raison complètement différente pour les longs temps d'exécution de calcul que leur modèle initial, les chercheurs ont trouvé que leur approche fonctionnait toujours, montrant qu'il est généralisable à de nombreux systèmes biologiques complexes différents.
Les chercheurs essaient maintenant d'utiliser leur nouvelle approche sur des systèmes biologiques plus complexes. En plus de l'exécuter sur des ordinateurs dotés de GPU plus rapides, ils essaient de programmer l'algorithme pour qu'il soit aussi efficace que possible.
"Nous avons entraîné le réseau de neurones avec 100, 000 jeux de données, mais c'était peut-être exagéré, ", a déclaré Wang. "Nous développons un algorithme où le réseau de neurones peut interagir avec des simulations en temps réel pour aider à accélérer les choses."
"Notre premier objectif était un système relativement simple, " a déclaré You. " Maintenant, nous voulons améliorer ces systèmes de réseaux neuronaux pour fournir une fenêtre sur la dynamique sous-jacente de circuits biologiques plus complexes. "