
Architecture de télescope :le gestionnaire de travaux rassemble les informations sur les travaux en se connectant au cluster cible via son gestionnaire de connexion. La base de données locale du télescope conserve des enregistrements de ces informations, qui est rendu par l'interface utilisateur dans une page Web (conviviale pour les mobiles). Crédit :Brito et al.
Une équipe de chercheurs de l'UCLA, l'Université de São Paulo, l'Université fédérale de São Carlos et l'Université de Californie du Sud ont récemment développé un outil interactif pour gérer les analyses bioinformatiques à grande échelle en temps réel et à partir d'appareils portables. Ce nouvel outil, appelé Télescope, a été présenté pour la première fois dans un article prépublié le arXiv .
L'équipe qui a développé Telescope comprend des boursiers postdoctoraux de l'UCLA Collaboratory, un laboratoire de recherche à l'Institute for Quantitative and Computational Biosciences de l'université. Travailler à UCLA Collaboratory permet à ces chercheurs d'interagir avec d'autres étudiants et membres du corps professoral de l'université qui ont différents niveaux d'expertise en biologie computationnelle. Ces interactions sont ce qui a finalement inspiré l'équipe à développer un outil convivial pour gérer les analyses bioinformatiques à grande échelle.
« La plupart des études en sciences de la vie et biomédicales ciblent aujourd'hui des effets si subtils et si difficiles à détecter que de grandes quantités de données et de puissance de calcul sont devenues le nouveau statu quo, " les chercheurs ont dit TechXplore par e-mail. " Souvent, cependant, les chercheurs décrivent la dépendance excessive vis-à-vis des outils de ligne de commande comme étant encombrante et très peu intuitive pour ceux qui manquent de formation informatique formelle. »
Les initiatives de formation spécialisée et les ateliers se sont avérés particulièrement efficaces pour encourager les chercheurs à commencer à utiliser des méthodes puissantes pour les analyses bioinformatiques à grande échelle. Cependant, la plupart des techniques pour accéder à ces méthodes sur des installations informatiques ne sont pas très bien intégrées aux environnements de laboratoire humide, où la plupart des expériences de biologie sont effectuées. Avec ça en tête, les chercheurs ont entrepris de développer un outil qui permettrait une meilleure intégration entre les outils bioinformatiques et la recherche en laboratoire humide.
"Telescope a été proposé pour favoriser l'intégration et une collaboration plus étroite entre la recherche en laboratoire humide et la bioinformatique, réduire la courbe d'apprentissage traditionnellement requise pour exécuter des analyses bioinformatiques complexes dans des installations de calcul, " a déclaré l'équipe à TechXplore. " Le défi, cependant, garantissait que la solution finale comblerait effectivement les lacunes et serait intuitive pour la plupart des chercheurs. »
Afin de présenter leur solution à la fois aux chercheurs férus de technologie et à ceux ayant des compétences informatiques limitées, les chercheurs ont décidé de présenter Telescope lors de l'événement II Hackathon début 2018. Le Hackathon rassemble des chercheurs ayant des compétences en informatique et en codage, en leur offrant l'opportunité d'acquérir des compétences informatiques en groupe, par exemple, explorer comment créer des scripts et appliquer des outils bioinformatiques aux ensembles de données.
« Notre idée a attiré à la fois des débutants qui voulaient se salir les mains en travaillant directement avec des données le plus rapidement possible et des experts en technologie qui voulaient simplement un moyen plus simple de vérifier leur travail depuis leur téléphone. », expliquent les chercheurs. « Telescope a donc été conçu par et pour la communauté.
La plupart des chercheurs qui effectuent des expériences dans des environnements de laboratoire humide doivent répondre à des exigences de temps considérables. Telescope est conçu pour les aider à mieux gérer leur charge de travail, superviser et ajuster les analyses bioinformatiques en temps réel.
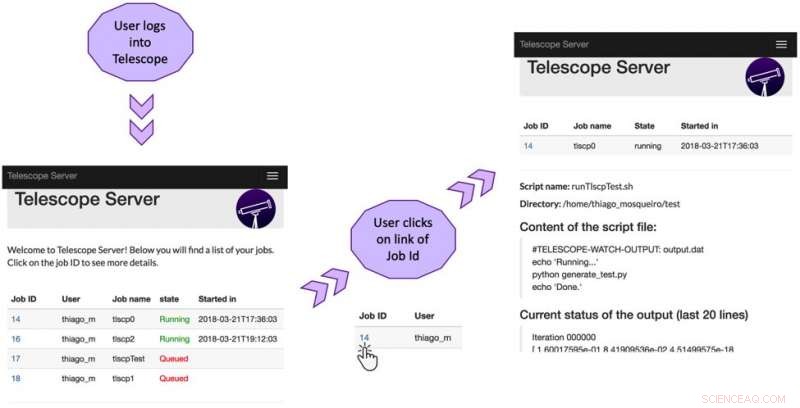
Interface utilisateur du télescope :le premier écran affiche l'état des travaux sur le cluster. L'écran suivant affiche des informations détaillées sur le premier travail répertorié :répertoire source, nom et contenu du fichier de script, et les dernières lignes de la sortie de la tâche en cours. Crédit :Brito et al.
Essentiellement, Telescope renforce la relation entre les expériences en laboratoire humide et les outils d'analyse de grandes quantités de données biologiques. Pour ce faire, il fournit une plate-forme sécurisée et conviviale sur laquelle les utilisateurs peuvent lancer leurs recherches et gérer les tâches pertinentes pour les travaux de bioinformatique exécutés dans des clusters hautes performances.
"Telescope s'appuie sur des frameworks Web courants tels que Bootstrap de Twitter pour offrir une expérience utilisateur agréable et prendre en charge une interface extensible, ", ont déclaré les chercheurs. " Dans l'intention de soutenir les utilisateurs nouveaux et expérimentés, Le cœur de Telescope gère et stocke les clés d'accès des utilisateurs en utilisant les normes de l'industrie."
Les clés d'accès stockées par Telescope sont utilisées pour connecter des clusters de calcul hautes performances via un shell sécurisé afin de recevoir les dernières mises à jour de l'état des travaux et d'émettre de nouvelles commandes chaque fois que nécessaire (par exemple, suppression d'anciennes tâches, etc.). Telescope permet aux scientifiques des laboratoires humides de suivre l'état d'avancement de leurs travaux et d'accéder facilement aux résultats préliminaires de leurs analyses bioinformatiques directement sur leurs téléphones portables. Cela signifie qu'ils peuvent également identifier les problèmes potentiels avec leurs analyses dès le début et les annuler sans avoir à utiliser un ordinateur.
"Par rapport à l'approche habituelle consistant à utiliser des lignes de commande sur les terminaux Linux, Le télescope est un outil plus intuitif et interactif, " les chercheurs ont expliqué. " Par exemple, le partage des résultats d'analyse est aussi simple que le partage d'un lien, comme vous le feriez sur Twitter ou Facebook. Néanmoins, nous pensons que notre réalisation la plus significative a été l'engagement avec la communauté traditionnellement non informatique des sciences de la vie au début du développement."
Ce nouvel outil de gestion des analyses bioinformatiques a pris en compte les besoins des chercheurs en biologie. En réalité, l'équipe a profité de l'événement Hackathon pour recueillir les commentaires et les commentaires d'experts en biologie ayant divers degrés de compétence et d'expérience en informatique en utilisant des outils d'analyse informatique.
« Nous avons recueilli les commentaires d'utilisateurs novices en informatique, comme nous voulions créer un outil qui aborde les points les plus difficiles de l'utilisation de lignes de commande peu intuitives, ", ont déclaré les chercheurs. "Nous avons ensuite développé une interface Web simple et minimale qui peut fonctionner sur divers appareils, y compris les téléphones portables, avec zéro configuration. Le défi était de concilier simplicité et sécurité, et nous avons atteint un bon équilibre en utilisant des protocoles standard de l'industrie, comme le stockage de clés à l'aide de PDBKS."
L'outil développé par ce groupe de chercheurs, qui est maintenant disponible sur GitHub, peut assister les experts en biologie dans leur travail, en les aidant à gérer leurs analyses bioinformatiques de manière plus simple et plus intuitive. En plus de permettre aux chercheurs biomédicaux d'accéder à la puissance de grandes installations informatiques à partir de leurs appareils portables, Telescope est un logiciel personnalisable et extensible. Cela signifie que les utilisateurs peuvent y ajouter de nouvelles fonctionnalités ou aider à le développer davantage.
"Notre projet sera désormais maintenu par le Mangul Lab de l'USC en tant que logiciel open source, ", ont déclaré les chercheurs. "Nous prévoyons également d'intégrer Telescope dans la plupart des pipelines d'analyse de notre laboratoire afin d'évaluer davantage l'expérience utilisateur. Comme prochaine étape, ces tests seront étendus à un sondage de bêta-testeurs d'une communauté plus large. Notre objectif primordial est de déployer Telescope pour les utilisateurs du cluster haute performance de l'USC à l'avenir. Tout au long de ce processus, le laboratoire Mangul s'efforcera de s'engager avec la communauté pour favoriser l'adoption et garantir que Telescope reste pertinent. »
© 2019 Réseau Science X