
Un modèle de réponse immunitaire innée à base d'agents simule mécaniquement la septicémie en 2D. Crédit :Laboratoire national Lawrence Livermore
Une approche d'apprentissage en profondeur conçue à l'origine pour enseigner aux ordinateurs comment jouer aux jeux vidéo mieux que les humains pourrait aider à développer un traitement médical personnalisé pour la septicémie, une maladie qui cause environ 300, 000 décès par an et pour lesquels il n'existe aucun remède connu.
Laboratoire national Lawrence Livermore (LLNL), en collaboration avec des chercheurs de l'Université du Vermont, explore comment l'apprentissage par renforcement en profondeur peut découvrir des stratégies médicamenteuses thérapeutiques pour la septicémie en utilisant une simulation du système immunitaire inné d'un patient comme plate-forme pour des expériences virtuelles. L'apprentissage par renforcement profond est une approche d'apprentissage automatique de pointe développée à l'origine par Google DeepMind pour enseigner à un réseau de neurones comment jouer à des jeux vidéo, donné uniquement des pixels en entrée et le score du jeu en tant que signal d'apprentissage. Les algorithmes dépassent souvent les performances humaines, bien qu'il n'ait aucune connaissance de la mécanique du jeu.
L'approche d'apprentissage en profondeur de LLNL traite la simulation du système immunitaire développée par leurs collaborateurs comme un jeu vidéo. En utilisant les sorties de la simulation, un « score » basé sur la santé du patient et un algorithme d'optimisation, le réseau neuronal apprend à manipuler 12 médiateurs cytokines différents (régulateurs du système immunitaire) pour ramener la réponse immunitaire à l'infection à des niveaux normaux. La recherche apparaît dans un article publié par la Conférence internationale sur l'apprentissage automatique.
"C'est un système complexe, " a déclaré Dan Faissol, chercheur au LLNL, chercheur principal du projet. "Les enquêtes précédentes ont jusqu'à présent été basées sur la manipulation d'un seul médiateur/cytokine, généralement administré avec une dose unique ou sur un cycle très court. Nous pensons que notre approche a un grand potentiel car elle explore beaucoup plus complexe, stratégies thérapeutiques prêtes à l'emploi qui traitent chaque patient différemment en fonction des mesures du patient au fil du temps."
La stratégie thérapeutique proposée par les chercheurs est adaptative et personnalisée, s'améliorant sur une boucle de rétroaction en observant en permanence les niveaux de cytokines et en prescrivant des médicaments spécifiques à chaque patient. Chaque exécution de la simulation représente un type de patient différent et des conditions initiales d'infection différentes.
"Le défi était de garder les choses cliniquement pertinentes, " a expliqué le chercheur du LLNL Brenden Petersen, le responsable technique du projet. "Nous devions nous assurer que tous les aspects du problème simulé étaient pertinents dans le monde réel - que l'ordinateur n'utilisait aucune information qui ne serait pas vraiment disponible dans un hôpital. Alors, nous n'avons fourni au réseau de neurones que des informations qui peuvent effectivement être mesurées cliniquement, comme les niveaux de cytokines et le nombre de cellules d'une prise de sang."
En utilisant le modèle basé sur les agents avec un apprentissage par renforcement approfondi, les chercheurs ont identifié une politique de traitement qui permet d'atteindre un taux de survie de 100 pour cent pour les patients sur lesquels elle a été formée, et une mortalité inférieure à 1 pour cent sur 500 patients choisis au hasard.
"La simulation est de nature mécaniste, ce qui signifie que nous pouvons expérimenter virtuellement des médicaments et des combinaisons de médicaments qui n'ont pas été testés auparavant pour voir s'ils pourraient être prometteurs, " a déclaré Faissol. " Le nombre de stratégies de traitement possibles est énorme, surtout lorsqu'on envisage des stratégies multidrogues qui varient dans le temps. Sans utiliser de simulation, il n'y a aucun moyen de les évaluer tous. Le plus difficile est de découvrir une stratégie qui fonctionne pour tous les types de patients. L'infection de chacun est différente, et le corps de chacun est différent."
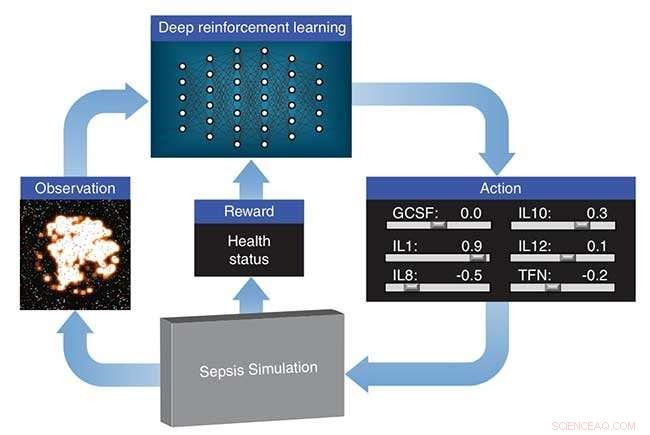
L'approche d'apprentissage en profondeur de LLNL traite la simulation du système immunitaire développée par leurs collaborateurs comme un jeu vidéo. En utilisant les sorties de la simulation, un « score » basé sur la santé du patient et un algorithme d'optimisation, le réseau neuronal apprend à manipuler 12 médiateurs cytokines différents - des régulateurs du système immunitaire - pour ramener la réponse immunitaire à l'infection à des niveaux normaux. Crédit :Laboratoire national Lawrence Livermore
Les recherches de l'équipe ont montré que cette approche adaptative peut conduire à de nouvelles perspectives, et les chercheurs espèrent convaincre d'autres d'adopter l'approche sur la septicémie et d'autres maladies.
"Notre grand, la vision à long terme est un système de chevet en « boucle fermée » où les mesures d'un patient sont intégrées à un outil d'aide à la décision, qui administre ensuite les bons médicaments aux bonnes doses aux bons moments, " Petersen a déclaré. " De telles stratégies de traitement devraient d'abord être vérifiées et affinées dans des modèles de laboratoire humide et d'animaux, éventuellement informer de vrais traitements."
Petersen a déclaré que la plupart du matériel pour exécuter un tel système en boucle fermée existe déjà, comme avec des systèmes plus simples comme les pompes à insuline qui surveillent en permanence le sang et administrent l'insuline au bon moment.
L'approche d'apprentissage par renforcement profond du Lab n'a pas encore été testée dans le monde réel, mais basé sur le succès en utilisant la simulation, les National Institutes of Health ont accordé aux chercheurs du LLNL et de l'Université du Vermont une subvention de cinq ans pour poursuivre les travaux, principalement sur le sepsis mais aussi sur le cancer.
"C'est un projet passionnant, " dit Gary An, un médecin de soins intensifs à l'Université du Vermont et un informaticien qui a développé la version originale de la simulation de la septicémie. « Il s'agit d'un projet incroyablement novateur qui rassemble trois domaines de pointe de la recherche informatique :des simulations multi-échelles à haute résolution de processus biologiques, l'extension de l'apprentissage par renforcement en profondeur à la recherche biomédicale et l'utilisation du calcul haute performance pour tout rassembler. »
Le directeur de la bio-ingénierie de LLNL, Shankar Sundaram, a décrit l'approche comme "un exemple illustratif du laboratoire contribuant au développement d'une solution thérapeutique potentielle à un problème de santé complexe essentiel à notre mission de biosécurité, appliquer et faire progresser nos capacités de pointe en apprentissage automatique scientifique et cibler une causalité améliorée, compréhension mécaniste."
Les chercheurs du LLNL ont également initié une collaboration avec le Moffitt Cancer Center en Floride pour voir si une approche similaire pourrait apprendre des stratégies de thérapie médicamenteuse efficaces en utilisant une simulation de cancer. Moffitt a publié une version de jeu vidéo de leur simulation appelée "Cancer Crusade" qui fonctionne sur les téléphones mobiles.
"Une stratégie consiste à externaliser l'apprentissage en analysant les traitements enregistrés par les meilleurs joueurs du monde, " a déclaré Petersen. " Nous avons appliqué notre approche d'apprentissage en profondeur et nous voulons voir comment nos traitements informatiques se comparent aux meilleurs joueurs - une confrontation " homme contre machine ". "
Le projet sepsis a également conduit à un nouvel effort au LLNL pour rechercher des stratégies de cyberdéfense adaptatives et autonomes utilisant la simulation et l'apprentissage par renforcement en profondeur.