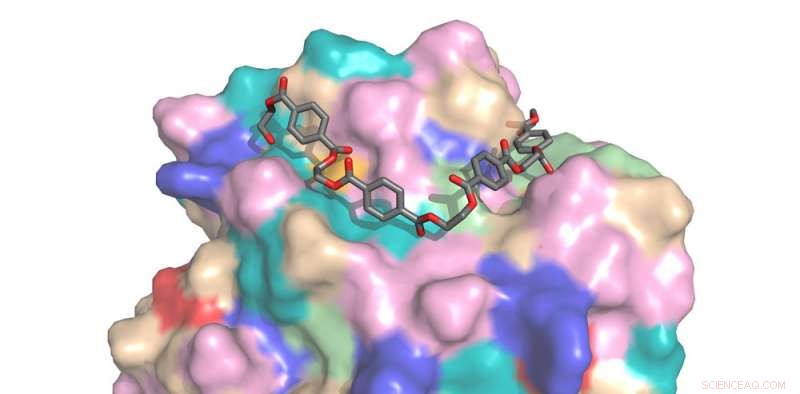
Les superordinateurs ont aidé à étudier la liaison d'une enzyme dégradant le plastique, PETase, ce qui pourrait conduire à développer le recyclage du plastique à l'échelle industrielle pour les bouteilles jetables et les tapis. Distribution du potentiel électrostatique de la structure de la PETase illustrée. Crédit :Lee Woodcock
La valeur d'un camion à benne basculante de plastique se déverse dans l'océan chaque minute. À l'échelle mondiale, l'humanité produit plus de 300 millions de tonnes de plastique chaque année, dont une grande partie devrait durer des siècles à des millénaires et pollue les environnements aquatiques et terrestres. Plastique PET, abréviation de polyéthylène téréphtalate, est le quatrième plastique le plus produit et est utilisé pour fabriquer des choses comme des bouteilles de boisson et des tapis, ces derniers n'étant pratiquement pas recyclés. Certains scientifiques espèrent changer cela, utilisant des superordinateurs pour concevoir une enzyme qui décompose le PET. Ils disent que c'est une étape sur un long chemin vers le recyclage du PET et d'autres plastiques en matériaux de valeur commerciale à l'échelle industrielle.
"Nous allons idéalement d'un endroit où les plastiques sont difficiles à recycler à un endroit où nous utilisons la nature et des millions d'années d'évolution pour diriger les choses de manière à rendre le plastique facile à recycler, " dit Lee Woodcock, professeur agrégé de chimie à l'Université de Floride du Sud. Woodcock a co-écrit une étude qui a étudié la structure d'une enzyme pour dégrader le PET et a été publiée en mars 2018 dans le Actes de l'Académie nationale des sciences .
L'étude s'appuie sur une découverte en 2016 par Yoshida et al. d'une bactérie, Ideonella sakaiensis 201-F6, qui se nourrit de plastique PET comme source de carbone et d'énergie. Les PNAS les auteurs de l'étude se sont concentrés sur l'enzyme de dégradation du plastique de la bactérie, appelé PETase. Les membres de l'équipe de l'Université de Portsmouth, dirigé par le professeur John McGeehan, utilisé la cristallographie aux rayons X à la Diamond Light Source au Royaume-Uni pour résoudre la structure cristalline à haute résolution de la PETase.
"Nous avons ensuite utilisé des simulations informatiques pour comprendre comment un ligand polymère comme le PET serait capable de se lier à l'enzyme, " a déclaré le co-auteur de l'étude Gregg Beckham, chercheur principal et chef de groupe au National Renewable Energy Laboratory (NREL) des États-Unis. « Nous avons également mené des travaux expérimentaux pour montrer qu'en effet, la PETase peut décomposer les bouteilles d'eau ou de soda, films PET industriellement pertinents, et un autre plastique, furanoate de polyéthylène."
Après avoir fait ce travail sur la structure et la fonction de l'enzyme PETase, les auteurs ont ensuite essayé de comprendre son évolution et de se tourner vers une enzyme similaire, une famille de cutinases, qui dégradent la cutine polymère cireuse présente à la surface des plantes.
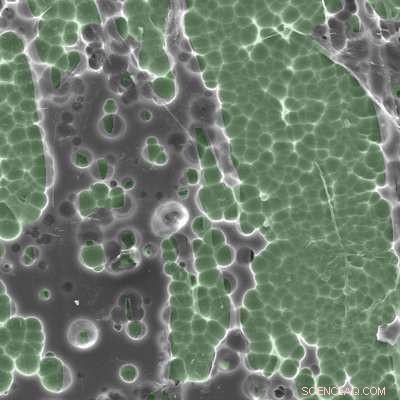
Images au microscope électronique montrant l'interaction de l'enzyme PETase avec le plastique PET. Crédit :Gregg Beckham
"Nous avons développé l'hypothèse que si nous rendons l'enzyme PETase plus semblable à une cutinase, alors nous devrions aggraver l'enzyme. Quand nous avons fait ce travail, en fait, nous avons fini par améliorer légèrement l'enzyme en faisant cela, ", a déclaré Woodcock.
"C'était incroyablement surprenant pour nous, " a expliqué Beckham. " Quand nous l'avons fait plus cutinase, l'enzyme a été légèrement améliorée. C'est en fait l'un des aspects clés de l'intervention du calcul, car il nous a permis de prédire ou de suggérer essentiellement des interactions aromatiques-aromatiques dans l'enzyme avec le polyester aromatique PET pourraient potentiellement être responsables de son activité améliorée. Mais ce fut une surprise pour nous, " a déclaré Beckham.
Les superordinateurs leur ont permis de s'attaquer à des questions scientifiques difficiles sur la PETase, tels que les détails de la façon dont il interagit à l'échelle moléculaire lié à un substrat, quelque chose au-delà de la portée de ce qui pourrait être déterminé en connaissant sa structure cristalline.
Les chercheurs ont profité pour cette étude des ressources de calcul de XSEDE, l'environnement de découverte des sciences et de l'ingénierie extrêmes, financé par la National Science Foundation.
« Avoir accès aux ressources XSEDE ouvre vraiment la possibilité de pouvoir modéliser et pouvoir étudier quel type de conformation à grande échelle voire locale, de petits changements structurels se produisent en fonction à la fois de la liaison au substrat et, en outre, quels sont les changements structurels à grande échelle ou locaux, changements structurels à petite échelle qui se produisent dans l'enzyme après que nous ayons fait les mutations. C'était une grande partie de ce que nous examinions, ", a déclaré Woodcock.
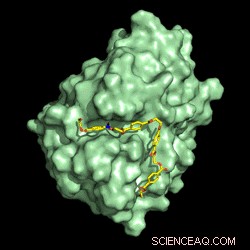
Distribution de potentiel électrostatique de la structure de la PETase. Crédit :Gregg Beckham
Woodcock a expliqué qu'ils ont simulé les longues échelles de temps de l'enzyme en utilisant le champ de force et le programme de chimie à Harvard Macromolecular Mechanics (CHARMM) lui-même, ainsi que le logiciel Nanoscale Molecular Dynamics (NAMD).
XSEDE a attribué à Gregg Beckham des allocations sur les systèmes Stampede1 et Stampede2 du Texas Advanced Computing Center (TACC) et sur le système Comet du San Diego Supercomputer Center (SDSC).
"Notre expérience à ce jour sur Stampede2 a été absolument merveilleuse, " dit Beckham. " Pour tous les codes que nous utilisons là-bas, ça a été une machine fantastique. Nous traversons rapidement les files d'attente. Nous produisons beaucoup de science de grande qualité à travers le spectre de ce que nos groupes font collectivement ensemble en utilisant Stampede2 en ce moment. Certainement, pour la recherche sur l'enzyme de dégradation des plastiques, nous l'utilisons pour les manuscrits et les études à venir sur ce même sujet."
"Une bonne chose à propos de Comet, " Woodcock a dit, "est-ce que tu as, pour les travaux que vous devez réaliser à haut débit, SDSC a une file d'attente partagée, ce qui vous permet de soumettre des travaux beaucoup plus petits mais de le faire à très haut débit, car ils peuvent partager des cœurs sur les nœuds de Comet. Cela a été particulièrement utile."
Les deux chercheurs ont convenu que le calcul aide à faire des découvertes scientifiques. "Les expérimentateurs et les informaticiens travaillent main dans la main de plus en plus fréquemment, " Woodcock a dit. " Et sans accès à des ressources comme celle-ci, cela nous ferait vraiment reculer, ou plusieurs pas en arrière pour produire les plus hauts niveaux de science et être vraiment en mesure de résoudre les problèmes les plus difficiles du monde, c'est ce que nous avons fait dans cette étude particulière, fait en partenariat avec des groupes expérimentaux de haut niveau comme nos collaborateurs au Royaume-Uni et avec nous ici aux États-Unis."

Le supercalculateur Stampede2 du Texas Advanced Computing Center, Université du Texas à Austin. Crédit :TACC
Beckham a déclaré que leur travail venait de commencer sur les enzymes qui nettoient la pollution plastique. "Nous commençons à peine à comprendre comment cette enzyme a évolué, " a déclaré Beckham. Il veut utiliser le calcul pour tirer parti des grandes bases de données de génomique et de métagénomique sur les enzymes pour trouver les aiguilles dans la botte de foin qui peuvent dégrader les plastiques.
"L'autre chose qui nous intéresse aussi, " Beckham a dit, « est-ce que si nous sommes capables de le faire à une température beaucoup plus élevée, qui serait capable d'accélérer la dégradation du PET et de nous amener dans des domaines qui pourraient potentiellement être pertinents sur le plan industriel en termes d'utilisation d'une enzyme pour dégrader le PET, puis le convertir en matériaux de plus haute valeur, ce qui pourrait inciter à des taux de remise en état plus élevés, en particulier dans les pays en développement où beaucoup de déchets plastiques se retrouvent dans l'océan."
Lee Woodcock voit les nouvelles techniques de calcul comme un changement dans la modélisation des champs de force non médicamenteux qui abordent les interactions polymères de manière plus réaliste que CHARMM et NAMD ne le peuvent aujourd'hui. "Je travaille avec des collègues du NREL pour m'assurer que nous pouvons améliorer les champs de force de manière très rapide, de sorte que si quelqu'un entre et dit que nous devons examiner ce polymère ensuite, nous sommes convaincus que nous pouvons élaborer une stratégie de modélisation en très peu de temps pour obtenir un délai rapide lorsque nous devons modéliser de nombreux polymères différents.
Les scientifiques espèrent que leur travail rendra un jour le monde en dehors du laboratoire meilleur. « Comprendre comment nous pouvons mieux concevoir des processus pour recycler les plastiques et les récupérer est un problème mondial grave et c'est quelque chose pour lequel la communauté scientifique et technique doit trouver des solutions, " a déclaré Beckham.
L'étude, "Caractérisation et ingénierie d'une polyestérase aromatique dégradant les plastiques, " a été publié en mars 2018 dans le Actes de l'Académie nationale des sciences . Les auteurs sont Harry P. Austin, Mark D. Allen, Alan W. Thorne, John E. McGeehan de l'Université de Portsmouth; Bryon S. Donohoe, Rodrigo L. Silveira, Michael F. Crowley, Antonella Amore, Nicolas A. Rorrer, Graham Dominique, William E. Michener, Christopher W. Johnson, Gregg T. Beckham du Laboratoire national des énergies renouvelables; Fiona L. Kearns, Benjamin C. Pollard, H. Lee Woodcock de l'Université de Floride du Sud; Munir S. Skaf de l'Université de Campinas; Ramona Duman, Kamel El Omari, Vitaliy Mykhaylyk, Armin Wagner de la Source de Lumière Diamant, Campus des sciences et de l'innovation Harwell. Le programme de recherche et développement dirigé par le Laboratoire national des énergies renouvelables a financé l'étude, avec le temps informatique fourni par l'allocation MCB-090159 de l'Extreme Science and Engineering Discovery Environment (XSEDE).