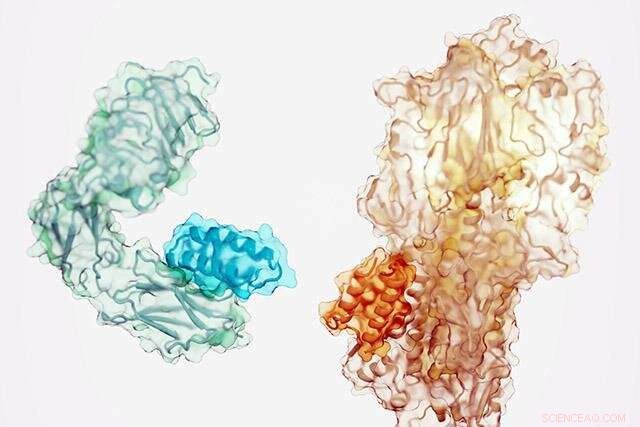
Petites protéines (teinte plus foncée) conçues pour se lier au récepteur de l'insuline (à gauche) et à un composant du virus de la grippe (à droite). Crédit :Ian C. Haydon/Institute for Protein Design
Une équipe de scientifiques a créé une nouvelle méthode puissante pour générer des médicaments protéiques. À l'aide d'ordinateurs, ils ont conçu des molécules capables de cibler des protéines importantes dans l'organisme, telles que le récepteur de l'insuline, ainsi que des protéines vulnérables à la surface des virus. Cela résout un défi de longue date dans le développement de médicaments et peut conduire à de nouveaux traitements contre le cancer, le diabète, l'infection, l'inflammation et au-delà.
La recherche, publiée aujourd'hui dans la revue Nature , a été dirigée par des scientifiques du laboratoire de David Baker, professeur de biochimie à la faculté de médecine de l'Université de Washington et lauréat du prix Breakthrough 2021 en sciences de la vie.
« La capacité de générer de nouvelles protéines qui se lient étroitement et spécifiquement à toute cible moléculaire souhaitée est un changement de paradigme dans le développement de médicaments et la biologie moléculaire plus largement », a déclaré Baker.
Les anticorps sont les médicaments à base de protéines les plus courants d'aujourd'hui. Ils fonctionnent généralement en se liant à une cible moléculaire spécifique, qui devient alors soit activée, soit désactivée. Les anticorps peuvent traiter un large éventail de troubles de santé, y compris le COVID-19 et le cancer, mais en générer de nouveaux est un défi. Les anticorps peuvent également être coûteux à fabriquer.
Une équipe dirigée par deux chercheurs postdoctoraux du laboratoire Baker - Longxing Cao et Brian Coventry - a combiné les avancées récentes dans le domaine de la conception informatique de protéines pour arriver à une stratégie de création de nouvelles protéines qui se lient à des cibles moléculaires d'une manière similaire aux anticorps. Ils ont développé un logiciel capable de scanner une molécule cible, d'identifier des sites de liaison potentiels, de générer des protéines ciblant ces sites, puis de sélectionner des millions de protéines de liaison candidates pour identifier celles qui sont les plus susceptibles de fonctionner.
L'équipe a utilisé le nouveau logiciel pour générer des protéines de liaison de haute affinité contre 12 cibles moléculaires distinctes. Ces cibles comprennent d'importants récepteurs cellulaires tels que TrkA, EGFR, Tie2 et le récepteur de l'insuline, ainsi que des protéines à la surface du virus de la grippe et du SARS-CoV-2 (le virus qui cause le COVID-19).
"Quand il s'agit de créer de nouveaux médicaments, il y a des cibles faciles et des cibles difficiles", a déclaré Cao, qui est maintenant professeur adjoint à l'Université Westlake. "Dans cet article, nous montrons que même des cibles très dures se prêtent à cette approche. Nous avons pu fabriquer des protéines de liaison à certaines cibles qui n'avaient pas de partenaires de liaison ou d'anticorps connus,"
Au total, l'équipe a produit plus d'un demi-million de protéines de liaison candidates pour les 12 cibles moléculaires sélectionnées. Les données recueillies sur ce grand pool de protéines de liaison candidates ont été utilisées pour améliorer la méthode globale.
"Nous sommes impatients de voir comment ces molécules pourraient être utilisées dans un contexte clinique, et plus important encore, comment cette nouvelle méthode de conception de médicaments protéiques pourrait conduire à des composés encore plus prometteurs à l'avenir", a déclaré Coventry. Vers un médicament pour traiter tous les coronavirus