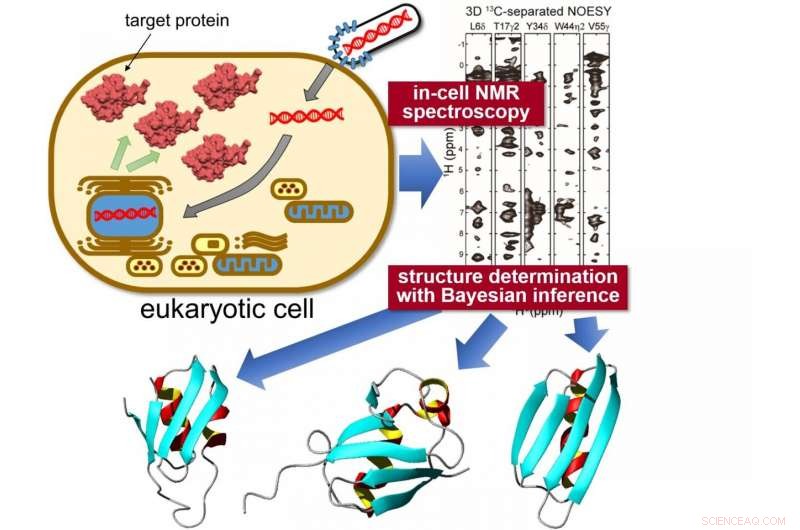
Les protéines cibles sont exprimées à l'intérieur sf9 cellules, puis mesuré par spectroscopie RMN "in-cell". Analyse statistique unique à l'aide de Inférence bayésienne est appliqué pour calculer la structure 3D précise des protéines avec une précision sans précédent. Crédit :Université métropolitaine de Tokyo
Des chercheurs de l'Université métropolitaine de Tokyo ont déterminé avec succès la haute résolution, structure tridimensionnelle des protéines à l'intérieur des cellules eucaryotes vivantes. Ils ont combiné la spectroscopie de résonance magnétique nucléaire (RMN) "in-cell", un système de bioréacteur et des algorithmes informatiques de pointe pour déterminer pour la première fois les structures des protéines dans des environnements intracellulaires surpeuplés. La technique promet un aperçu du comportement intracellulaire des protéines causant des maladies et de nouvelles applications de criblage de médicaments, permettant une visualisation in situ de la façon dont les protéines répondent aux stimuli biochimiques.
Les cellules eucaryotes sont les éléments constitutifs d'une vaste gamme d'organismes, y compris tous les champignons, plantes et animaux. Leur structure interne est extrêmement complexe et variée, avec une hiérarchie structurelle complexe et une vaste gamme de biomacromolécules distribuées autour d'un réseau cytosquelettique. Cela a rendu difficile de voir ce que chaque protéine à l'intérieur des cellules fait dans son environnement naturel, malgré les avantages biomédicaux évidents de la connaissance, par exemple. comment une protéine particulière réagit lorsque les cellules sont soumises à des stimuli chimiques, comme les médicaments pharmaceutiques.
Pour relever ce défi, une équipe de l'Université métropolitaine de Tokyo dirigée par le professeur assistant Teppei Ikeya et le professeur Yutaka Ito a appliqué des mesures de spectroscopie par résonance magnétique nucléaire (RMN) à des protéines spécifiques exprimées à l'intérieur sf9 cellules d'insectes cultivées, une souche de cellules dérivée à l'origine d'un type de larve de papillon largement utilisé pour la production de protéines. Les travaux de RMN pionniers de l'équipe avaient déjà réussi à élucider les structures protéiques à haute résolution à l'intérieur des bactéries (non eucaryotes). Le problème d'appliquer simplement les mêmes techniques aux protéines dans sf9 cellules était la concentration significativement plus faible de protéines cibles et la courte durée de vie des cellules, ce qui rend difficile la collecte de spectres RMN multidimensionnels de haute qualité pour la spectroscopie à effet Overhauser nucléaire (NOESY) qui donneraient des informations précises sur la façon dont les différents atomes sont espacés à l'intérieur des molécules individuelles. Ainsi, ils ont combiné un schéma de mesure RMN rapide basé sur un échantillonnage clairsemé avec des méthodes de calcul de pointe utilisant des techniques statistiques telles que l'inférence bayésienne, méthodes adaptées pour élucider efficacement les structures protéiques sur la base d'une quantité limitée d'informations structurelles provenant de spectres RMN cellulaires avec une faible sensibilité intrinsèque. Un système de bioréacteur a également été équipé à l'intérieur de l'appareil RMN qui a maintenu les cellules dans un état sain pendant les mesures.
Avec ces nouvelles données, l'équipe a pu élucider la structure 3-D de trois protéines modèles avec une résolution sans précédent, avec une précision de 0,5 Angströms (0,05 nanomètre) pour la position des atomes de la chaîne principale de la protéine. En particulier, ils ont identifié une conformation significativement différente dans une région localisée de l'une des protéines par rapport à sa structure de référence en solution diluée. La différence de conformation entre les protéines "dans les cellules" et "dans les tubes à essai" a probablement été causée par des interactions non spécifiques avec d'autres molécules à l'intérieur des cellules. Il devient clair que ces interactions contribuent aux fonctions biologiques des protéines :la capacité de localiser et de quantifier les changements structurels des protéines dans un environnement intracellulaire devrait avoir un impact significatif sur la recherche biomédicale, permettant de voir comment différentes conditions, par ex. les maladies neurodégénératives affectent les conformations des protéines in situ, et évaluer quantitativement l'impact des traitements sur les anomalies structurelles.