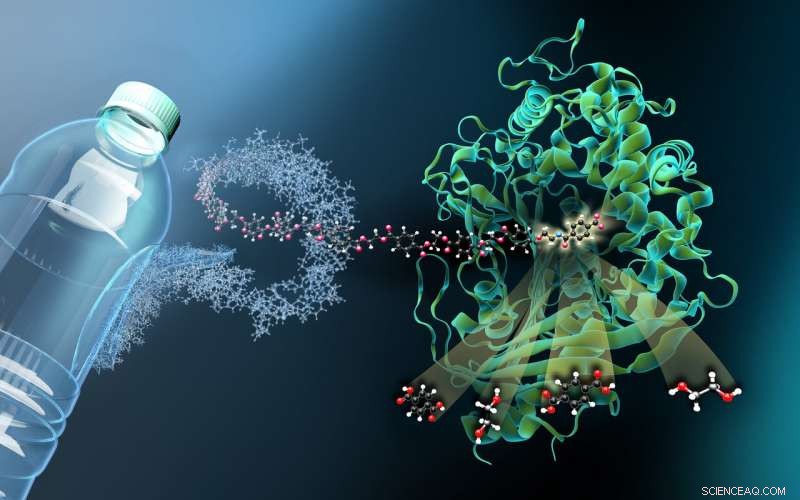
L'image représente la structure de la MHETase liée à un ligand dérivé du PET non hydrolysable (MHETA). Des fibres polymères symboliques relient MHETA à une bouteille PET typique, indiquant le grand potentiel des variantes améliorées de la PETase et de la MHETase dans le recyclage enzymatique du plastique. À cet égard, nos données structurelles ont facilité la génération de variantes initiales de MHETase avec une activité améliorée et une spécificité de substrat étendue. Nos résultats représentent donc une étape importante sur la voie d'un système PETase / MHETase amélioré où le recyclage du PET est découplé de sa dépendance au pétrole brut de manière économe en énergie et durable. Crédit :Martin Künsting/HZB
Une équipe de recherche de l'Université de Greifswald et Helmholtz-Zentrum-Berlin (HZB) a résolu la structure moléculaire de l'enzyme MHETase à BESSY II. La MHETase a été découverte dans des bactéries, et avec une seconde enzyme, PETase, est capable de décomposer le PET plastique largement utilisé en ses blocs de construction de base. Cette structure 3D a déjà permis aux chercheurs de produire un variant de MHETase avec une activité optimisée afin de l'utiliser, avec la PETase, pour un recyclage durable du PET.
Les plastiques sont extrêmement polyvalents et presque éternellement durables. Mais c'est aussi un problème, car après seulement environ 100 ans de production de plastique, les particules de plastique se trouvent maintenant partout - dans les eaux souterraines, dans les océans, dans l'air, et dans la chaîne alimentaire. Environ 50 millions de tonnes de PET polymère d'importance industrielle sont produites chaque année. Seule une infime fraction du plastique est actuellement recyclée via des processus coûteux et énergivores qui donnent soit des produits déclassés, soit dépendent à leur tour de l'ajout de pétrole brut « frais ».
En 2016, un groupe de chercheurs japonais a découvert une bactérie qui se développe sur le PET et s'en nourrit partiellement. Ils ont découvert que la bactérie possède deux enzymes spéciales, PETase et MHETase, qui digèrent les polymères plastiques PET. PETase décompose le plastique en plus petits blocs de construction PET, principalement MHET, et la MHETase le divise en deux éléments précurseurs de base du PET, l'acide téréphtalique et l'éthylène glycol. Les deux composants sont précieux pour synthétiser un nouveau PET sans ajout de pétrole brut pour un cycle fermé de production et de récupération durable.
En avril 2018, la structure de la PETase a finalement été résolue indépendamment par plusieurs groupes de recherche. La source lumineuse Diamond a également été impliquée dans les expériences. Cependant, La PETase n'est qu'une partie de la solution. Il est également important de caractériser la structure de la deuxième enzyme, MHETase.
"La MHETase est considérablement plus grande que la PETase et encore plus complexe. Une seule molécule de MHETase est constituée de 600 acides aminés, ou environ 4000 atomes. La MHETase a une surface environ deux fois plus grande que la surface de la PETase et a donc considérablement plus de potentiel d'optimisation vers la décomposition du PET, " explique le biochimiste et biologiste structural Dr. Gert Weber de Helmholtz-Zentrum Berlin et Freie Universität Berlin.
Au cours d'une chaire intérimaire à l'Université de Greifswald, Weber a contacté le biotechnologue Prof. Uwe Bornscheuer à l'Institut de biochimie, qui était déjà impliqué dans les enzymes dégradant le plastique. Ensemble, ils ont développé l'idée de résoudre la structure de la MHETase, puis d'utiliser cette connaissance pour optimiser l'enzyme pour des applications dans le recyclage du PET. Pour faire ça, ils devaient d'abord extraire l'enzyme des cellules bactériennes et la purifier. Au sein de cette collaboration, les équipes ont réussi à obtenir l'architecture tridimensionnelle complexe de MHETase à BESSY II, la source synchrotron du HZB à Berlin.
"Afin de voir comment la MHETase se lie au PET et le décompose, vous avez besoin d'un fragment de plastique qui se lie à la MHETase mais n'est pas clivé par elle, " explique Weber. Membre de l'équipe de recherche antérieure de Weber à Greifswald, Dr Gottfried Palm, découper une bouteille PET, a décomposé chimiquement le polymère PET et en a synthétisé un petit fragment chimique qui se lie à la MHETase mais ne peut plus être clivé par celle-ci. À partir de cette MHETase « bloquée », de minuscules cristaux ont été cultivés pour des études structurelles au HZB. "Les investigations structurelles nous ont permis de regarder virtuellement la MHETase "à l'œuvre" et de développer des stratégies pour optimiser cette enzyme, " explique Weber.
"Grâce au format de groupe de recherche conjoint, nous avons les moyens de proposer un accès beamtime sur les lignes très demandées BESSY II MX pour des mesures très rapides à tout moment, " dit le Dr Manfred Weiss, responsable des lignes de lumière BESSY II MX. The three-dimensional architecture of MHETase actually displays some special features:enzymes such as MHETase bind to their target molecule first before a chemical reaction occurs. For breakdown of a molecule you need a tailor-made enzyme:"We can now exactly localise where the MHET molecule docks to MHETase and how MHET is then split into its two building blocks terephthalic acid and ethylene glycol, " dit Weber.
Cependant, neither PETase nor MHETase are particularly efficient. "Plastics have only been around on this scale for a few decades—even bacteria with their rapid successions of generations and rapid adaptability have not managed to develop a perfect solution through the evolutionary process of trial and error over such a short time, " explains Weber. "Thanks to the clarification of the structure of this very important enzyme, we have now also been able to plan, produce and biochemically characterise variants that show significantly higher activity than natural MHETase and are even active against another intermediate product of PET degradation, BHET, " adds Uwe Bornscheuer.
Dans le futur, Uwe Bornscheuer will work on systematically optimising the enzymes PETase and MHETase for their task—the decomposition of PET. Gert Weber plans to supplement these studies with further work on biological structures in order to systematically develop plastic-digesting enzymes for environmental applications. Access to the measuring stations and the IT infrastructure of HZB is indispensable for this.
Producing these kinds of enzymes in closed biotechnological cycles, par exemple, could be a way to really break down PET plastics and other polymers into their basic building blocks. This would also be the key to ideal recycling and a long-term solution to the plastic waste problem:production of plastic would be a closed cycle and no longer dependent on crude oil.
L'étude est publiée dans Communication Nature .