
Une nouvelle technique permettra de détecter les virus sur le terrain. Dans ce cas, une parcelle d'igname en Guadeloupe. Crédit :D. Filloux, Cirad
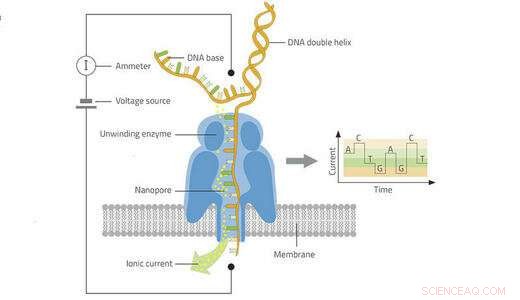
Dans une première en virologie végétale, une équipe du Cirad a récemment utilisé la technologie des nanopores pour séquencer les génomes entiers de deux virus à ARN d'igname. Cette technique de biologie moléculaire peu utilisée mais prometteuse ouvre la voie à de nouveaux outils de diagnostic de terrain des plantes, maladies animales et humaines.
Un nouveau, haut débit, La technique de séquençage portable a été développée ces dernières années à des fins de santé humaine et animale. Il utilise des laboratoires mobiles pour diagnostiquer des virus comme Ebola ou Zika presque instantanément, Sur le terrain. Le diagnostic est rapide et précoce, ce qui évite le transfert d'échantillons contaminés.
« La technologie se caractérise par la production de longues séquences nucléotidiques, qui permet de séquencer l'ensemble du génome viral, " Philippe Roumagnac, un virologue au Cirad, explique. Le Cirad a été l'un des premiers laboratoires au monde à tester et valider son utilisation en virologie végétale. "En utilisant un plant d'igname malade, il ne nous a fallu que quelques heures pour séquencer le génome entier de deux virus à ARN simple brin, un macluravirus et un potyvirus, " ajoute son collègue Denis Filloux.
Comme pour la virologie humaine, le fait que la technique soit désormais validée dans un laboratoire de virologie végétale ouvre la voie au temps réel, détection mobile des chroniques, virus végétaux saisonniers ou émergents, même dans les zones isolées. En raccourcissant le temps qui s'écoule entre le prélèvement et le diagnostic, la technologie aidera les réseaux d'épidémiosurveillance à détecter les organismes nuisibles à un stade plus précoce.
Crédit :Kerstin Göpfrich