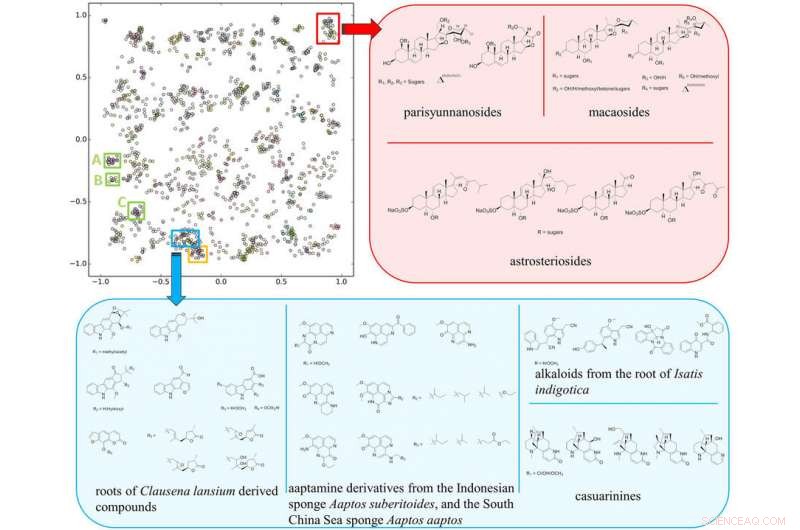
La carte de cluster SMART basée sur le résultat de la formation de 2, 054 spectres HSQC sur 83, 000 itérations, avec des encadrés représentant les différentes classes de composés abordées dans le texte. Crédit :Université de Californie - San Diego
Une équipe interdisciplinaire de chercheurs de l'Université de Californie à San Diego a développé une méthode pour identifier les structures moléculaires des produits naturels qui est nettement plus rapide et plus précise que les méthodes existantes. La méthode fonctionne comme la reconnaissance faciale pour les structures moléculaires :elle utilise une donnée spectrale unique à chaque molécule, puis la fait passer par un réseau neuronal d'apprentissage en profondeur pour placer la molécule inconnue dans un groupe de molécules avec des structures similaires.
Le nouveau système s'appelle "SMART, " qui signifie Small Molecule Accurate Recognition Technology, et a le potentiel d'accélérer le processus d'identification de la structure moléculaire par dix. Ce développement pourrait représenter un changement de paradigme dans l'analyse chimique, domaines pharmaceutiques et de découverte de médicaments puisque 70 pour cent de tous les médicaments approuvés par la Food and Drug Administration (FDA) sont basés sur des produits naturels tels que les micro-organismes du sol, les plantes terrestres et, de plus en plus, formes de vie marine telles que les algues.
"La structure d'une molécule est l'information habilitante, " dit Bill Gerwick, professeur d'océanographie et de sciences pharmaceutiques à la Scripps Institution of Oceanography de l'UC San Diego. « Vous devez avoir la structure pour toute approbation de la FDA. Si vous voulez avoir la propriété intellectuelle, vous devez breveter cette structure. Si vous voulez faire des analogues de cette molécule, vous devez savoir quelle est la molécule de départ. C'est une information critique."
Chen Zhang, un doctorat en nano-ingénierie. étudiant à l'UC San Diego collaborant avec Gerwick et le premier auteur de l'article publié dans Rapports scientifiques sur la nature , a déclaré que la détermination de la structure d'une molécule peut être un goulot d'étranglement dans le processus de recherche de produits naturels, il faut des mois et même des années à des experts pour déterminer avec précision la structure correcte et complète. Bien que chaque molécule et sa chronologie d'identification soient différentes, l'approche SMART donne aux chercheurs un indice précoce sur la famille à laquelle appartient une nouvelle molécule, réduire drastiquement le temps de caractérisation d'un nouveau produit naturel.
« La façon dont nous avons pu accélérer le processus consiste essentiellement à utiliser un logiciel de reconnaissance faciale pour examiner les informations clés que nous obtenons sur les molécules, " a déclaré Gerwick. L'information clé utilisée par l'équipe est appelée résonance magnétique nucléaire à cohérence quantique singulière hétéronucléaire, ou RMN HSQC, spectre. Il produit une carte topologique des taches qui révèlent quels protons de la molécule sont directement liés à quels atomes de carbone, un arrangement unique à chaque molécule.
Zhang et Gerwick ont fait équipe avec Gary Cottrell, professeur d'informatique et d'ingénierie à la UC San Diego Jacobs School of Engineering, pour développer un système d'apprentissage en profondeur formé avec des milliers de spectres HSQC tirés de recherches antérieures. Ce réseau neuronal convolutif prend une image 2D du spectre RMN HSQC d'une molécule inconnue et la mappe dans un espace à 10 dimensions regroupé à proximité de molécules similaires, permettant aux chercheurs d'élucider plus facilement la structure d'une molécule inconnue.
"Chen a adopté cette approche pour obtenir des spectres RMN de plus de 4, 000 composés de la littérature en découpant littéralement les images des PDF des articles, " a déclaré Cottrell. " C'était un effort impressionnant ! Toutefois, ce n'est normalement pas assez de données pour former un réseau profond, mais nous avons utilisé une technologie appelée réseau siamois, dans lequel vous vous entraînez sur des paires d'images. Cela amplifie votre ensemble d'entraînement d'environ le carré du nombre de composés dans une famille, et c'est ce qui a rendu ce projet réalisable."
Cette collaboration est la première fois que Gerwick a encadré un étudiant en génie, et l'échange d'idées s'est avéré fructueux.
"Ce fut une interaction merveilleuse. L'UC San Diego a quelque chose de vraiment magique, et c'est la profondeur de la collaboration qui se produit entre les départements—c'est phénoménal, " a déclaré Gerwick. "Lorsque vous essayez de prendre de manière réfléchie d'une autre discipline quelque chose qui est peut-être même banal dans cette discipline et de l'appliquer d'une manière nouvelle et unique dans notre discipline, c'est une opportunité d'avoir vraiment ce genre de changement de paradigme. Et je pense que cette technologie, avec un peu d'avancement, pourrait être un véritable changement de paradigme dans la façon dont nous effectuons toutes sortes d'analyses chimiques et chimiques. »