Crédit :Université d'Oxford
Des chercheurs de l'Université d'Oxford ont construit une « trémie moléculaire, " capable de déplacer des brins simples d'ADN à travers un nanotube de protéine.
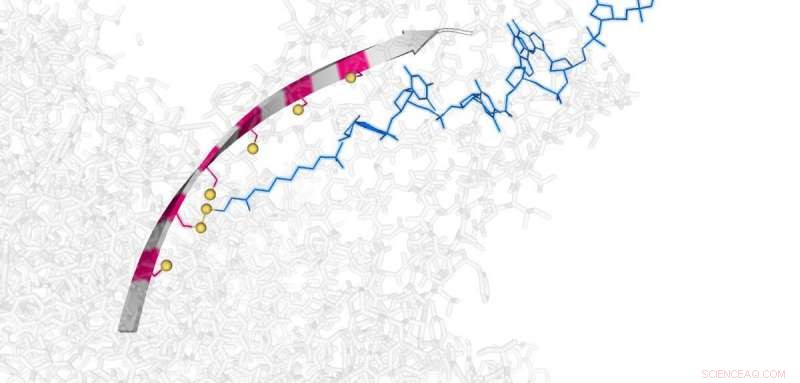
La petite trémie fonctionne en créant et en rompant en séquence des liaisons chimiques simples qui la relient à une piste à l'échelle nanométrique. Cela peut être activé, éteint ou inversé par un petit potentiel électrique, ce qui pourrait finalement le rendre approprié pour une utilisation dans des dispositifs de séquençage d'ADN à nanopores.
Professeur Hagan Bayley du Département de chimie de l'Université d'Oxford, qui a dirigé la recherche, a déclaré:"Être capable de contrôler le mouvement moléculaire est le Saint Graal de la construction de machines à l'échelle nanométrique. Être capable de traiter des molécules uniques d'ADN sous un contrôle chimique précis peut fournir une alternative à l'utilisation d'enzymes dans les technologies de séquençage de l'ADN, améliorant leur vitesse et le nombre de molécules pouvant être analysées en parallèle."
Le prix Nobel 2016 a été décerné en partie pour la construction de molécules à éléments coulissants et rotatifs, démontrant l'importance de cette technologie dans de nombreux domaines. L'équipe d'Oxford a fait progresser considérablement cette technologie en produisant des molécules qui effectuent des sauts sub-nanométriques pouvant être détectés un à un et soumis à un contrôle externe.
La trémie prend actuellement quelques secondes pour chaque étape, et les chercheurs cherchent maintenant à augmenter la vitesse de la chimie ainsi que la longueur de la piste, qui est actuellement limité à six prises.
Comment fonctionne la trémie
Le mouvement de saut utilise une chimie très simple basée sur 3 atomes de soufre [échange thiol/disulfure], qui se produit dans l'eau à température ambiante. La trémie fait des pas sub-nanométriques (0,7 nm), et est alimenté et contrôlé par un champ électrique ; le sens du saut peut être commuté en inversant le champ électrique. Tout cela est suivi en temps réel au niveau d'une seule molécule.
Un mouvement de cliquet est nécessaire pour le séquençage des nanopores, qui est actuellement obtenu en utilisant une enzyme. Le mouvement de saut dans le dispositif nouvellement publié est un cliquet chimique et ce principe pourrait être appliqué au séquençage de l'ADN et de l'ARN car la taille du pas est similaire à la distance internucléotidique dans l'ADN simple brin.
Le papier complet, "Contrôle directionnel d'une trémie moléculaire processive, " est publié dans la revue Science .