Crédit :Wiley
Quelques petites cellules différentes des autres peuvent avoir un grand effet. Par exemple, des cellules cancéreuses individuelles peuvent être résistantes à une chimiothérapie spécifique, provoquant une rechute chez un patient qui serait autrement guéri. Dans la revue Angewandte Chemie , les scientifiques ont maintenant introduit une puce basée sur la microfluidique pour la manipulation et l'analyse ultérieure des acides nucléiques de cellules individuelles. La technique utilise des champs électriques locaux pour « piéger » très efficacement les cellules (diélectrophorèse).
Des analyses moléculaires de cellules individuelles sont nécessaires pour mieux comprendre le rôle des populations cellulaires hétérogènes dans le développement de maladies et pour développer des thérapies efficaces pour la médecine personnalisée. L'identification de cellules individuelles dans une masse d'autres cellules est un énorme défi en médecine diagnostique. Les cellules doivent être triées, tenu, transféré dans un autre récipient avec un volume extrêmement petit ( <1 L) et doit ensuite subir une analyse moléculaire. Les méthodes conventionnelles sont généralement très longues et complexes, ainsi que peu fiable et inefficace. Ils peuvent également compromettre la viabilité des cellules, nécessitent de gros volumes d'échantillons, présentent un risque élevé de contamination, et/ou nécessitent des instruments coûteux.
Des scientifiques de l'Université de Washington (Seattle, ETATS-UNIS), Iowa State University (Ames, ETATS-UNIS), et Centre de recherche sur le cancer Fred Hutchinson (Seattle, USA) ont utilisé la technologie microfluidique pour surmonter ces problèmes. Toutes les étapes nécessaires se déroulent de manière fiable sur une micropuce spécialement développée en utilisant des quantités minimales de solvant et sans nécessiter le marquage des cellules. Contrairement aux puces microfluidiques classiques, celui-ci ne nécessite ni technologie de fabrication complexe ni composants tels que vannes ou agitateurs.
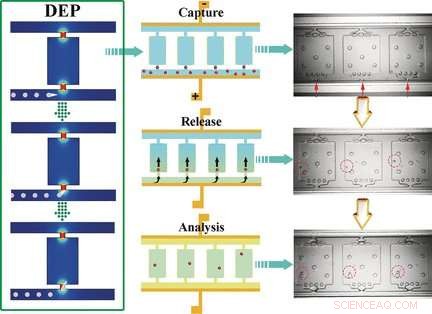
La puce autonumérique diélectrophorétique (SD-DEP) a à peu près la taille d'une pièce de monnaie et possède deux microcanaux parallèles (50 m de profondeur x 35 m de largeur x 3,2 cm de longueur) reliés par de nombreuses petites chambres minuscules. Les ouvertures des microcanaux n'ont que 15 µm de large. Une fine électrode est étirée le long des canaux. Les canaux et les chambres sont remplis d'un tampon, une tension alternative est appliquée, et l'échantillon est ajouté à l'un des microcanaux. L'équipe dirigée par Robbyn K. Anand et Daniel T. Chiu a utilisé des cellules leucémiques dans leurs expériences.
Des maxima locaux du champ électrique se produisent aux entrées étroites des chambres. Les cellules qui entrent dans les chambres sont "piégées". Parce que les dimensions de l'entrée sont similaires à la taille moyenne d'une cellule, une seule cellule peut être piégée par chaque entrée de chambre. Lorsque le courant alternatif est coupé et que le débit est augmenté par injection des réactifs nécessaires à l'analyse ultérieure, les cellules sont lavées dans les chambres. Une huile est ensuite ajoutée pour sceller les chambres. Les cellules sont ensuite dissoutes, et les acides nucléiques sont libérés et multipliés et peuvent être identifiés comme des cellules leucémiques par un gène marqueur.
Dans les études futures, les chercheurs espèrent utiliser la puce pour déterminer la distribution des mutations génétiques liées à la résistance des cellules leucémiques et pouvant ainsi provoquer des rechutes.