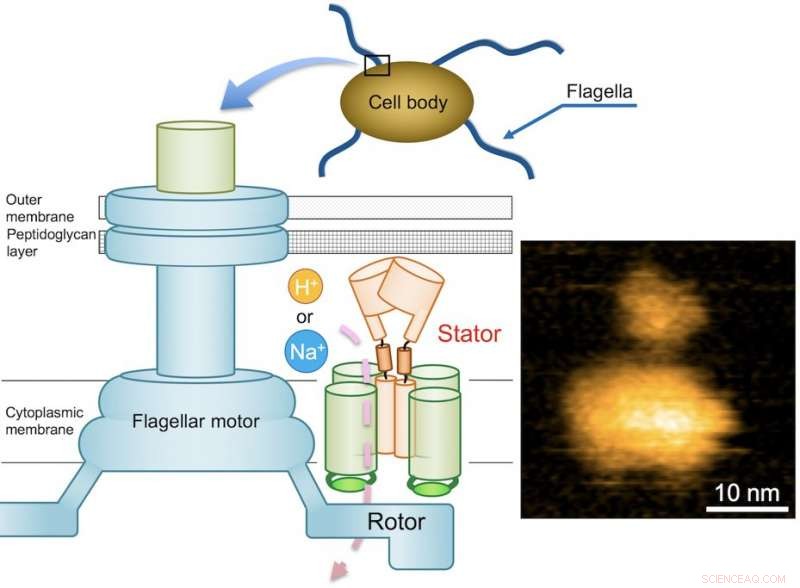
Fig. 1. Diagramme schématique du moteur flagellaire bactérien et une image AFM typique du complexe MotPS purifié. Crédit :Université d'Osaka
Les bactéries nagent de différentes manières, et les moteurs qui entraînent leur nage sont très variés, impliquant une réponse adaptative à un environnement. L'un des moteurs les plus communément identifiés est le flagelle. Bien que la motilité soit une caractéristique principale des flagelles, des propriétés adhésives et autres leur ont également été attribuées.
Le moteur flagellaire est composé d'un rotor et d'une dizaine de stator, et est entraîné par l'énergie créée par les ions lorsqu'ils migrent à travers la membrane cellulaire. Alors que le moteur flagellaire de certaines bactéries d'eau douce et entériques, comme les salmonelles, est alimenté par des protons (ions hydrogène, H + ), qui se compose de quatre copies de la protéine MotA et de deux copies de MotB, les moteurs d'autres bactéries qui vivent dans des environnements d'eau salée sont alimentés par des ions sodium (Na + ).
Dans le cas de Bacillus subtilis (B. subtilis) , une bactérie que l'on trouve couramment dans le sol, le moteur flagellaire comporte deux types distincts de blocs statoriques :le MotAB de type H+ et le MotPS de type Na+. Pendant que les composants sont identifiés, le mécanisme qui alimente le moteur flagellaire reste inconnu. Ce mystère est encore aggravé par le fait que les flagelles ne sont pas clairement visibles au microscope optique. Ces défis ont mis une équipe de chercheurs de l'Université d'Osaka dans une quête pour élucider le mécanisme inconnu. Ils ont fait part de leurs découvertes dans Avancées scientifiques .
"Des analyses précises de la dynamique structurelle du moteur flagellaire à l'aide de la microscopie à force atomique à grande vitesse (HS-AFM) combinées à une analyse mutationnelle ont fourni des informations sur le mécanisme de génération de couple du moteur flagellaire, " dit le premier auteur de l'étude Naoya Terahara. " Nous avons fait des observations en temps réel des changements structurels induits par Na+ dans le domaine de MotS qui lie le peptidoglycane, un polymère constitué de sucres et d'acides aminés dans les bactéries."
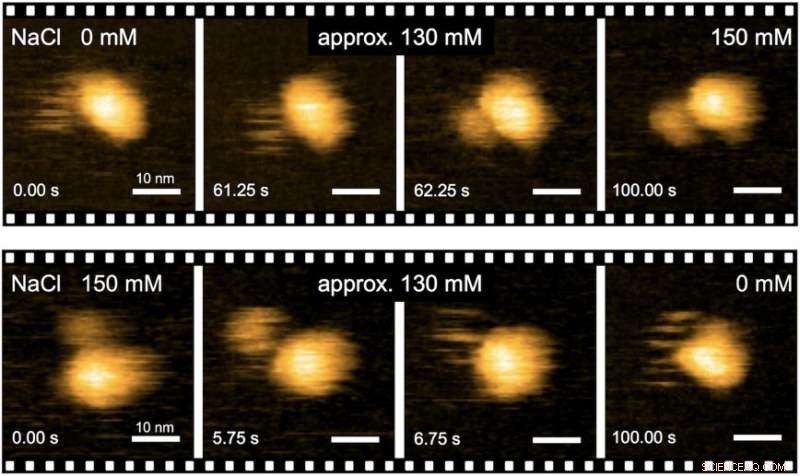
Fig. 2. Imagerie en temps réel des transitions structurelles induites par Na+ du complexe MotPS par HS-AFM. Crédit :Université d'Osaka
Pour comprendre comment le complexe protéique MotPS répond aux changements de l'environnement, les chercheurs ont d'abord caractérisé la relation couple-vitesse du moteur de B. subtilis sur une large gamme de concentrations de Na+. La vitesse maximale du moteur a été réduite d'environ 200 à 80 Hz lorsque la concentration externe de Na+ a été augmentée de 0 à 200 mM, bien que le couple de décrochage n'ait pas du tout été modifié.
Notamment, lorsque le domaine de MotB qui se lie au peptidoglycane a été remplacé par celui de MotS, le moteur chimérique maintenait la même courbe couple-vitesse que le moteur MotAB en présence d'ions Na+ mais pas en l'absence. Cela signifie que Na+ est le principal moteur de l'assemblage et de l'activation du moteur flagellaire, quelle que soit sa composition.
"En l'absence d'ions sodium, MotPS existe sous une forme inactive dans la membrane cytoplasmique parce que MotSc, le domaine périplasmique C-terminal de MotS, adopte une conformation dépliée. Lorsque la concentration en ions sodium augmente, la liaison des ions à MotSc induit le repliement du domaine PGB et facilite sa dimérisation de manière très coopérative, ", dit l'auteur correspondant Tohru Minamino.
A travers cette étude, l'équipe a également démontré la faisabilité de l'utilisation de la HS-AFM combinée à l'analyse mutationnelle pour étudier des structures bactériennes complexes, qui peuvent fournir des informations précieuses pour la production industrielle de produits commercialement viables, comme les protéases et les thérapeutiques, comme dans le cas du B. subtilis.
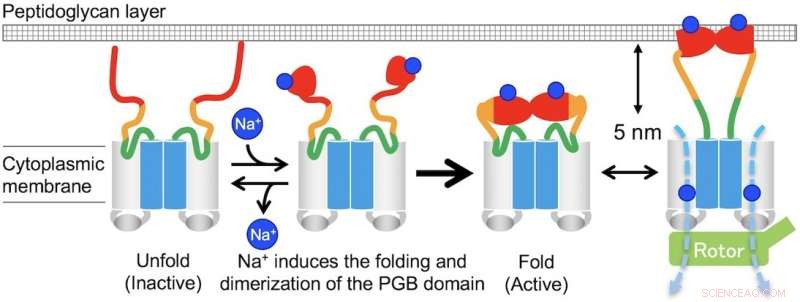
Fig. 3. Modèle pour l'assemblage et le désassemblage induits par Na+ du complexe statorique MotPS. Crédit :Université d'Osaka